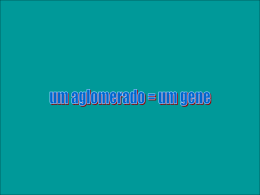Uso da bioinformática na análise genômica Início Bioinformática Receber Processar Anotar Depositar Fim Processamento de seqüências 30 20 cromatograma 10 acgatctcgctagctgctactgtagccgcgattattcgcgatctacgtatatcgcgatcgatc • O programa Phred lê o cromatograma e nomeia as bases • Cada base tem uma chance de erro de sua nomeação (10% = 0,1) DNA: • A escala de Phred é semelhante à de pH multiplicado por 10: FASTA - chance de erro de 0,001 = 10-3 = Phred 30 Seq.qual • A nomeação é praticamente aleatória no início e no final, onde a chance de erro é alta (baixo valor de Phred) Crescimento do GenBank Seqüências 16.000.000 15 milhões 14.000.000 24h Europeu 12.000.000 Japonês 10.000.000 8.000.000 6.000.000 4.000.000 2.000.000 606 0 Ano Seqüencias do DNA Seqüências do mRNA Repetição calculada •draft = 5x •finished = 10x Amostragem tecidos momentos (genoma) Genoma pequeno (seqüenciador grande) (genes expressos) Eucariotos mRNA TR cDNA Seqüenciamento parcial de transcritos Seqüênciamento de genes expressos: Documentar a existência de transcritos gênicos num transcriptoma [otorrin... e ...damonh...] • EST (Etiqueta de Seqüência Expressa) – seqüenciamento único de cada cDNA – extremidades 5’ ou 3’ • ORESTES (ESTs ricas em ORFs) – seqüenciamento único do amplicon derivado de cDNA por PCR inespecífico – prevalece o centro do cDNA (cds) Um mRNA & suas ESTs AUG ATG cDNA (fita +) ATCATGACTTACGGGCGCGCGAT cDNA (fita -) AUG (A)20 (A) 18 0(T)18 cDNA (fita +) GGCGCGCGATATCC cDNA (fita -) (A)20 (A) 18 0(T)18 Quantas ESTs de Schistosoma mansoni há no NCBI? Quantas proteínas não mitocondriais? PCR inespecífico & seu ORESTES AUG ampliconGGGCGCGCGATATCGAAAAATTTATAAGGCTAG (fita +) CCCCGGCGGCTCGGCCGGGGAGATCGATCATGAC AGATCGATCATGACTTACGGGCGCGCGATATCG amplicon cDNA (fita -) Iniciador PCR (60ºC(60ºC)37ºC) Quantos ORESTES estão hoje no NCBI? Eles são ESTs? Só há ORESTES humanos? (A)200 250 200 150 100 2. Estudando coleções de sequências • Alinhamento de um grupo pequeno de sequências com MultiAlign • Descarregando um grande número de sequências através do BatchEntrez • Formação de aglomerados de sequências com Icatools, com Cap3 ou Megablast • Estimando distância evolutiva com Phylip Aglomerados ou Clusters • Uma das atividades em bioinformática é formar aglomerados de todas as sequências geradas no projeto (as figurinhas de um álbum) • Podemos saber quantas vezes um gene foi seqüenciado, e detectar os freqüentes! • E quantos dos genes foram detectados – Usa-se também para validar bibliotecas – Pode usar dados originais ou descarregados do NCBI • Alinhamento global: usar para publicar • Quando se quer comparar globalmente as sequências, busca-se o melhor alinhamento global • Um bom algoritmo computacional é o usado no Fast Alignment, apelidado de FASTA Descarregando muitas seqüências com BatchEntrez • Inicialmente busque suas seqüências • Descarregue uma lista dos indicadores delas (GI) • Use o BatchEntrez para baixar o FASTA das seqüências • Estude-as localmente com seu programa predileto Alinhamento Global • Comparação exaustiva • Visando • Use o BatchEntrez para baixar o FASTA das seqüências • Estude-as localmente com seu programa predileto Phylip • Genes ortólogos são os homólogos mais próximos entre dois organismos (BestHit) • A evolução os faz diferentes • Phylip DNAdist mede • Phylip Protdist para proteínas • Plylip Neighbor faz uma matriz • E um desenhador de árvore: Programas para aglomerar • • • • • • Icatools Phrap Cap3, Cap4 Swat BLAST MegaBLAST Um aglomerado = Um gene UniGene • Organização das sequências do GenBank em um conjunto de aglomerados • Cada aglomerado do UniGene contém as sequências que representam um gene único • E também informações relacionadas, como em que tecidos o gene é expresso, etc. • E também onde está mapeado MegaBLAST gera o UniGene Todas ESTs contra todas Detecção de homologia > 96% de identidade > 70% do potencial Aglomerar Qualidade das bibliotecas (100 primeiras ESTs) Boa biblioteca? 1 2 3 4 5 7 9 11 Freqüência em que uma EST foi amostrada catcgatcgatcgtcgtagctacgtagctagctagctagctagctagctaactagctgactg catcgatcgatcgtcgtagctacgtagctagctagctagctagctagctagctagctgactg catcgatcgatcgtcgtagctacgtagctagctagctagctagctagctagctagctgactg catcgatcgatggtcgtagctacgtagctagctagctagctagctagctagctagctgactg catcgatcgatcgtcgtagctacgtagctagctagctagctagctagctagctagctgactg catcgatcgatcgtcgtagctacgtagctagctagctagctagctagctagctagctgactg catcgatcgatcgtcgtagctacgtagctagctagctagctagctagctagctagctgactg catcgatcgatcgtcgtagctacgtagctagctagctagctagctagctagctagctgactg catcgatcgatcgtcgtagctacgtagctagctagctagctagctagctagctagctgactg catcgatcgatcgtcgtagctacgtagctatctagctagctagctagctagctagctgactg catcgatcgatcgtcgtagctacgtagctagctagctagctagctagctagctagctgactg catcgattgatcgtcgtagctacgtagctagctagctagctagctagctagctagctgactg catcgatcgatcgtcgtagctacgtagctagctagctagctagctagctagctagctgactg catcgatcgatcgtcgtagctacgtagctagctagctagctagctagctagctagctgactg catcgatcgatcgtcgtagctacgtagctagctagctagctagctagctagctagctgactg catcgatcgatcgtcgtagctacgtagctagctagctagctagctagctagctagctgactg catcgatcgatcgtcgtagctacgtagctagctagctagctagctagctagctagctgactg
Baixar