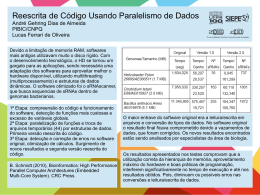
Uma abordagem para detecção e remoção de artefatos em seqüências ESTs Christian Baudet Zanoni Dias (Orientador) Instituto de Computação – Unicamp Campinas, 01 de Dezembro de 2006 Roteiro Motivação Conceitos Nova estratégia de detecção de artefatos Detecção e remoção de derrapagem Detecção e remoção de baixa qualidade Novo conjunto de procedimentos de detecção e remoção de artefatos Conclusões e trabalhos futuros Motivação Importância da pesquisa genômica Identificação de genes pode trazer: Descoberta da cura de doenças Descoberta de características de interesses comerciais Conhecimento sobre evolução dos organismos Necessidade de dados confiáveis Transcrição e Tradução O que são ESTs? EST – Expressed Sequence Tags Transcrição Adams et al, 1991 – “Complementary DNA sequencing: expressed sequence tags and the human genome project” Gene transcrito para mRNA Produção de cDNA Enzima transcriptase reversa E o que são artefatos? Artefatos são trechos que: Não pertencem ao organismo alvo do estudo Baixa qualidade ou baixa complexidade Remoção necessária Podem influenciar negativamente a análise dos dados Seqüência original Artefatos de baixa qualidade Artefato de vetor Artefato de adaptador Artefato de cauda poli-A Seqüência final Detecção e remoção de artefatos Conjuntos de procedimentos variam muito de projeto para projeto Os conjuntos normalmente são formados por várias etapas As etapas são dependentes entre si Execução é feita seqüencialmente Cada etapa depende do resultado da etapa anterior Nova Estratégia Detecções de diferentes artefatos Etapas independentes Cada etapa analisa a seqüência original Etapa final Combina todos os artefatos encontrados Identifica a região do inserto Comparação das estratégias Avaliação da Nova Estratégia Processamento das seqüências do projeto Cattle EST (Bos taurus) Procedimentos baseados no trabalho de Telles e da Silva, 2001 – “Trimming and clustering sugarcane ESTs” Simplificação dos métodos de detecção de vetor e de caudas poli-A/T Detecção de adaptador separada da detecção de vetor Algoritmo de subseqüência máxima para detecção de baixa qualidade Sem detecção de derrapagem Avaliação da Nova Estratégia Todos os tipos de artefatos foram detectados Análise dos artefatos produzidos mostrou: 2,46% de um total de 12.520 seqüências possuíam artefatos que não seriam identificados caso as etapas não fossem independentes Remoção de seqüências ribossomais também aplicável a mamíferos Clustering com maior grau de agrupamento de seqüências Menor número de singletons e clusters Derrapagem Artefato de seqüenciamento Três novos métodos Regiões ricas em grupos ecoados Trabalho de Telles e da Silva único na literatura Média Aritmética Média Geométrica Cobertura por Ecos Duas estratégias Sufixo Subseqüência Média Aritmética Média Geométrica Cobertura por Ecos Avaliação dos Métodos Testes Variação de parâmetros Processamento de seqüências do projeto SUCEST (291.689 ESTs) Melhor estratégia: Subseqüência Encontra artefatos na porção inicial da seqüência Melhor método: Cobertura por Ecos Menor perda de hits de BLAST Baixa Qualidade Baixa Qualidade Avaliação de dois algoritmos Janela deslizante Subseqüência máxima Variação exaustiva dos parâmetros Análise de 1950 execuções distintas Critério de seleção de alternativas 2,5% de erro médio (25 bases 5’) 5,0% de erro médio (25 bases 3’) 80,0% de preservação média do BLAST hit Baixa Qualidade Adição de procedimento para detecção de ilhas de baixa qualidade Janela deslizante Tamanho: 10 bases Erro médio de 20,0% no mínimo Reprocessamento dos fragmentos Melhor algoritmo: Subseqüência máxima Qualidade mínima 11 Procedimento de detecção de ilhas Novo conjunto de procedimentos Combinação dos resultados dos estudos realizados Nova estratégia Simplificação de métodos Adoção das decisões dos estudos de derrapagem e baixa qualidade Realização de testes com o conjunto completo de ESTs do projeto SUCEST SUCEST x Novo Conjunto Menor descarte de seqüências SUCEST Novo Conjunto 18,44% 12,97% Menor tamanho 641,57 ± 139,79 bp 472,05 ± 121,68 bp médio Maior qualidade média 27,74 ± 14,30 33,25 ± 14,78 SUCEST x Novo Conjunto Produção de clustering: 145.845 seqüências SUCEST: 118.991 seqüências 20.202 singletons e 16.394 contigs (36.596) Novo conjunto: 126.988 seqüências 22.479 singletons e 17.486 contigs (39.965) Melhores consistências interna e externa Menor redundância Redução do número de hits Full-Length Diminuição de SNPs e aumento de INDELs Conclusão Nova estratégia apresenta bons resultados Novas alternativas para detecção de derrapagem Estudo de baixa qualidade Análise aprofundada Adição de procedimento para detecção de ilhas de baixa qualidade Novo conjunto de procedimentos Trabalhos Futuros Estudo mais detalhados dos demais artefatos Estudo de outras variações de parâmetros dos algoritmos para detecção de derrapagem Estudo do efeito da variação de exigência da qualidade média sobre a construção de clusterings Aperfeiçoamento de algoritmos de clusterização de seqüências (Projeto de Doutorado) Trabalhos Publicados Nova estratégia C. Baudet and Z. Dias, “New EST trimming strategy”. In J.C. Setubal and S. Verjovski-Almeida, editors, Lectures Notes on Bioinformatics, volume 3594, pages 206-209. Springer-Verlag Berlin Heildelberg, July 2005. Brazilian Symposium on Bioinformatics 2005 (BSB2005), extended abstract. C. Baudet and Z. Dias, “New EST trimming strategy”, Technical Report: IC-05-09, Institute of Computing – University of Campinas, May 2005. Trabalhos Publicados Derrapagem C. Baudet and Z. Dias, “Analysis of slipped sequences in EST projects”. Genetics and Molecular Research, 5(1):169-181, 2006. C. Baudet and Z. Dias, “Analysis of slipped sequences in EST projects”. X-Meeting 2005, Caxambu – MG, October 2005, poster. Trabalhos Publicados Baixa qualidade C. Baudet and Z. Dias, “Low quality trimming on SUCEST ESTs”. X-Meeting 2006, Fortaleza – CE, August 2006, poster. Novo conjunto de procedimentos C. Baudet and Z. Dias, “New EST trimming procedure applied to SUCEST sequences”. 14th Annual International Conference On Intelligent Systems For Molecular Biology (ISMB2006), Fortaleza – CE, August 2006, poster.
Baixar