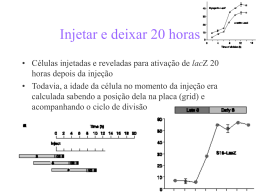
Modulação por íons cálcio da expressão do gene rfaL de Herbaspirillum seropedicae. Estevan Rafael Tomazini - Iniciação ciêntifica- PIBIC/ Fundação Araucária Rose Adele Monteiro; Marcelo Bueno Batista; Eduardo Balsanelli. Introdução: Resultados/Discussão: Herbaspirillum seropedicae é uma bactéria diazotrófica capaz de se associar à gramíneas através de reentracias nas raizes de forma não invasiva. Essa associação é dependente de LPS íntegro e sua biossíntese é dependente do gene rfal, modulado por íons cálcio. O objetivo desse trabalho é determinar a porção do gene rfaL que contem o sítio de modulação por cálcio em H. Seropedicae. Fonte: BALSANELLI, 2010. Fonte: Esse trabalho. Método: Para essa análise foram construídas 4 fusões plasmidiais: pLig4 (rfaL::lacZ integro), pLig2 (rfaL::lacZ com deleção de 200pb), pLig3 (rfaL::lacZ com deleção de 400pb) e pLig1 (rfaL::lacZ com deleção de 600pb), estas fusões foram transformadas em H. seropedicae. A expressão dessas fusões foi avaliada em ensaios de β-galactosidase com cultivo sob presença e ausência de cálcio. Referências: BALSANELLI, E. et al. Herbaspirillum seropedicae rfbB and rfbC genes are required for maize coloniza tion. Environmental Microbiology. v.12, n.8, p.2233–2244, 2010. Balsanelli et al. (2010) mostrou que os genes rfbB e rfbC envolvidos na biossíntese de ramnose, são induzidos por íons cálcio. Os dados obtidos nesse trabalho mostram uma baixa atividade de β-galactosidase em todas as condições e estirpes testadas. A expressão da fusão rfaL::lacZ contendo a região intacta não ocorreu da como observado nos experimentos realizados por Balsanelli em 2010. Conclusões: Esses experimentos devem ser repetidos com o objetivo de otimizarmos as condições do ensaio para que possamos determinar realmente o padrão de expressão das frações testadas.
Baixar