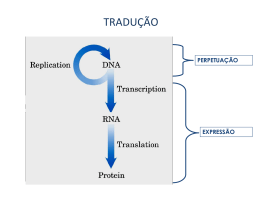
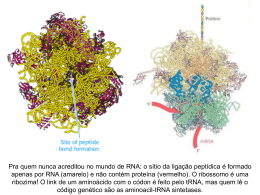
TRADUÇÃO SÍNTESE PROTEICA Fluxo da Informação Genética Código genético 3 bases nitrogenadas 1 amino ácido Marcos no metabolismo de proteínas • 1. ~1950- síntese proteica em pequenas partículas de ribonucleoproteínas (Paul Zamecnik- fracionamento celular, radioativo)- ribossomos • 2. aminoacil-tRNA sintetases- tRNA, ATP Hoagland & Zamecnick) • 3. hipótese do adaptador (tRNA, anticodon Francis Crick) Síntese proteica • ~300 moléculas envolvidas • ~90% da energia gasta nos processos biossintéticos • Bactérias: ~35% do peso: 20.000 ribossomos, 100.000 proteinas (fatores e enzimas), 200.000 tRNAs • Processo rápido: ~20 resíduos/seg • Erro: 1 a cada 10.000 aa adicionados Síntese e Processamento de Proteínas Transcrição Transcrito primário Processamento pós-traducional Processamento pós-transcricional mRNA maduro Tradução Dobramento Modificações covalentes nos aminoácidos Proteína (inativa) Proteína ativa Código genético (degenerado) Outras seqüências de mRNA podem especificar a mesma seqüência de aminoácidos Código genético alternativo em mitocôndrias Troca de “frame” Virus (sarcoma de Rous): gag e pol (1/20) • Gag: ... ACA AAU UUA UAG GGA GGG • Pol: .... ACA AAU UUAUA GGG AGG Código genético • Códon de iniciação- AUG (raro- GUG, UUG) • Códon de terminação- UAA, UAG, UGA RNAs Tipo Tamanho Função tRNA Pequeno Transporte de aa para o local de síntese rRNA Diversos Forma os ribossomos, juntamente com proteínas mRNA Diversos Determina a sequência de aa na prteína snRNA Pequeno Processa o mRNA inicial nos eucariotos miRNA Pequeno Afeta a expressão gênica (crescimento, desenvolvimento) siRNA pequeno Afeta a expressão gênica. Cientistas utilizam para bloquear a expressão do gene de interesse tRNA O ribossomo acomoda dois tRNAs carregados Etapas da síntese de proteínas 1. Ativação do aminoácido 2. Iniciação 3. Elongação 4. Terminação 5. Dobramento/processamento pós-tradução 1. Ativação do aminoácido Aminoacil tRNA sintetase ATP + aa aminoacil-AMP + Ppi aminoacil-AMP + tRNA aminoacil-tRNA + AMP Aminoacil tRNA sintetase 1. Ativação do aminoácido Aminoacil tRNA sintetase aminoacil-AMP Classe I Classe II aminoacil-AMP aminoacil-tRNA 1. Ativação do aminoácido Ligação do aminoácido ao tRNA Aminoacil t-RNA A etapa de ativação do aminoácido é determinante na fidelidade da tradução 2. Iniciação (bactéria) Ribossomo bacteriano reconhece uma sequência no mRNA- Shine Delgarno Nas bactérias, o primeiro amino ácido é formilmetionina fMet-tRNAf tem características que o distinguem como tRNA iniciador (tRNAi) Só tRNAi liga no sítio P e ao fator de iniciação (IF1 ou IF2) Eucariotos- primeiro aa é metionina Adição do grupo formil no metionil-tRNAf: dependente de ácido fólico tRNA para formil-metionina é diferente da tRNA para metionina H 3. Elongação AA2 Peptidil-transferase- 23S? Sítio E- bactéria Translocação 4. Terminação Proteínas de terminação (RFs): diferentes de acordo com o códon de terminação (bactérias) RFs- estrutura semelhante aos tRNAs, mas ligam em regiões distintas nos ribossomos Túnel de saída do ribossomo para a proteína- 12- 20 Å Polissomo :10-100 ribossomos Fatores proteicos necessários para iniciação da tradução Bactéria: IF-1 – Previne ligação prematura dos tRNAs ao sítio A IF-2 – Facilita ligação do fMet-tRNAf a subunidade 30S (liga GTP) IF-3 – Liga suunidade 30S e previne associação prematura da subunidade 50S. Aumenta especificidade do sítio P pelo fMet-tRNAf Eucariotos: eIF1- Liga mRNA eIF2 –Facilita ligação do Met-tRNAi a subunidade 40S (liga GTP) eIF2B, eIF3 – Ligam subunidade 40S/ mRNA eIF4A – Helicase de RNA para remover estrutura secundária do mRNA eIF4B – Liga mRNA, facilita varredura para localizar o primeiro AUG eIF4E – Liga 5´cap do mRNA eIF4G – Liga e IF4E e protéina que liga cauda poliA (PAB) eIF5 – Promove dissociação de outros fatores para facilitar associação de 60S eIF6 – Facilita dissociação de 80S inativo em 40S e 60S Processamento após (ou durante) a síntese de proteínas Dobramento (Folding) Clivagem proteolítica (incluindo aminoterminal) Modificações covalentes nos aminoácidos Degradação • Influencia a estrutura e função de proteínas 20 aminoácidos diferentes Modificações covalentes nos aminoácidos (modificações póstraducionais): Ex: Metilação Acetilação Hidroxilação Glicosilação Fosforilação ~200 aminoácidos Grupos prostéticos Acilação diferentes (modificados covalentemente) Fosforilação Modulação da atividade de proteínas, modulação de interações moleculares, sinalização celular Fosfo-serina Fosfo-histidina Fosfo-tirosina Fosfo-treonina Glicosilação Os carboidratos são ligados aos resíduos de aminoácidos através de ligações N- ou O- glicosídicas Endereçamento de proteínas Peptídeo sinal direciona proteínas secretadas e/ou glicosiladas para o retículo endoplasmático (ER) Síntese de proteínas acoplada ao ER Glicosilação Os carboidratos são ligados aos resíduos de aminoácidos através de ligações N- ou O- glicosídicas Puromicina: estrutura similar a extremidade 3’ do aminoacil-tRNA, ocorrendo a formação de peptidil-puromicina e interrupção da síntese proteica Inibição da síntese proteica por antibióticos e toxinas Estreptomicina Causa leitura incorreta dos códons e inibe iniciação Tetraciclina Bloqueia o sítio A do ribossomo bacteriano e inibe associação do aminoacil-tRNA Inibição da síntese proteica por antibióticos e toxinas Cloranfenicol Bloqueia atividade de peptidil transferase de ribossomos bacterianos e mitocondriais Cicloheximida Bloqueia atividade de peptidil transferase do ribossomo eucariótico Inibição da síntese proteica por antibióticos e toxinas Toxinas proteicas que inibem a tradução: Toxina da difteria: inativa o fator eEF2 (ADP-ribosila histidina) Ricina : inativa a subunidade 60S (depurina uma adenosina do rRNA 23S) Ribossomos Nucleoide (DNA compactado) Pili Flagelo Envelope celular (membrana) Ribossomos Informação genética- DNA nuclear nos eucariotos Núcleo Envelope nuclear Ribossomos Mitocondrias Cloroplasto A T G C T C A A Estrutura do DNA DNA fita dupla: cadeias antiparalelas Decifrando o Código Genético 1.Quantos nucleotídeos seriam necessários? 4 nucleotídeos diferentes no DNA Código de um nucleotídeo 20 aminoácidos diferentes na proteína = 4 combinações Código de dois nucleotídeos (42) = 16 combinações Código de três nucleotídeos (43) = 64 combinações Decifrando o Código Genético 2.O código não é superposto aa1 aa2 ABCDCD aa1 aa2 aa3 aa4 Análise da seqüência de aminoácidos de mutantes da proteína da capa do vírus mosaico do tabaco mostraram que o código não era superposto. A mutação em um nucleotídeo leva a mudança de um aminoácido e não de três aminoácidos Decifrando o Código Genético 3.O código não tem pausas Fase de leitura 1 Fase de leitura 2 Fase de leitura 3 5’ A U G C A C U U U A C U A A O código é lido sequencialmente sem pausas a partir do início determinado (3 fases de leitura são possíveis). Decifrando o Código Genético 4. O código é degenerado 64 códons para codificar 20 aminoácidos??? 61 códons codificam 20 aminoácidos. Para a maioria dos aminoácidos há mais de um códon 3 códons não codificam aminoácidos. São códons de terminação da síntese de proteínas Decifrando o Código Genético 5. Como o código foi decifrado? Tanto as sequências de bases do DNA com a de aminoácidos das proteínas eram desconhecidas!!! Nirenberg em 1961 mostrou que a adição de o RNA sintético poliuridilato (poli U) em um sistema de síntese proteíca livre de células (extrato de E.coli) levava a síntese de polifenilalanina: UUU = Phe Poli U foi sintetizado in vitro pela polinucleotídeo fosforilase... Ribossomo Direção da Transcrição Tradução Direção da Tradução - Síntese das proteínas da célula - Ocorre nos ribossomos Nas bactérias, transcrição e tradução estão acopladas....
Baixar