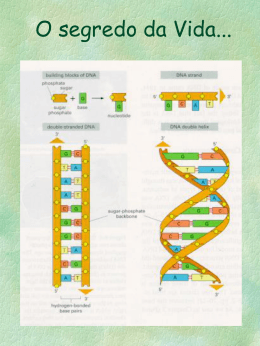
REVISTA MULTIDISCIPLINAR DA UNIESP 144 COMPARAÇÃO DE SEQÜÊNCIAS DE DNA PUCCI NETO, João1 Resumo: A comparação de seqüências é uma operação básica muito importante na área de biologia computacional. Neste trabalho, é implementado um algoritmo de comparação de seqüências de DNA, que pode auxiliar os biólogos em pesquisas de biologia molecular. O sistema tem como características principais: configurações do algoritmo como o valor do gap, valor do erro, valor do sucesso e valor agregado; comparação de similaridade; matriz de pontos (graficamente mostra a similaridade) e quadro de melhor alinhamento (onde é processado o melhor alinhamento para as duas seqüências entradas). Com esse trabalho, é possível realizar comparações de seqüências fornecidas pelo usuário ou adquiridas dos bancos de dados genéticos, como o GenBank por exemplo. Palavras-chave: Seqüências de DNA – Similaridade de Seqüências de DNA – Biologia Computacional. Abstract: The comparison of sequences is a very important basic operation in the area of biology computation. In this work, an algorithm of comparison of sequences of DNA is implemented, that can aid the biologists in researches of molecular biology. The system has as main characteristics: configurations of the algorithm as the value of gap, value of the mistake, value of the success and value attaché; similarity comparison; dot matrix (graphically exhibition the similarity) and the result of better alignment (where the best alignment is processed for the two sequences entrances). With that work, it is possible to accomplish comparisons of sequences supplied by the user or acquired of the genetic databases, like GenBanK for example. Key-Words: Sequences of DNA – similarity of DNA sequences – Computational Biology. Introdução Com a anunciada finalização do seqüenciamento do DNA humano pelos 18 países que participam do Projeto Genoma Humano, os três bilhões de pares de bases do genoma do homem estão mapeados. Com esse mapa, é possível identificar os genes humanos, que são cerca de 30 mil, e que representam cerca de 1% da molécula de DNA (os chamados exons). Os outros 99% são regiões no genoma que não apresentam funções definidas e são denominadas de introns (ZAHA, 2000). Os genes codificam SABER ACADÊMICO - n º 06 - Dez. 2008/ ISSN 1980-5950 REVISTA MULTIDISCIPLINAR DA UNIESP 145 proteínas, que são fundamentais para as funções biológicas da célula e conseqüentemente da vida. Identificar todas as proteínas e as suas interações pode auxiliar na cura, prevenção e redução de doenças, como o câncer por exemplo, que é resultado de "erros" genéticos que causam a perda do controle sobre o crescimento celular. Cerca de 2% dos recém-nascidos apresentam defeitos genéticos; 30% de todas as crianças e 10% dos adultos são internados em hospitais, pelo menos uma vez na vida, em função de distúrbios genéticos. Com o desenvolvimento do Projeto Genoma Humano, uma quantidade muito grande de seqüências de DNA foram depositadas em bancos de dados públicos. O Genbank, considerado o mais importante, é mantido pelo National Center of Biotecnology and Informations e possui aproximadamente três milhões de seqüências de genes expressos ou Expressed Sequence Tags (EST) de humanos. Um dos grandes desafios da ciência atualmente, é compreender como, a partir do código genético, a vida se estabelece. Para isso, é necessário conhecer o código genético humano e principalmente compreender como o código genético codifica as proteínas e de que forma estas estão relacionadas com as doenças, o que possibilitará o desenvolvimento de novas drogas para prevenção, cura e até mesmo a eliminação de algumas doenças (ZAHA, 2000). A seguir é apresentado um resumo básico de biologia molecular e como o genoma é estudado. Em seguida trataremos do algoritmo de similaridade e o sistema implementado é discutido e são mostrados alguns resultados. Conceitos Básicos da Biologia Molecular DNA O DNA é o principal armazenador da informação genética e consiste em duas longas cadeias polinucleotídicas compostas de quatro tipos de subunidades de nucleotídeos. Cada uma dessas cadeias é chamada de cadeia de DNA ou uma fita de DNA. Os genes carregam a informação biológica que deve ser precisamente copiada e transmitida quando uma célula se divide para formar duas células-filhas. O DNA SABER ACADÊMICO - n º 06 - Dez. 2008/ ISSN 1980-5950 REVISTA MULTIDISCIPLINAR DA UNIESP 146 codifica a informação na ordem, ou seqüência, dos nucleotídeos ao longo de cada fita. Cada base – A, C, G ou T – pode ser considerada como uma letra em um alfabeto de quatro letras que é utilizado para codificar as mensagens biológicas na estrutura química do DNA. Os organismos diferem um do outro porque as suas respectivas moléculas de DNA têm diferentes mensagens biológicas. (ZAHA, 2000). O conjunto completo de informações no DNA de um organismo é chamado de genoma. Na tabela a seguir, é comparado o tamanho do genoma de vários organismos de alguns animais e plantas. Animais/Plantas Lilium formosarum (lírio) Allium cepa (alho) Cebus apella (macaco) Canis familiaris (cão) Homo sapiens (homem) Genoma (em bilhões de pares de 36 18 3,6 3,3 3,2 RNA O RNA é outro ácido nucléico semelhante, no que diz respeito à sua estrutura, ao DNA, com exceção da substituição da ribose pela desoxirribose e da base Uracila (U) pela Timina (T). Existem três classes de ácido ribonucléico: o RNA mensageiro (mRNA), o RNA transportador (tRNA) e o ribossômico (rRNA). Todos estão envolvidos na síntese de proteína. (ZAHA, 2000). O Projeto Genoma Humano No genoma humano, a informação básica que se busca extrair de qualquer pedaço de DNA é sua seqüência de pares de bases. O processo de obter esta informação é chamado de seqüenciamento. Um cromossomo humano tem 108 pares de bases. Os SABER ACADÊMICO - n º 06 - Dez. 2008/ ISSN 1980-5950 REVISTA MULTIDISCIPLINAR DA UNIESP 147 pedaços maiores de DNA que podem ser seqüenciados no laboratório são muito longos em torno de 700 bp (MEIDANIS, 1997). Isto significa que há uma diferença de aproximadamente 105 entre a escala do que se pode atualizar e o tamanho real do cromossomo. Essas diferenças são um dos principais problemas encontrados na biologia computacional. O principal objetivo do estudo do genoma é descrever o conteúdo de cada cromossomo humano. Para se obter essas informações é necessário dividir os cromossomos em fragmentos menores que possam ser seqüenciados e caracterizados e depois ordenar esses fragmentos, de forma a corresponderem às suas respectivas posições nos cromossomos (mapeamento). (MEIDANIS, 1997). O Projeto Genoma Humano é um empreendimento internacional, iniciado formalmente em 1990 e projetado para durar 15 anos, com os seguintes objetivos: identificar e fazer o mapeamento dos cerca de 30 mil genes que supõe-se existir no DNA; determinar a seqüência dos 3 bilhões de bases químicas que compõem o DNA; armazenar essa informação em bancos de dados, desenvolver ferramentas eficientes para analisar esses dados e torná-los acessíveis para novas pesquisas biológicas. As últimas atualizações no Banco de Dados em 16 de Dezembro de 2001, trouxe como resultados o mapeamento genético - 10.986 genes foram mapeados. Além destes, o Banco de Dados do Projeto Genoma traz 1.183 genes sem identificação, formando um total de 12.169 genes. Dados esses encontrados na fonte http://www.gdb.org/gdbreports/CountGeneByChromosome.html. Banco de Dados de Seqüências Nas últimas décadas foram criados alguns bancos de dados para armazenar o grande seqüenciamento de DNA, RNA e proteínas e com isso também foram desenvolvidas técnicas computacionais para permitir uma procura rápida nesses bancos de dados (MEIDANIS, 1997). É importante frisar que esses bancos de dados do genoma, são bancos diferentes dos usados na área de informática que são por exemplo, do tipo relacional, SABER ACADÊMICO - n º 06 - Dez. 2008/ ISSN 1980-5950 REVISTA MULTIDISCIPLINAR DA UNIESP 148 como por exemplo o Interbase. No caso do genoma as informações são armazenados e atualizadas pelos biólogos em arquivos do tipo texto e que por eles são chamados de banco de dados (MEIDANIS, 1997). Principais bancos de dados representativos de seqüenciamentos: GenBank: é um banco de dados público de seqüências de nucleotídeos e proteínas com informações biológicas, produzido e distribuído pelo National Center for Biotechnology Information (NCBI), uma divisão do National Library of Medicine (NLM), localizado no campus da US National Institutes of Health (NIH); PIR-International: Protein Information Resource – International é um banco de dados de seqüências de proteínas que foi iniciado no National Biomedical Research Foundation (NBRF) no início do ano de 1960. Além dos dados da seqüência, o banco de dados contém informações que concentram: (1) o nome e a classificação da proteína e do organismo onde esta ocorre; (2) referências à literatura principal, incluindo informações sobre a determinação da seqüência; (3) as características funcionais e gerais da proteína; e (4) locais/sítios de interesse biológico dentro da seqüência. (LEMOS, 2000). Os registros do banco de dados possuem referências cruzadas para os bancos de dados originais. Conceitualmente o banco de dados consiste em três componentes principais: literatura, seqüência, e anotações da proteína. Algoritmo de similaridade entre duas seqüências Este algoritmo é baseado no método de programação dinâmica. Algumas extensões dele foram feitas para que o algoritmo se tornasse mais adequado para determinadas tarefas. (MEIDANIS, 1997) A complexidade quadrática do algoritmo básico e de suas extensões faz com que eles se tornem inviáveis em determinadas aplicações. Um exemplo importante desse tipo de aplicação é a busca em bases de dados moleculares. Algumas destas bases possuem milhões de seqüências. O problema típico dessa aplicação é a comparação de uma dada seqüência nova com todas as depositadas na base de dados. Isto significa que milhares de comparações precisam ser feitas. (MEIDANIS, 1997). SABER ACADÊMICO - n º 06 - Dez. 2008/ ISSN 1980-5950 REVISTA MULTIDISCIPLINAR DA UNIESP 149 Por este motivo, vários métodos mais rápidos surgiram. Geralmente eles são baseados em heurísticas. Alguns deles são baseados em uma janela da seqüência s, que é simplesmente um fator de s. A idéia desses métodos é que, se duas seqüências são parecidas, então elas terão muitas janelas em comum. Como as técnicas de seqüenciamento de nucleotídeos e proteínas têm melhorado muito, e conseqüentemente os repositórios desses dados estão crescendo, a aplicação de busca em bases de dados moleculares tem se tornado a cada dia que passa mais importante. Para auxiliar nessa busca utiliza-se famílias de algoritmos chamadas FAST e BLAST. Os programas dessas famílias estão baseados em heurística que trazem uma melhora significativa nos tempos de respostas das buscas em bases de seqüências. Idéia geral do algoritmo Existem dois tipos básicos de medidas de similaridade de seqüências e que normalmente são classificadas como globais ou locais. Algoritmos de similaridade global fazem a otimização do alinhamento total das duas seqüências, onde pode incluir grandes pedaços com baixa similaridade. Já os algoritmos de similaridade local se diferem pois apenas consultam seqüências. Uma simples comparação pode resultar em muitos alinhamentos de seqüências distintas e regiões não conservativas que não contribuem para se medir a similaridade. Medidas de similaridades locais são mais usadas em buscas em bancos de dados. A família FAST, como a BLAST, utiliza o método de similaridade local. Várias medidas de similaridades, inclusive a do BLAST, se iniciam com uma matriz de pontuações para todos os pares de resíduos. Identidades e substituições conservativas tem pontuações positivas, enquanto as substituições não comuns tem pontuações negativas. A pontuação de similaridade para dois segmentos alinhados do mesmo tamanho é a soma dos valores das similaridades para cada par de resíduos alinhados. Um biólogo molecular, pode por acaso estar com interesse em todas as regiões conservativas compartilhadas entre duas proteínas, e não só no par que tiver maior SABER ACADÊMICO - n º 06 - Dez. 2008/ ISSN 1980-5950 REVISTA MULTIDISCIPLINAR DA UNIESP 150 pontuação. Por este motivo, o BLAST faz uma busca em todos os pares de segmentos de máximo local com pontuações maiores que um valor limite. Passos do algoritmo Na implementação do BLAST, existem detalhes do algoritmo que mudam de acordo com o banco de dados que está sendo procurado (se ele é de seqüências de proteínas ou de DNA). A seguir três passos básicos independentes do banco de dados pesquisado, que são: 1. compilar uma lista de palavras de alta pontuação; 2. percorrer o banco de dados por acertos; 3. estender os acertos. Para se entender melhor cada passo será detalhado. Como entrada do algoritmo tem-se uma seqüência de consulta, que é um segmento ou um trecho de resíduos. Esta seqüência nova deve ser comparada a todas as outras seqüências do banco de dados. Esta comparação utilizará uma matriz de pontuação, como os algoritmos da família FAST. 1º Passo. Para cada palavra de tamanho w da seqüência de consulta, determinase quais palavras alinhadas com ela têm pontuação de no mínimo T. Dessa maneira é gerada uma lista de palavras para cada palavra de tamanho w da seqüência de consulta; 2º Passo. Para cada palavra de cada lista construída no 1º passo , percorre-se novamente o banco de dados até se encontrar os acertos relativos a tal palavra; 3º Passo. Para cada acerto encontrado, verifica-se se ele está dentro de um alinhamento cuja pontuação seja suficiente para ser notificada (pontuação deve ser maior ou igual a um valor S). Isto é feito estendendo o acerto em ambas direções, até que a pontuação de alinhamento atinja uma diferença de X abaixo da pontuação máxima já alcançada. Esses acertos estendidos são os chamados Pares Sequenciais Máximo’s (PSM). O 3º passo produz como saída os PSMs encontrados. Quanto menor o valor de T, menor a possibilidade de perder PSMs com a pontuação S requerida. No entanto valores baixos de T também aumentam o tamanho da lista de acertos geradas no 1º passo e conseqüentemente o tempo de execução e a memória necessária. Na prática o algoritmo deve se comprometer com valores de T e X para balancear as necessidades e a sensibilidade (ALTSCHUL, 1997). SABER ACADÊMICO - n º 06 - Dez. 2008/ ISSN 1980-5950 REVISTA MULTIDISCIPLINAR DA UNIESP 151 Metodologia do trabalho O objetivo principal é apresentar um algoritmo eficiente que, dadas duas seqüências, computa o melhor alinhamento entre elas. Tomando como exemplo a seqüência: GACGCATTAG e GATCGGAATAG. Elas são semelhantes, sendo que as únicas diferenças são um T a mais na segunda seqüência e uma troca de A por T na quarta posição da direita para a esquerda. Foi preciso introduzir um gap (buraco) na primeira seqüência para que as bases iguais antes e depois do gap se alinhassem nas duas seqüências. Um alinhamento pode ser definido como inserindo gap’s em pontos arbitrários ao longo das seqüências de forma que elas fiquem com o mesmo comprimento, e na disposição das duas cadeias resultantes uma em cima da outra de maneira que todos os caracteres ou gap’s de uma das cadeias seja comparado a um único caracter ou a um único gap na outra seqüência. Fora essas condições, não é permitido que um gap em uma das seqüências esteja alinhado com um gap na outra. E nem que, os gap‘s possam ser alocados no início ou no final das seqüências. Fornecido um alinhamento, pode-se atribuir uma pontuação. Cada coluna do alinhamento receberá um certo número de pontos. A pontuação total do alinhamento será a soma das pontuações. Através de um exemplo, será usado o seguinte esquema de pontuação: se a coluna tiver duas bases iguais receberá 1, se tiver duas bases diferentes receberá –1, e se possuir uma base e um buraco receberá –2. Esses valores são usados na prática (MEIDANIS, 1997). Procura-se valorizar bases iguais alinhadas e penalizar alinhamentos de bases desiguais e gaps. O alinhamento ótimo é o que tem a maior pontuação. A similaridade entre duas seqüências é o nome que se dá à pontuação máxima. Como demonstração do funcionamento do algoritmo será ilustrado o problema de obter o alinhamento ótimo para o par de seqüências: AAAC e AGC. Inicialmente tem-se uma idéia que se resume em analisar todas as hipóteses de alinhamento para a última coluna e identificar qual delas é o alinhamento ótimo. A seguir serão usadas três possibilidades para a última coluna: alinhar C com C, C com SABER ACADÊMICO - n º 06 - Dez. 2008/ ISSN 1980-5950 REVISTA MULTIDISCIPLINAR DA UNIESP 152 gap, ou gap com C; já que não é possível alinhar gap com gap. A própria ilustração a seguir mostra as possibilidades de alinhamento da última coluna: AAA AG AAAC AAA AG AGC Depois de encontrar todas as possibilidades, calcula-se a pontuação para cada uma delas, e faz-se o mesmo para o restante das seqüências. O raciocínio do esquema a seguir chega a um método recursivo: • calcule similaridade(AAAC,AGC); • calcule similaridade(AAA,AG); • calcule similaridade(AAAC,AG); • calcule similaridade(AAA,AGC); • escolha o maior dentre os valores acima. Como maior problema, este método, se aplicado diretamente, vai criar um número exponencial de chamadas recursivas, e com isso muitas destas chamadas são redundantes. Não é preciso fazer o cálculo mais de uma vez em uma comparação, basta que os resultados sejam guardados de forma que possam ser consultados rapidamente. Usando a primeira chamada da recursão e as seqüências originais AGC e AAAC é possível calcular o valor do alinhamento ótimo delas. Na célula estará apresentado o valor da pontuação da comparação entre as duas subseqüências. A pontuação de qualquer célula da matriz depende da pontuação de suas vizinhas. As células vizinhas à esquerda e acima da célula que está sendo calculada a pontuação devem ter suas pontuações somadas a (-2), assim como o segundo e o terceiro filho do nó está sendo calculado a pontuação. A célula vizinha n diagonal esquerda, deve ter sua pontuação somada a (-1) ou a (+1). Obtendo as pontuações de todas as células calculadas torna-se como opção calcular o alinhamento ótimo. Para ocorrer isto, é preciso percorrer as células da matriz. Complexidade O algoritmo básico preenche a matriz com os valores das pontuações, e possui quatro malhas tipo for. As duas primeiras malhas, gastam tempo O(m) e O(n), respectivamente. As duas últimas malhas são encaixadas e preenchem a matriz. (LEMOS, 2000). É ele que calcula o alinhamento a partir da matriz gasta tempo O(t), SABER ACADÊMICO - n º 06 - Dez. 2008/ ISSN 1980-5950 REVISTA MULTIDISCIPLINAR DA UNIESP 153 onde t é o tamanho do alinhamento retornado. Neste algoritmo, há basicamente uma chamada recursiva por coluna de alinhamento, e cada chamada consome tempo O(1). A complexidade do algoritmo básico é quadrática, o que dificulta sua aplicação para seqüências muito longas. Conclusão O objetivo principal deste trabalho foi a apresentação de algoritmos clássicos de comparações de seqüências de DNA. A complexidade de tempo destes algoritmos é quadrática, o que faz com que eles se tornem inviáveis para algumas aplicações, como a comparação de uma seqüência nova com todas as seqüências armazenadas em um banco de dados. Por tal motivo, alguns métodos surgiram, como as famílias de algoritmos FAST e BLAST. Os programas destas famílias estão baseados em heurísticas, que traz grande melhora nos tempos de respostas das buscas em bases de seqüências. Atualmente as famílias de algoritmos FAST e BLAST são as mais utilizadas pelos biólogos e, por isso melhoras nestes algoritmos ou em estruturas de dados do banco de dados que facilitem ainda mais a execução destes algoritmos são muito importante. Este trabalho teve a finalidade de introduzir a área de bioinformática no curso de Ciência da Computação, implementando um algoritmo de comparação de seqüências global e uma visualização gráfica do mesmo. REFERÊNCIAS BIBLIOGRÁFICAS ALTSCHUL, S.F., MADDEN, T.L., SCHÄFFER, A.A. et al. "Gapped BLAST and PSI-BLAST: a new generation of protein database search programs". Nucleic Acids Research, [s,l], v.25, n.17, p.3389-3402, jun./jul. 1997. LEMOS, Melissa Algoritmos para análises de seqüências. Rio de Janeiro, 2000. 74p. Monografia (Bacharel em Ciência da Computação) – Departamento de Informática, PUC-Rio, 2000. SABER ACADÊMICO - n º 06 - Dez. 2008/ ISSN 1980-5950 REVISTA MULTIDISCIPLINAR DA UNIESP 154 MEIDANIS, João, SETUBAL, João Carlos. Introduction To Computational Molecular Biology. 1.ed. [s,l] : IE-Thomson, 1997. THE HOSPITAL FOR SICK CHILDREN, TORONTO, ONTARIO - CANADA. Count of Mapped Genes by Chromosome. 2001. Disponível em: < http://www.gdb.org /gdbreports/CountGeneByChromosome.html>. Acesso em: 14 Dez. 2001. ZAHA, Arnaldo; Biologia Molecular Básica. 2.ed. Porto Alegre: Mercado Aberto, 2000. 336p. 1 Especialista em Docência no Ensino Superior e Docente do curso de Sistemas de Informação da Faculdade de Presidente Prudente (UNIESP). SABER ACADÊMICO - n º 06 - Dez. 2008/ ISSN 1980-5950
Baixar