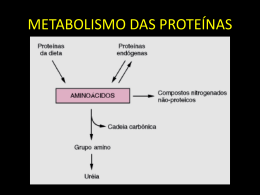
Os cavaleiros medievais usavam armadura mas nem por isso eram Ecdysozoa: o uso de aminoácidos essenciais nos proteomas do verme, da mosca e do homem Hipótese Ecdysozoa origem monofilética para artrópode e nematóide 18S rRNA Adoutte A, Balavoine G, Lartillot N, de Rosa R, 1999. Hipótese Coelomata e ramificação entre protostomia e deuterostomia Hernán Dopazo and Joaquín Dopazo, 2005 disputa técnica Hipótese Coelomata -protostomia -deuterostomia nematóides Hipótese Ecdysozoa artrópodos cordados É certo que alguns aminiácidos são requeridos na dieta de moscas e humanos • Apesar de bastante claro nos livros de bioquímica • Dados experimentais não são muitos • Todavia: IFKVWLTM = R NYDSEHGAPQC = EAA NEAA É certo que aminoácidos podem ser trocados sem nenhum efeito na função da proteina • Substituições conservativas dentro de domínios • Substituições quase livres em largos trechos de estrutura estendida • Então: – Que tal trocaralguns EAA por NEAA tanto quanto possível em organismos que requerem EAA na dieta? – Logo, homens e moscas poderiam um dia vir a usar mais NEAA que leveduras e plantas – Ou não... ...Demorou testar? Exemplos do número de proteínas e AA disponíveis Database KOG REFSEQ Organism # proteins # aa hsa 26,324 12,112,669 dme 10,517 5,520,517 cel 17,101 7,798,531 ath 24,154 10,547,967 sce 4,841 2,535,856 spo 4,233 2,014,184 Total KOG 87,170 40,529,724 hsa 27,978 13,993,476 dme 18,759 10,563,712 cel 21,136 9,221,006 ath 29,157 12,120,468 sce 5,868 2,913,021 spo 5,010 2,269,364 107,908 51,081,047 Total RefSeq A set of amino acids found to occur more frequently in human and fly than in plant and yeast proteomes consists of non-essential amino acids Francisco Prosdocimi, Maurício A. Mudado and J. Miguel Ortega Computers in Biology and Medicine, 2006 Nu _ of _ EA EI ( Tota _ num _ of _ aa ) ( N _ o _ R ) The first analysis performed was simply the calculation of EAA percentage in the proteome • A porcentagem de EAA foi determinada para cada proteína RefSeq, ou para cada cluster KOG, UniRef50 ou KEGG • A média da porcentagem de EAA das várias entradas foi calculada • O desvio padrão foi alto: 5-7%, todavia o erro padrão é baixo • Hsa e Dme apresentam menor conteúdo de EAA Percentage of KOGs with a higher EI (voting) hsa dme cel ath sce spo hsa higher - 53.27% 35.31% 45.28% 34.00% 39.54% dme higher 2000 (4280) - 31.88% 44.98% 32.69% 38.44% cel higher 2689 (4157) 2724 (3999) - 60.76% 46.18% 53.34% ath higher 1670 (3052) 1568 (2850) 1087 (2770) - 36.89% 42.83% sce higher 1613 (2444) 1538 (2285) 1213 (2254) 1466 (2323) - 58.51% spo higher 1535 (2539) 1456 (2365) 1084 (2323) 1372 (2400) 1033 (2490) - KOG database presented the highest number of shared clusters (42,531 tuples); UniRef and KEGG clusters yielded 4,989 and 11,623 tuples, respectively. Hsa-Ath most different KOGs in essentiality index (EI) KOG DIFF* Description KOG4752 38% (J) Ribosomal protein L41 KOG0002 24% (J) 60s ribosomal protein L39 KOG3491 19% (S) Predicted membrane protein KOG3445 17% (J) Mitochondrial/chloroplast ribosomal protein 36a KOG3500 15% (C) Vacuolar H+-ATPase V0 sector, subunit M9.7 (M9.2) KOG1793 14% (S) Uncharacterized conserved protein KOG4293 14% (T) Predicted membrane protein, contains DoH and Cytochrome b561/ferric reductase transmembrane domains KOG3423 14% (K) Transcription initiation factor TFIID, subunit TAF10 (also component of histone acetyltransferase SAGA) KOG2346 13% (S) Uncharacterized conserved protein Hsa KOGs with highest essentiality index (EI) KOG Description KOG1721 FOG: Zn-finger KOG0613 Projectin/twitchin and related proteins KOG3594* FOG: Cadherin repeats KOG3656* FOG: 7 transmembrane receptor KOG3544* Collagens (type IV and type XIII), and related proteins KOG2177 Predicted E3 ubiquitin ligase KOG0619 FOG: Leucine rich repeat KOG0516* Dystonin, GAS (Growth-arrest-specific protein), and related proteins KOG3595* Dyneins, heavy chain KOG1217* Fibrillins and related proteins containing Ca2+-binding EGF-like domains * ausentes em Ath E se queimássemos o Lehninger? % A _ U M PR lo AA 2 % AA _ U N A preference ratio (PR) was defined as the percentage of each amino acid in MO divided by its percentage in NMO, normalized by log2 • A porcentagem de uso de cada AA nas bases RefSeq e KEGG foi determinada para cada organismo • Metazoários foram comparados com não metazoários par a par • O log na base 2 da razão das porcentagens foi calculado • Os resultados foram clusterizados • Um grupo aminoácidos foi selecionado (SAA): R, H, G, A, P, QeC • Comparações par a par entre homem/planta ou homem/levedura apresentam um viés de enriquecimento de NEAA (símbolos cheios) dentre os AA mais freqüentes no organismo metazoário • Comparações par a par entre homem/outra levedura e mosca/levedura também apresentam um viés de enriquecimento de NEAA (símbolos cheios) dentre os AA mais freqüentes no organismo metazoário • Comparações par a par envolvendo o verme naum apresentam o mesmo viés de utilização diferenciada de AA, nem o viés de NEAA dentre os pouco mais freqüentes N _ o _ S S ( T _ n _ o _ a ) We evaluated the percentage of the proteomes that consisted of these selected amino acids (SAA) • O viés de uso de SAA por Hsa e Dme é ainda maior (cerca de 5% em comparação a cerca de 2%) e o verme não apresenta esse efeito 3Percentage of KOGs with higher selected index (SI) hsa higher dme higher cel higher ath higher sce higher spo higher hsa dme cel ath sce spo - 56.10% 75.90% 74.30% 91.00% 84.52% 1879 - 72.12% 70.78% 89.37% 80.90% 1001 1116 - 49.26% 81.75% 68.55% 785 834 1406 - 82.31% 67.97% 220 243 412 411 - 26.67% 393 452 731 768 1823 - • Um grupo de aminoácidos (SAA) é mais freqüente no homem e na mosca, favorecendo a hipótese Coelomata em detrimento de Ecdysozoa • A separação entre Protostomia e Deuterostomia é precoce nos celomados Resumo We compared the amino acid composition in human, worm and fly proteomes, organisms that cannot synthesize all amino acids, with the amino acids of the proteomes of plant, bakers yeast and budding yeast, which are capable of synthesizing them. The analysis covered 212,197,907 amino acids. Conclusão The data suggest a bias towards the usage of nonessential amino acids (mostly the set GAPQC) by metazoan organisms, except for the worm, a Pseudocoelomata. Our results support the hypothesis that non-essential amino acids have been substituting essential ones in the Coelomata clade. Perspectivas •Comparação entre domínios e extra-domínios •Comparação entre proteínas novas e velhas •Comparação entre proteínas muito e pouco expressas •Investigação detalhada dos taxa
Baixar