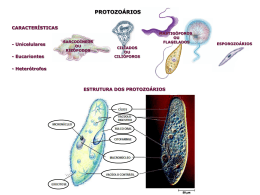
O BANCO DE MOLÉCULAS DA AMAZÔNIA E SUA APLICAÇÃO CONTRA O ALVO ENZIMÁTICO DA DOENÇA DE CHAGAS Isabella Menezes QUEIROZ1; [email protected] Ana Alice Farias da COSTA1; [email protected] Krisnna Mariana Aranda ALVES1; [email protected] Jaqueline Bianca Carvalho DUARTE2; [email protected] Fábio José Bonfim CARDOSO1; [email protected] Ana Paula da Silva FONSECA1; [email protected] Fábio Alberto de MOLFETTA1; [email protected] 1 Faculdade de Química, ICEN, Universidade Federal do Pará Instituto de Tecnologia (ITEC), Universidade Federal do Pará. 2 RESUMO: Estimam-se que existam de 6 a 7 milhões de pessoas infectadas em todo o mundo pela doença de Chagas, sendo que os fármacos disponíveis no mercado, o Nifurtimox e Benzonidazol, apresentam elevada toxidez e ineficácia na fase crônica da doença. A enzima cruzaína foi escolhida para ser utilizada como alvo biológico, para produção de novos fármacos, pois está envolvida em todo o ciclo de vida do parasito. O Banco de Moléculas da Amazônia (BMA), criado pelo Laboratório de Modelagem Molecular da UFPA, com compostos obtidos de produtos naturais da região amazônica. O método de docagem molecular foi feito utilizando as estruturas do BMA, para assim selecionar moléculas potencialmente ativas contra a cruzaína. Os cálculos de docagem foram realizados com os programas AutoDock Vina 1.1.0 e DOCK 6.3. Com base nos valores de energias e análise consensual foram selecionadas 20 estruturas. Sendo feita a verificação da presença de grupos tóxicos nas moléculas. E por fim foi verificado as possíveis interações com o alvo biológico. Com isso foi selecionada a estrutura (-)-Anonaina, que apresentou duas importantes ligações de hidrogênio com os resíduos de aminoácido Trp26 e Cys25. Estes resíduos pertencem ao sítio ativo da cruzaína. Com isso a estrutura selecionada foi submetida à dinâmica molecular, através do programa fDynamo, onde foram calculados 5.000 picosegundos, e foi comprovada a estabilidade da estrutura selecionada com a enzima cruzaína, uma vez que seus valores de RMSD estabilizaram durante a simulação. Com isso, a (-)-anonaina, mostra-se promissora para o tratamento da doença de Chagas. Palavras-chave: Cruzaina, Docagem, Dinâmica Molecular. 1. INTRODUÇÃO As doenças infecciosas parasitárias afetam milhões de pessoas nas diferentes regiões geográficas mais pobres do planeta e representam uma crescente ameaça mundial. As doenças parasitárias continuam sendo um obstáculo para o desenvolvimento social e econômico dos países mais pobres, sendo que a maioria absoluta das mortes devido às doenças parasitárias ocorre em regiões situadas abaixo da linha do Equador (DIAS et al., 2009). A doença de Chagas ou tripanossomíase americana é uma infecção causada pelo protozoário flagelado Trypanosoma cruzi (T. cruzi). Segundo a Organização Mundial de Saúde (OMS), a doença atinge aproximadamente 6 a 7 milhões de pessoas em todo o mundo, no qual a América Latina é região endêmica da doença, onde atualmente mais de 25 milhões de indivíduos vivem em áreas de risco de infecção, causando cerca de 10.000 mortes por ano. Tal doença na sua fase crônica causa deficiências significativas, tendo um alto impacto social e econômico, incluindo desemprego e diminuição de capacidade produtiva. (DIAS e DESSOY et al., 2013; VALDÉZ et al., 2012; OMS, 2015; ESPERANDIM et al., 2013; DNDi América Latina, 2014). A propagação da parasitose coincide com a distribuição dos vetores triatomíneos, que se estende do sul dos Estados Unidos da América (EUA) a Patagônia Argentina. No entanto, a principal área endêmica da doença de Chagas abrange cerca de 20 países da América Latina (do México até o norte 744 da Argentina) e um número significativo de casos de infecção por vetores foi relatado no sul dos EUA (DORN e cols, 2007). Apesar disso, milhares de pessoas atingidas podem estar vivendo na América do Norte, Europa e em outros países, como resultado da migração de pessoas infectadas para essas áreas (COURA E VIÑAS, 2010). As formas de transmissão são por via oral, transfusão sanguínea, transplante de órgãos, congênita e a mais conhecida, e também responsável pela maior parte dos casos de infecção em seres humanos e outros vertebrados, ocorre via fezes ou urina do inseto triatomíneo hematófago infectado, conhecido vulgarmente como “barbeiro”, que pode ser inoculada dentro da pele pela picada do inseto ou através de feridas ou escoriações causadas pelo ato de coçar (ESPERANDIM, 2010). O ciclo biológico do T. cruzi que envolve um hospedeiro vertebrado, humanos e outros mamíferos, e um invertebrado, geralmente os triatomíneos, que pode ser encontrado nas seguintes formas: amastigotas, epimastigotas e tripomastigotas, onde esta última é a forma infectante da doença (TEIXEIRA et al., 2012). Para o tratamento da doença há apenas dois fármacos disponíveis: Nifurtimox e Benzonidazol, comercializados com os nomes de Lampit® e Rochagan®, respectivamente (COURA, 2005). Estes dois fármacos desenvolvidos na década de 1970, são eficazes em curar a infecção principalmente na fase aguda, no qual apresenta cura para esta fase em torno de até 80%, mas são quase ineficazes em pacientes cronicamente infectados. Além disso, devido aos seus efeitos colaterais, como elevada toxidez, o Nifurtimox não é mais comercializado na maioria dos países latino-americano (COURA E DE CASTRO, 2002). Além dos efeitos colaterais graves da quimioterapia disponível, tem sido observado resistência em algumas cepas dos tripanossomatídeos (URBINA E DOCAMPO, 2003). Assim, é de extrema importância o investimento em programas de pesquisa e desenvolvimento (P&D) para a descoberta de novos medicamentos mais seguros e eficazes no tratamento da doença de Chagas. (WIGGERS et al., 2013). A cruzaína (ou cruzipaína) é uma enzima importante e está envolvida na infecção das células hospedeiras no processo de replicação e no metabolismo de todo o ciclo de vida do parasito. A inibição seletiva desta enzima bloqueia a replicação, diferenciação e o crescimento do organismo, além de ser fundamental dentro das células para o seu desenvolvimento e sobrevivência (GIAROLLA E FERREIRA, et al., 2013; WIGGERS et al., 2011). A enzima cruzaína é um alvo validado e por esse motivo uma grande busca por inibidores foi realizada por vários grupos de pesquisa em todo o mundo nos últimos vinte anos. Assim, a enzima cruzaína torna-se um alvo interessante para o desenvolvimento de inibidores enzimáticos com elevado potencial de desenvolvimento clínico (WIGGERS et al., 2011; LOZANO et al., 2012). Por esse motivo, o alvo biológico utilizado foi a enzima cruzaína, principal cisteíno protease presente no T. cruzi. O sítio ativo dessa enzima possui quatro subsítios de ligação, denominados S1, S2, S3 e S4 (Figura 1). O subsítio S1 é onde se localiza a tríade catalítica da enzima, o S2 é o principal responsável pela especificidade da enzima, sendo que esse subsítio é delimitado pelas cadeias laterais dos resíduos hidrofóbicos LEU67, MET68, ALA133, LEU157, GLY160 e GLU205, que é essencial para a especificidade da enzima (BOURGUIGNON et al., 2011), e os subsítios S3 e S4 são mais superficiais e possuem também características hidrofóbicas (WIGGERS et al., 2011). Figura 1. Subsítios da enzima Cruzaína. Fonte: WIGGERS et al., 2011. 745 O processo de descoberta de novos fármacos é fundamentado no conhecimento da estrutura e das propriedades de fármacos ativos contra um alvo biológico (SÁNCHEZ-QUITIAN, 2010). Neste trabalho, foram utilizados os métodos de docagem molecular, pois este é frequentemente aplicado na busca de novos fármacos, no qual reduz o número de moléculas a serem submetidas a testes experimentais, se tornando um método chave na química computacional (PATIL, 2013). Neste estudo, os cálculos de docagem molecular usaram o Banco de Moléculas da Amazônia (BMA), este banco possui atualmente 866 compostos provenientes de produtos naturais extraídos de espécies vegetais da Região Amazônica, área detentora da mais rica biodiversidade no mundo e importante fonte de componentes terapeuticamente eficazes (CALDERON et al., 2009). Dessa forma, o Laboratório de Modelagem Molecular (LMM) da Universidade Federal do Pará criou em 2009 e vem implementando este banco, com a finalidade de reunir compostos orgânicos oriundos da região, para realizar triagens virtuais de moléculas que possam apresentar atividade inibitória em doenças negligenciadas, tais como a doença de Chagas, Malária e Leishmaniose. Com as estruturas melhores pontuadas no ensaio virtual, foram efetuados cálculos de dinâmica molecular para selecionar estruturas mais promissoras em inibir a enzima cruzaína. Desta forma, contribuir com o desenvolvimento de novos fármacos que auxiliem na terapia contra a doença de Chagas. 2. OBJETIVO Selecionar compostos oriundos do BMA que sejam seletivos contra a enzima cruzaína da Doença de Chagas; Realizar cálculos de Dinâmica Molecular com a estrutura selecionada através do programa computacional fDynamo; Avaliar os resultados das interações dos resíduos da enzima com a molécula selecionada através da dinâmica molecular observando os valores de RMSD. 3. METODOLOGIA 3.1. Banco de moléculas da Amazônia O Brasil possui uma biodiversidade extremamente rica no qual reúne aproximadamente 20% de todas as espécies do planeta. A Amazônia tem um enorme potencial para a produção de conhecimentos e de produtos com valor agregado, incluindo medicamentos naturais ou derivados, suplementos alimentares, cosméticos e materiais para controle de pragas e parasitas agrícolas (VALLI, 2013). A região Amazônica possui grande potencial como fonte de novos fármacos, devido sua enorme biodiversidade ou quimiodiversidade (PAZ-Y-MIÑO et al., 2012), no qual os produtos naturais são importantes fontes de compostos biologicamente ativos e muitos deles constituem modelos para síntese de vários fármacos (VALLI, 2013). O BMA dispõem de uma diversidade química baseada nas diferentes classes de compostos, como quinonas, flavonóides, alcalóides e outras classes. Atualmente, o BMA possui 866 moléculas de produtos naturais da Amazônia. Neste estudo o programa Instant JChem 6.1 (ChemAxon, 2013) foi utilizado para a construção do BMA, uma vez que este viabiliza o armazenamento e posterior recuperação das estruturas. Deste modo, a partir dessas informações é possível verificar a possibilidade da síntese de estruturas para futuros testes biológicos. 3.2. Docagem Molecular Os métodos de docagem molecular receptor-ligante são métodos computacionais para a identificação do modo de ligação de moléculas candidatas a fármacos, no sítio ativo de proteínas consideradas como alvos moleculares para o tratamento quimioterápico de doenças. Estes métodos são importantes no planejamento de fármacos baseados em estruturas e possuem grande potencial para auxiliar na descoberta de novos medicamentos (MAGALHÃES, 2006) 746 A docagem molecular é uma importante ferramenta computacional na triagem in silico para a descoberta de novos fármacos, uma vez que auxilia na seleção de compostos promissores como ligantes de alvos terapêuticos de interesse, sejam eles agonistas ou antagonistas de receptores, ou ainda inibidores enzimáticos (WINK, 2012; RODRIGUES, 2012). Os protocolos de docagem são descritos como a combinação de duas componentes: as funções de pontuação e de busca. As funções de pontuação são usadas para prever o encaixe, ou seja, a afinidades de ligação dos ligantes com a proteína, enquanto que as funções de busca geram as possíveis conformações dos ligantes (RODRIGUES et al., 2012). Porém, os resultados obtidos podem variar de acordo com as características do receptor e do programa escolhido (FREITAS, 2009). 3.3. Dinâmica Molecular As simulações de dinâmica molecular são ferramentas importantes para uma melhor compreensão da flexibilidade da enzima e amostras conformacionais relevantes para planejamento de fármacos (DURRANT et al., 2009). O método de dinâmica molecular utilizado neste trabalho é dividido em três partes, uma região tratada com mecânica quântica (QM), uma região tratada como mecânica molecular (MM), mediante os campos de força da mecânica molecular clássica, e a outra região de contorno, que corresponde a um conjunto de restrições a serem aplicadas. Com isso, as simulações de dinâmica molecular combinada com o método híbrido QM/MM tem sido muito utilizadas para estudos de interação inibidores/proteínas e de mecanismo catalítico enzimático (CARNEIRO et al., 2011). Para realização das simulações de dinâmica molecular foi utilizada a livraria computacional fDynamo (FIELD, 2000), esta livraria aplica os potenciais híbridos da mecânica quântica (QM) e mecânica molecular (MM). O programa fDynamo permite realizar cálculos AM1, usar o campo de força OPLS-AA e o modelo de solvatação explicito TIP3, ele realiza cálculos de energia, otimização de geometrias e pesquisa de estruturas de transição. Além disso, utiliza o método híbrido QM/MM. Para visualização das estruturas foi utilizado o programa VMD (HUMPHREY et al, 1996) pois este possui uma boa interface gráfica. 4. RESULTADOS E DISCUSSÃO A partir do BMA foi feita a triagem virtual no qual as estruturas foram submetidas à docagem molecular nos programas AutoDock Vina 1.1.0, e Dock 6.3. Estes programas apresentam métodos diferentes na abordagem dos ligantes, com diferentes funções de busca e pontuação. Após a realização da análise consensual baseados nos valores de energia foram selecionadas 20 estruturas do BMA. Foram encontradas nove estruturas com massa molar acima de 500 Daltons, e segundo a regra de Lipinski e colaboradores (Regra dos Cinco) (LIPINSKI et al. 1997) um fármaco ativo de administração oral não deve violar essa condição. Dessa forma, essas estruturas foram excluídas dos compostos selecionados. Também foi analisada a toxidez das moléculas selecionadas por meio do servidor OChem (SUSHKO, 2012). Esse servidor faz a análise se uma determinada estrutura apresenta grupos tóxicos. A análise de toxidez se faz necessário, pois muitos compostos são excluídos durante o processo de descoberta e desenvolvimento de fármacos devido a efeitos indesejados ou de toxidez apresentados por essas estruturas (DINGERMANN et al, 2004). Os compostos que não apresentaram grupos tóxicos segundo o programa foram submetidos a análise através da ferramenta computacional LigPlus 1.4.5 para verificação de interações com os resíduos chave da enzima Cruzaína. Com isso, foram selecionadas duas estruturas, os compostos 314 e 383. Em seguida, realizou-se a docagem com os dois ligantes selecionados, usando o AutoDock Vina 1.1.0 e o DOCK 6.3, na Catepsina L (código PDB 1MHW), que é a enzima homóloga humana, como forma de testar se os ligantes apresentavam atividade contra tal enzima. Após a docagem na Catepsina L, verificou-se através do programa LigPlus, que o ligante 314 não fez interações de hidrogênio com tal enzima, onde o mesmo não ocorreu com o ligante 383, ocasionando no descarte 747 deste último ligante. Desta forma, foi selecionado o ligante 314 que apresentou interações com a enzima cruzaína (Figura 2) e não com a homóloga humana, a Catepsina L. Um dos representantes mais importantes das classes cisteíno-proteases, são as catepsinas. A cruzaína, uma cisteíno-protease da família de catepsina L, é considerada a protease mais abundante em T. cruzi, sendo expressa em todos os estágios do parasito e apresenta ampla especificidade catalítica (MOURA, 2011). A partir disto, busca-se um composto no qual não apresente interação com a Catepsina L, para que não ocorra sua inibição e prejudique suas atividades benéficas para o ser humano, tais como a perda da sua ação no sistema imunológico. Figura 2: Ligações de hidrogênio (em verde) entre o ligante 314 com a enzima 1ME3, no qual o ligante faz ligações de hidrogênio com os resíduos de Trp26 e Cys25. Figura obtida através do programa LigPlus [PETTERSEN, et al, 2004]. A estrutura selecionada (ligante 314) foi analisada através do programa LigPlus 1.4.5 (LASKOWSKI, 2011) para verificar as possíveis interações de hidrogênio entre receptor-ligante. Através dos resultados obtidos foram observadas duas importantes ligações de hidrogênio com os resíduos de aminoácidos Trp26 e Cys25 localizado no subsítio S1, que pertence ao sítio ativo da enzima cruzaína (Figura 2). Segundo Moreira e colaboradores estes resíduos mostram-se importantes para estabilização do ligante no sítio da cruzaína, por pertencerem ao subsítio S1, região no qual se encontra a tríade catalítica da enzima (MOREIRA et al. 2014). Além disso, pesquisas revelam que o ligante selecionado 314 o (-)-Anonaina apresenta atividade como antidepressivo, antioxidante, antifúngico, além da ação sobre o Plasmodium falciparum (HSING-TAN LI et al.,2013) que nos abre a possibilidade que ele possa atuar com ação tripanossomicida através da enzima cruzaína. Após a seleção da estrutura 314, foi feito o estudo in silico do comportamento da enzima e do ligante no meio biológico, por meio de dinâmica molecular. Por esse método, é possível simular a dinâmica do complexo enzima-ligante em solução, e verificar se as interações de hidrogênio com os resíduos de aminoácidos observadas pelo método de docagem molecular são conservadas e fortes o suficiente para manter a pequena molécula complexada na macromolécula em uma situação estável 748 no sistema. Além disso, permite observar possíveis modificações conformacionais na presença do ligante complexado (SARTORI, 2012). Após cálculos de dinâmica molecular a análise do RMSD (Root Mean Square Desviation) foi realizada a partir do ligante selecionado, cujo período de simulação foi equivalente a 5000 picosegundos, como mostrado na Figura 3. Figura 3: Gráfico de RMSD para o sistema, enzima Cruzaína complexada com o ligante 314, a (-)Anonaina. Ligante 314 A Figura 3 mostra dos valores de RMSD para o ligante 314 (-)-anonaina, onde pode-se acompanhar o aumento gradual até a estabilização do ligante no sitio ativo da enzima cruzaína, que ocorre a partir de 2500 picosegundos. Através dos resultados do gráfico de interação por resíduo, observa-se a preservação da ligação com o resíduo de aminoácido Cys25 obtido através da docagem, como pode-se ver na Figura 4. Figura 4: Contribuição individual dos resíduos de aminoácidos para o ligante 314, (-)-anonaina, pelo método híbrido QM/MM, onde os valores apresentados estão em kJ/mol. A Figura 4 apresenta a contribuição individual de cada resíduo para energia de interação total enzima-inibidor durante os últimos 500 picosegundos de simulação. A partir desse gráfico pode-se observar que o resíduo Asp158 apresentou valor positivo, ocasionando repulsão, enquanto que o resíduo Cys25 apresentou valor negativo, o que indica interação. De acordo com a literatura, a Cys25 é um importante resíduo para estabilização da molécula, fixando o inibidor no sitio ativo da enzima, pois faz parte do subsítio S1, onde se encontra a tríade catalítica da enzima cruzaína (WIGGERS et al., 2011). Segundo Chen, o resíduo de aminoácido Asp158 está localizado no subsítio S2, onde há a presença de doadores e aceptores de hidrogênio entre os subsítios S1 e S2 (CHEN, 2008). O subsítio 749 S2 é o principal responsável pela especificidade da enzima cruzaína, sendo pouco exposto ao solvente e é delimitado pelas cadeias laterais dos resíduos hidrofóbicos, conferindo assim a este subsítio especificidades por grupos hidrofóbicos (SOUZA, 2012). Assim, pode-se concluir que estrutura selecionada na docagem realizou interação com um resíduo importante, pertencente ao sítio ativo da enzima cruzaína, e se manteve estável durante a dinâmica molecular, mostrando que a (-)-anonaina pode possuir atividade contra a enzima, ou seja, essa interação com o resíduo encontrado nos cálculos de dinâmica permitirá o planejamento de novos inibidores para o alvo biológico em estudo. 5. CONCLUSÃO A partir dos cálculos de docagem molecular realizadas através dos programas AutoDock Vina 1.1.0 e Dock 6.3, e através da analise consensual baseado na avaliação das energias foram selecionados 20 estruturas. Posteriormente, foram avaliadas as interações com resíduos chave do sítio ativo e a toxidez, e desta forma, foram selecionados duas estruturas. Estas estruturas foram docadas com os programas supracitados e verificou-se que apenas o composto 314, a (-)-Anonaina, não apresentou interação com a enzima homóloga humana, a Catepsina L. Pode-se destacar que a estrutura selecionada para a dinâmica molecular já apresenta resultados na literatura com outras atividades, o que a torna uma molécula promissora para futuros estudos contra a cruzaína do T. cruzi. A simulação de dinâmica molecular acoplada ao método hibrido QM/MM mostraram a estabilidade do composto selecionado no sítio ativo da enzima e a partir dos resultados da contribuição individual de resíduos para a energia de interação, juntamente com as interações observadas nas conformações preditas pela docagem molecular, foi verificada interação com o resíduo Cys25 que faz parte da tríade catalítica da enzima sendo importante para a estabilização. Com isso a estrutura selecionada, a (-)-anonaina, mostra-se promissora para o tratamento da doença de Chagas. 6. REFERÊNCIAS 1. BOURGUIGNON S. C., CAVALCANTI, D. F. B., DE SOUZA A. M. T., CASTRO, H. C.,RODRIGUES C. R., ALBUQUERQUE, M. G.; SANTOS, D. O., DA SILVA, G. G, DA SILVA, F. C., FERREIRA, V. F., DE PINHO, R. T.; ALVES, C. R. Trypanosoma cruzi: Insights into naphthoquinone effects on growth and proteinase activity. Experimental Parasitology, v. 127, n. 1, p. 160-166, Jan 2011. 2. COURA, J. R., VIÑAS, P. A. Chagas disease: a new worldwide challenge. Nature. 2010 Jun; 465: S6-S7. 3. COURA, J. R., CASTRO, S. L. A Critical review on Chagas disease chemotherapy. Memorias do Instituto Oswaldo Cruz. V. 97, p. 3-24, 2002 4. COURA, J. R., VIÑAS, P. A. Chagas disease: a new worldwide challenge. Nature. 2010 Jun; 465: S6-S7. 5. DIAS, L. C. NEGLECTED TROPICAL DISEASES: A NEW ERA OF CHALLENGES AND OPPORTUNITIES. Quimica Nova, v. 36, n. 10, p. 1552-1556, 2009. 6. DIAS, L. C., DESSOY, M. A. Quimioterapia da doença de Chagas: Estado da arte e perspectivas no desenvolvimento de novos fármacos. Química Nova, v. 32, n. 9, p. 2444-2457, 2013. 7. DNDi América Latina. Disponível em: http://www.dndial.org/pt/doencas-negligenciadas/doencade-chagas.html/. Acessado em: 01 de Março de 2014. 8. DORN, P. L., PERNICIARO, L., YABSLEY, M. J., ROELLING, D. M., BALSAMO, G., DIAZ, J., WESSON, D. Autochthonous transmission of Trypanosoma cruzi, Louisiana. Emerg Infect Dis. 2007; 13:605-607 9. ESPERANDIM, V. R., FERREIRA, D. S., REZ ENDE, K. C. S., CUNHA, W. R., SARAIVA, J., BASTOS, J. K., SILVA, M. L. A., ALBURQUERQUE, S. In vivo infection by Trypanosoma cruzi: a 750 morphometric study of tissue changes in mice. Parasitology Research, v. 112, n. 1, p. 431-436, Jan 2013. 10. GIAROLLA, J., FERREIRA, E. I. Planejamento de fármacos na área de doença de Chagas: Avanços e Desafios. Revista Virtual de Química. p. 68-88, 2013. 11. LIPINSKI, C. A., LOMBARDO, F., DOMINY, W., FEENEY, P.J. Experimental and computational approaches to estimate solubility and permeability in drug discovery and development settings. Advanced Drug Delivery Reviews, v. 23, n. 1-3, p. 3-25, Jan 1997. 12. HSING-TAN, LI., WU, H., CHEN, H., LIU, C., CHEN. C. The Pharmacological Activities of ()-Anonaine. Molecules, v 18, p. 8257-8263, 2013. 13. MOREIRA, D. R. M., LEITE, A. C. L., CARDOSO, M. V. O., SRIVASTAVA, R. M., HERNANDES, M. Z., RABELLO, M. M., CRUZ, L. F., FERREIRA, R. S., SIMONE, C. A., MEIRA, C. S., GUIMARÃES, E. T., SILVA, A. C., SANTOS, T. A. R., PEREIRA, V. R. A., SOARES, M. B. P. Structural Design, Synthesis and Structure-Activity Relationships of Thiazolidinones with Enhanced Anti-Trypanosoma cruzi Activity. ChemMedChem, v. 9, p. 177-188, 2014. 14. LOZANO, N. B. H., WEBER, K. C., HONORIO, K. M., GUIDO, R. V. C., ANDRICOPULO, A. D., DA SILVA, A. B. F. Interactive Multimedia to Teach the Life Cycle of Trypanosoma cruzi, the Causative Agent of Chagas Disease. Plos Neglected Tropical Diseases, v. 6, n. 8, Aug 2012. 19. PATIL, A. D. Antidyslipidemic drugs in metabolic syndrome. Indian J Endocrinol Metab. Vol. 17, n. 6 p. 1133. 2013. 15. PAZ-Y-MIÑO, C., CUMBAL, N., SÁNCHEZ, M. E. Genotoxicity Studies Performed in the Ecuadorian Population. Molecular Biology International, v. 2012, 2012. 16. SARTORI, G. R. Planejamento de inibidores baseado em fragmentos moleculares para a enzima gliceraldeido-3-fosfato desidrogenasse de Trypanosoma cruzi. 160f. Tese (pós-graduação)-Instituto de Química de São Carlos, USP, São Carlos, 2012. 17. SOUZA, M. L. Identificação de novos inibidores da enzima cruzaína de Trypanosoma cruzi candidatos a fármacos contra a doença de Chagas. Dissertação de Mestrado. Universidade de São Paulo. São Carlos –SP, 2012. 18. SUSHKO I., SALMINA, E., POTEMKIN V. A., PODA, G., TETKO I. V. ToxAlerts: A Web Server of Structural Alerts for Toxic Chemicals and Compounds with Potential Adverse Reactions. J Chem Inf Model, v.52, n.8, p.2310-6, 2012. 19. TEIXEIRA, D. E., BENCHIMOL, M., CREPALDI, P. H., SOUZA, W. Interactive Multimedia to Teach the Life Cycle of Trypanosoma cruzi, the Causative Agent of Chagas Disease. Plos Neglected Tropical Diseases, v. 6, n. 8, Aug 2012. 20. URBINA, J. A., DOCAMPO, R. Specific chemotherapy of Chagas disease: controversies and advances. Trends Parasitol. V. 19, p. 495-501, 2003. 21. VALDÉZ, F. J. S.; BRANDÁNA, C. P.; ZAGO, M. P.; LABRIOLA, C., FERREIRA, A.;BASOMBRÍO, M. Á. Trypanosoma cruzi carrying a monoallelic deletion of the calreticulin (TcCRT) gene are susceptible to complement mediated killing and defective in their metacyclogenesis. Molecular Immunology, v. 53, p.198 – 205, 2012. 22. WIGGERS, H. J., ROCHA, J. R.,CHELESKI, J., MONTANARI, C. A. Integration of Ligandand Target-Based Virtual Screening for the Discovery of Cruzain Inhibitors. Molecular Informatics, v. 30, n. 6-7, p. 565-578, Jun 2011. 23. WIGGERS, H. J., ROCHA, J. R., FERNANDES, W. B., SESTI-COSTA, R., CARNEIRO, Z. A., CHELESKI, J., SILVA, A. B. F., JULIANO, L., CEZARI, M. H.S., SILVA, J. S., MCKERROW, J. H., MONTANARI, C. A. Non-peptidic Cruzain Inhibitors with Trypanocidal Activity Discovered by Virtual Screening and In Vitro Assay. PLOS Neglected Tropical Diseases, v. 7, e2370, 2013 24. WORLD HEALTH ORGANIZATION – WHO. Disponível em: http://www.who.int/neglected_diseases/diseases/chagas/en/. Acessado em: 07 de junho de 2015 751
Download