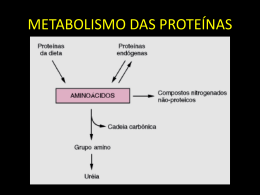
Síntese Protéica - Tradução Biologia Molecular Profª. Marília Scopel Andrighetti Estrutura dos tRNAs tRNA – transfere a informação contida no genoma para uma sequência de aminoácidos; Deve existir, pelo menos, um tRNA para cada aminoácido presente na célula; Os tRNAs de eucariotos e procariotos apresentam uma estrutura secundária de folha de trevo, mantida por sequências complementares na própria molécula; Em sua estrutura, apresentam 4 braços principais, contendo regiões pareadas (hastes) e regiões de fita simples (alças), e um braço variável. Estrutura dos tRNAs Braço aceptor: região conservada – CCA. A ligação com o aminoácido ocorre com o OH da ribose da adenina; Braço TC: Apresenta na alça uma base não usual, denominada pseudouridina; Braço D: Presença de bases modificadas; Braço do anticódon: contém o triplet do anticódon no centro da sequência, formando a alça; Braço variável: pode ser pequeno (75% das células) ou longo. Aminoacil-tRNA Os tRNAs são denominados aminoácido que representam; de acordo com o Quando estão ligados ao aminoácido, são chamados de aminoacil-tRNA; Um tRNA pode ligar-se a somente um aminoácido, ligando-se covalentemente a ele; O tRNA contém um aminoácido complementar ao códon do mRNA que representa o aminoácido. Aminoacil-tRNA funcional O aminoacil-tRNA funcional apresenta uma estrutura em forma de L; A haste da região aceptora e a da TC forma uma dupla hélice, enquanto que a haste do anticódon e do braço D formam uma segunda dupla hélice; O aminoácido e o anticódon são posicionados em ambas extremidades do L; A estrutura é mantida por pontes de hidrogênio entre diferentes regiões da molécula. Aminoacil-tRNA sintetase Enzima que sintetiza a ligação do tRNA com o aminoácido (existem, pelo menos, 20 sintetases); Ativação do aminoácido: reage com ATP e libera pirofosfato (PPi); O aminoácido ativado é transferido, então, para o tRNA, formando a aminoacil-tRNA; A enzima aminoacil-tRNA sintetase tem um mecanismo de correção de erro, para evitar a incorporação de um aminoácido incorreto no tRNA. Estrutura dos Ribossomos Estrutura compacta de ribonucleoproteínas com 2 subunidades. Cada subunidade é formada por proteínas associadas a moléculas de rRNAs; Ribossomos são organelas assimétricas, compostas por uma região de base e outra contendo uma cabeça, ou protuberância. Subunidades Ribossomais Procariotos: Subunidade 50S: maior Subunidade 30S: menor Eucariotos: Subunidade 60S: maior Subunidade 40S: menor 70S 80S Sítios Ativos dos Ribossomos Sítio A: subunidades 50S ou 60S; liga-se com o aminoacil-tRNA; Sítio P: subunidades 30S ou 40S; liga-se peptidil-tRNA; Sítio E: sítio sintetizadas. de saída das novas ao proteínas Pareamento entre rRNA e Ribossomo Para o início da síntese proteica precisa ocorrer a ligação entre o mRNA e o ribossomo: Procarioto Sítio de ligação do ribossomo (RBS): • Sequência de mRNA que é recoberta pelo ribossomo; • O códon de iniciação AUG está contida na sequência; • mRNA possui uma sequência parcialmente complementar à uma região do rRNA, denominada Shine-Delgarno. É uma sequência localizada a 7 nucleotídeos do códon AUG, em direção à extremidade 5’. Pareamento entre rRNA e Ribossomo Eucarioto Reconhecimento do Cap 5’: • A subunidade 40S do ribossomo reconhece o Cap 5’ e desloca-se pelo mRNA até encontrar o códon de iniciação AUG. Códon de iniciação e tRNA iniciador Em procariotos, o início da síntese proteica é definida pela RBS e pelo códon iniciador AUG (metionina); Nesse caso, o tRNA iniciador está ligado a uma metionina que contém um grupo formil ligado ao radical amino, formando tRNAfMet; Esse tRNA é utilizado somente como iniciador; Já o tRNAMet reconhece metioninas internas, nunca apresentando uma metionina formilada. tRNA iniciador Somente o tRNAfMet liga-se ao sítio P do ribossomo, o qual é formado pelo mRNA e a subunidade 30S (procariotos); A ligação do tRNAfMet adicionado ao mRNA e à subunidade 30S forma o complexo de iniciação da tradução. Diferença entre o início da síntese proteica em Eu e Procariotos Em bactérias, o complexo de iniciação forma-se em um local específico (RBS), próximo ao códon AUG; Em eucariotos, o Cap 5’ é reconhecido e a subunidade 40S desliza até o AUG. Tradução Durante a síntese de proteínas, os ribossomos deslocamse ao longo do mRNA, possibilitando um pareamento entre esse e os tRNAs que carregam os diferentes aminoácidos que irão compor as proteínas; Os ribossomos deslocam-se ao longo do mRNA, na direção 5’3’, sintetizando a proteína no sentido amino para carboxi-terminal; Um mesmo mRNA é traduzido por diferentes ribossomos Tradução O ribossomo se desloca expondo novos triplets nos sítios A e P; O sítio A expõe o códon correspondente ao aminoácido novo que será incorporado, sendo ocupado pelo aminoaciltRNA complementar a ele; O sítio P é ocupado pelo tRNA correspondente ao códon anterior, o qual também carregará a cadeia polipeptídica em formação (peptidil-tRNA). Início Complexo de iniciação: ribossomo, mRNA e aminoaciltRNA inicial 1º - liga-se a subunidade menor 30S no sítio RBS do mRNA, seguido pelo aminoacil-tRNA inicial, formando o complexo de iniciação; 2º - adição de 50S formando o ribossomo completo. No sítio P está o tRNAfMet e no A o códon do aminoácido seguinte Em eucariotos, a subunidade 40S reconhece o cap 5’ e desloca-se até o primeiro AUG. Após, liga-se o aminoaciltRNA inicial no sítio P, formando o complexo de iniciação. Por fim, a subunidade 60S une-se ao complexo, formando o ribossomo completo. Alongamento Os aminoácidos são adicionados isoladamente, devendo ocorrer o processo de forma cíclica; Dessa forma, sempre o aminoacil-tRNA a ser incorporado estará no sítio A e o peptídeo localizado no sítio P; O peptídeo ligado no sítio P é transferido para o aminoaciltRNA do sítio A pela peptidil-transferase, localizada na subunidade maior do ribossomo (50S ou 60S); O tRNA fica sem aminoácido ligado no sítio P e o peptidiltRNA no sítio A. Translocação O ribossomo avança 3 nucleotídeos no mRNA: O tRNA não carregado é liberado do sítio P; O peptidil-tRNA move-se do sítio A para o sítio P; Um novo códon é exposto no sítio A, o qual está preparado para receber o aminoacil-tRNA correspondente; O alongamento será sempre seguido pela translocação e assim por diante; Ambos eventos não acontecem simultaneamente. Terminação Aparece no sítio A um dos códons de terminação – UAG, UGA, ou UAA; Tais códons não são reconhecidos por tRNAs, mas sim por proteínas; A peptidil-transferase libera o peptídeo do tRNA; O tRNA é liberado do ribossomo; O peptídeo é liberado através do sítio E; As subunidades dos ribossomos são dissociadas. aa livre Ribossomo Phe Gly His Glu Proteína Asp Met Ala Cys 5’ tRNA 3’ AU G G CAU G C GAC GAAU U C G GACACAUA Molécula de mRNA Direção do avanço do ribossomo códon Phe Gly His Glu Asp Met Ala Cys 5’ AU G G CAU G C GAC GAAU U C G GACACAUA 3’ Phe Gly His Glu Met Ala Cys Asp 5’ AU G G CAU G C GAC GAAU U C G GACACAUA 3’ His Gly Met Ala Cys Asp Phe Glu 5’ AU G G CAU G C GAC GAAU U C G GACACAUA 3’ Ile Met Ala Cys Asp Glu His Gly Phe 5’ AU G G CAU G C GAC GAAU U C G GACACAUA 3’ Lys Met Ala Cys Asp Glu Phe Ile His Gly 5’ AU G G CAU G C GAC GAAU U C G GACACAUA 3’ Met Ala Cys Asp Glu Phe Gly Lys Ile His 5’ AU G G CAU G C GAC GAAU U C G GACACAUA 3’ Met Ala Cys Asp Glu Phe Gly His Lys Ile 5’ AU G G CAU G C GAC GAAU U C G GACACAUA 3’ Met Ala Cys Asp Glu Phe Gly His Ile Leu Lys 5’ G CAU G C GAC GAAU U C G GACACAUAAAA 3’ Met Ala Cys Asp Glu Phe Gly His Ile Lys Met Leu 5’ U G C GAC GAAU U C G GACACAUAAAAU UA 3’ Met Ala Cys Asp Glu Phe Gly His Ile Lys Leu Asn Met 5’ GAC GAAU U C G GACACAUAAAAU UAAU G 3’ Met Ala Cys Asp Glu Phe Gly His Ile Lys Leu Met Pro Asn 5’ GAAU U C G GACACAUAAAAU UAAU GAAC 3’ Met Ala Cys Asp Glu Phe Gly His Ile Lys Leu Met Asn Gln Pro 5’ U U C G GACACAUAAAAU UAAU GAAC C CA 3’ Met Ala Cys Asp Glu Phe Gly His Ile Lys Leu Met Asn Pro Gln 5’ G GACACAUAAAAU UAAU GAAC C CACAA 3’ Met Ala Cys Asp Glu Phe Gly His Ile Lys Leu Met Asn Pro Gln 5’ STOP 3’ CACAUAAAAU UAAU GAAC C CACAAUAA Ala Cys Asp Glu Phe Met Gly His Ile Lys Leu Met Asn Pro Gln 5’ STOP AUAAAAU UAAU GAAC C CACAAUAAAAA 3’ Ala Cys Asp Glu Phe Met Gly His Ile Lys Leu Met Asn Pro Gln 5’ STOP AUAAAAU UAAU GAAC C CACAAUAATAC 3’ Ala Cys Asp Glu Phe Met Gly His Ile Gln Lys Pro Leu Asn Met 5’ 3’ AUAAAAU UAAU GAAC C CACAAUAATAC VARIAÇÃO GENÉTICA = MUTAÇÃO E POLIMORFISMO Ala Cys Asp Glu Phe Met Gly His Ile Gln Lys Pro Leu Asn Met 5’ 3’ AUAAAAU UAAU GAAC C CACAAUAATAC Ala Cys Asp Glu Phe Met Gly His Ile Gln Lys Pro Leu Asn CYS 5’ Muda a forma e função 3’ A U A A A A U U A A U G A A C AA A C A A U A A T A C O Dogma Central da Biologia Molecular Define o paradigma da biologia molecular, em que a informação é perpetuada através da replicação do DNA e é traduzida através de dois processos: 1.Transcrição, que converte a informação do DNA em uma forma mais acessível (uma fita de RNA complementar); 2.Tradução, proteínas. que converte a informação contida no RNA em Núcleo RNA polimerase Gene Transcrição hnRNA Processamento mRNA Tradução Citoplasma proteína
Download