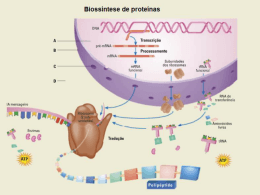
Antibióticos e Síntese Proteica Ana Sales Ana Nunes Petra Gouveia Rita Gonçalves O que são antibióticos “Substância produzida por um organismo ou obtida sinteticamente que, em soluções diluídas, destrói as bactérias e outros microrganismos ou inibe o seu desenvolvimento.” [1] Um antibiótico “...apresenta boa capacidade antimicrobiana em baixa concentração (para ser viável) além de apresentar uma boa toxidade selectiva (sendo tóxico para bactérias e não para as células do corpo)...” [2] A história dos antibióticos [3] Em 1889, Vuillemin propõe o termo “antibiose” – antagonismo dos seres vivos em geral. O termo “antibiótico” surge em 1942 por Waksman. A história dos antibióticos pode dividir-se em 3 eras: Era dos alcalóides Era dos compostos sintéticos Era moderna dos antibióticos Era dos alcalóides Tem início em 1619 com o sucesso no tratamento da malária e disenteria amebiana. Em 1860 Joseph Lister estudou a inibição provocada por produtos químicos sobre as bactérias. Lister usou fenol para esterilizar instrumentos com que praticava cirurgia (marcando o início da era antimicrobiana). Era dos compostos sintéticos Em 1909 descoberta do salvarsan por Paul Ehrlich – tratamento de tripanossomas e outros protozoários. Klarer e Meitzsch sintetizaram o prontosil (sulfonamida) em 1932. Os efeitos e resultados organizados por Gerhard Domagk garantiramlhe o Prémio Nobel da Medicina em 1938. Em 1929 Alexander Fleming sintetiza a penicilina. O seu efeito curativo demonstrado em ratos cedo possibilitou o estudo em pacientes com erisipela e outras infecções. Verificou-se que o prontosil não tinha actividade antibacteriana in vitro, daí que muitos químicos tentaram melhorar a molécula, dando origem, em 1938, à sulfapiridina, a primeira droga efectiva no tratamento das pneumonias pneumocócicas. Surgiram depois a sulfatiazolina e a sulfadiazina – melhoram cianose e vómitos provocados pelas sulfas antigas. Era moderna dos antibióticos Controlo das infecções por estreptococos e pneumococos com utilização clínica das sulfonamidas. No final dos anos 40, verificou-se resistência ao antibiótico. A resistência bacteriana leva à busca de novas substâncias. O uso clínico da penicilina deu origem à mais variada e utilizada classe de antibióticos: blactâmicos. Em 1944 Waksman e Schatz isolaram a estreptomicina, primeira droga efectiva contra a tuberculose. Actualmente sabe-se que os agentes terapêuticos de sucesso tinham propriedades em comum: Actividade microbiana letal ou inibitória em altas diluições Não influenciar a função do órgão, tecido Não causar danos Ser estáveis, solúveis, com baixa excreção e óptima difusão Mesmo após séculos de estudos, a resistência bacteriana continua a ser o principal desafio Classificação dos antibióticos [2] Com base na origem: Naturais (penicilina G) Semi-sintéticos (aminopenicilinas) Sintéticos (cloranfenicol) Com base na acção biológica Bactericidas: destrói as bactérias Bacteriostáticos: paralisam o crescimento das bactérias Quanto ao espectro de acção Pequeno espectro: atinge um grupo de bactérias Grande especto: atinge mais do que um grupo de bactérias Com base no mecanismos de acção Parede bacteriana: inibição da síntese da parede Membrana celular: alteração da permeabilidade selectiva Ribossomas: alteração na síntese ou interrupção da síntese proteica DNA e RNA: inibição da síntese do material genético Metabolitos essenciais: inibição dos mesmos Forma correcta de utilização Tomar antibiótico apenas sob indicação médica. Não tomar nem ceder a terceiros o antibiótico que possa ter em casa, visto que pode não ser o indicado para a infecção. Tomá-lo sempre à mesma hora, todos os dias e até ao fim do tratamento. Se após 3 dias não se verificarem melhorias contactar o médico. Antibióticos e síntese proteica Antibiótico Células-alvo Estreptomicina Procariótica Tetraciclina Procariótica Cloranfenicol Procariótica Eritromicina Procariótica Puromicina Procariótica e Eucariótica Cicloheximida Eucariótica Efeito - Inibe a iniciação - Provoca erro na leitura do RNAm - Inibe a ligação do aminoacilRNAt ao sítio A do ribossoma - Inibe a actividade da peptidil transferase - Liga-se à subunidade 50S do ribossoma e inibe a translocação - Provoca a terminação prematura da cadeia, actuando como um análogo do aminoacilRNAt - Inibe a actividade da peptidil transferase Síntese Proteica A sequência de nucleotídeos no DNA é transcrita em RNAm e a sequência de nucleotídeos do RNAm é traduzida numa sequência de aminoácidos que formam a proteína. Transcrição Transcrição Consiste na síntese de RNA a partir de uma cadeia de DNA. Esta síntese faz-se na presença da enzima RNA polimerase e na direcção 5’ 3’. A sequência de DNA na qual a RNA polimerase se liga para iniciar a transcrição é designada promotor. Após ligação ao promotor, a polimerase abre uma região do DNA, expondo os nucleotídeos de uma pequena zona de ambas as cadeias. Uma das duas sequências expostas funciona como molde para o emparelhamento de bases complementares. À medida que se move, a polimerase vai desenrolando o DNA molde à sua frente, e enrola-o novamente após a sua passagem. A síntese de RNA prossegue até que a polimerase encontre um sinal de terminação. Neste ponto a transcrição pára, o RNA é libertado da polimerase e a enzima dissocia-se do molde de DNA. Splicing do RNAm O produto primário da transcrição sofre ainda uma série de transformações no núcleo – maturação do RNAm. É necessário este processamento do RNAm de modo a que as sequências não codificávies, intrões, sejam retiradas e ocorra posterior união das sequências codificávies, exões, formando-se assim um RNAm activo ou funcional. Numa primeira etapa, o pré-RNAm é clivado na extremidade 5’ do intrão, que é então unida a um nucleotídeo de adenina dentro do intrão (perto da sua extremidade 3’). O intermediário resultante tem uma estrutura em forma de laço. Depois, ocorre clivagem na extremidade 3’ do intrão e a ligação entre os dois exões. Após splicing, as moléculas de RNAm são exportadas para o citoplasma, onde irão codificar proteínas. Tradução Consiste na transformação da mensagem contida no RNAm, via RNAt, na sequência de aminoácidos que constituem a proteína. Intervenientes: RNAm RNAt Ribossomas (RNAr) Aminoácidos Sistemas enzimáticos RNAt Apresenta uma estrutura tridimensional em folha de trevo. É o intermediário entre os a.a. e o RNAm. Selecciona e transporta o a.a. apropriado e reconhece o codão correspondente do RNAm. Tem a sequência CCA no seu terminal 3’, onde os a.a. são covalentemente ligados à ribose da adenosina terminal. Na outra extremidade, possui uma região constituída por 3 nucleotídeos chamada anticodão, que é complementar de um codão do RNAm. Activação dos aminoácidos síntese de aminoacil-RNAt A ligação dos a.a. a RNAt específicos é catalisada pela sintetase de aminoacil-RNAt. Numa primeira reacção, o a.a. é activado pela reacção com ATP. O a.a., então activado, é unido ao terminal 3’ do RNAt, formando o aminoacil-RNAt. Ribossomas Constituem a maquinaria molecular responsável pela tradução do RNAm a proteínas. Complexos constituídos por mais de 50 tipos de proteínas associadas a moléculas de RNAr (que tem um papel central na actividade catalítica do ribossoma). Compostos por 2 subunidades: A pequena subunidade estabelece a correspondência entre o RNAt e os codões do RNAm. A grande subunidade catalisa a formação das cadeias polipeptídicas. Cada ribossoma possui 3 sítios de ligação ao RNAt: sítios A (aminoacil-RNAt), P (peptidil-RNAt) e E (saída), e um sítio de ligação ao RNAm. Código genético Corresponde à relação entre a sequência de bases no DNA e a sequência de a.a. numa proteína. Uma sequência de 3 nucleotídeos (tripleto) especifica um a.a.. O código genético é basicamente o mesmo em todos os organismos. O código genético é degenerado (os a.a. são especificados por mais que um codão) e não é ambíguo (cada codão só especifica um a.a.). Etapas da tradução A síntese proteica ocorre em 3 etapas sucessivas: 1. Iniciação 2. Alongamento 3. Finalização 1. Iniciação 1 Estreptomicina 2 3 4 1. Iniciação A tradução inicia-se com um codão de iniciação AUG que corresponde a um RNAt iniciador que transporta sempre a metionina. Este RNAt iniciador liga-se à pequena subunidade ribossomal. Há também a ligação de factores de iniciação. A pequena subnidade ribossomal liga-se ao terminal 5’ do RNAm e percorre-o até encontrar o primeiro AUG. A grande subunidade ribossómica liga-se à pequena subunidade, formando um ribossoma funcional. O RNAt iniciador encontra-se no local P deixando o local A vazio, pronto para que outra molécula de aminoacilRNAt o ocupe, iniciando a síntese proteica. 2. Alongamento Puromicina Eritromicina Tetraciclina 2. Alongamento Após o complexo de iniciação ter sido formado, a tradução continua pelo alongamento da cadeia polipeptídica. O sítio A, até então vazio, é ocupado por um aminoacil-RNAt correspondente ao segundo codão do RNAm. A metionina solta-se do RNAt iniciador e liga-se por ligação peptídica ao a.a. recém-chegado no local A, formando um peptidilRNAt. De seguida, ocorre a translocação, em que o ribossoma se move 3 nucleotídeos ao longo do RNAm, posicionando o próximo codão num sítio A vazio. Assim, o peptidil-RNAt é translocado do sítio A para o P e o RNAt iniciador do sítio P para o E. A ligação de um novo aminoacil-RNAt ao sítio A, induz a libertação do RNAt iniciador do sítio E, deixando o ribossoma pronto para a inserção do próximo a.a. na cadeia polipeptídica em formação. O alongamento da cadeia polipeptídica prossegue até que um codão de STOP seja translocado no sítio A do ribossoma. Formação de ligações peptídicas O terminal COOH da cadeia polipeptídica ligada ao RNAt no sítio P é desligado (pelo rompimento da ligação) e ligado ao grupo amina livre do a.a. ligado ao RNAt no sítio A. Esta reacção é catalisada pela enzima peptidil transferase. Cloranfenicol Cicloheximida 3. Finalização 3. Finalização Após vários ciclos de alongamento surge um codão STOP (UAA, UAG, UGA) no local A. Estes codões não são reconhecidos por nenhum RNAt. Liga-se um factor de terminação ao codão STOP. Esta ligação altera a actividade da peptidil transferase, que catalisa a adição de H2O (em vez de um a.a.) ao peptidil-RNAt. Dá-se a hidrólise da ligação entre o peptídeo e o RNAt, com consequente libertação do peptídeo e do RNAt do ribossoma. O ribossoma liberta o RNAm e dissocia-se nas suas 2 subunidades. Síntese proteica: eucariontes vs procariontes Síntese proteica: eucariontes vs procariontes Etapa Eucariontes Procariontes Transcrição Têm 3 RNA polimerases que sintetizam diferentes RNAs: •Polimerase I: sintetiza RNAr de grandes dimensões. •Polimerase II: sintetiza o RNAnh (que origina o RNAm) e o RNAsn. •Polimerase III: sintetiza RNAr de pequena dimensão e RNAt. As RNA polimerases requerem factores de transcrição para se ligarem às sequências promotoras. Os genes são transcritos por uma única RNA polimerase. A RNA polimerase liga-se directamente às sequências promotoras. Processamento do RNAm Os transcritos primários de RNAm sofrem processamento por splicing, antes de serem usados como moldes para a síntese proteica. Os ribossomas têm acesso imediato ao RNAm e a tradução é iniciada enquanto a transcrição ainda está em progresso. Iniciação A síntese proteica é iniciada com metioninas não-modificadas. Factores de iniciação: eIF-1, eIF-1A, eIF-2, eIF-2B, eIF-3, eIF-4A, eIF-4B, eIF-4E, eIF-4G, eIF-5. A síntese proteica é iniciada com um resíduo de metionina modificada: N-formilmetionina. Factores de iniciação: IF-1, IF-2, IF-3. Alongamento Factores de alongamento: eEF-1α, eEF-1βδ, eEF-2. Factores de alongamento: EF-Ta, EF-Ts, EF-G. Factores de terminação: eRF-1, eRF-3. Factores de terminação: RF-1, RF-2, RF-3. Tradução Finalização Agradecimentos Mestre Clara Monteiro Instituto de Biologia Celular e Molecular Referências bibliográficas [1]Dicionário Enciclopédico da Língua Portuguesa [2]http://farmacologia.kit.net/antibioticos.htm [3]http://www.medstudents.com.br/historia/fleming/ fleming.htm Cooper,Geoffrey M.; A célula: Uma abordagem molecular; trad. Itabajara da Silva Vaz Junior et al; 2ª edição, Porto Alegre; Armed Editora; 2002 Alberts, Bruce; Johnson, Alexander; Lewis, Julian; Raff, Martin; Roberts, Keith; Walter Peter; Molecular Biology of the Cell; 4th ed.; Garland Science; 2002
Baixar