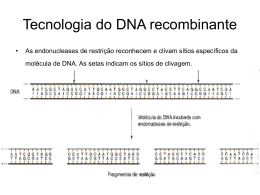
1 2 3 Entendem-se por compostos heterocíclicos, aqueles compostos orgânicos cíclicos estáveis, que contem no seu anel um ou mais átomos diferentes do carbono. 4 5 O prefixo “ribo” também é aceitável para os ribonucleosídeos e ribonucleotídeos; porém a nomenclatura mais curta é a mais usada. Timina é uma exceção: o nome ribotimina é usado para descrever sua ocorrência não usual no RNA. 6 7 8 9 É necessário que a hidroxila da posição 3’ de um nucleotídeo esteja livre para que a DNA polimerase possa catalisar o ataque nucleofílico ao fosfato (alfa) ligado ao carbono 5’ do desoxinucleosídeo 5’ trifosfato seguinte, promovendo liberação de pirofosfato inorgânico e de energia suficiente para que ocorra a ligação fosfodiéster. 10 11 Por definição, a extremidade 5’ não possui nucleotídeo ligado na posição 5’, e a extremidade 3’ não o possui na posição 3’. Outros grupos (na maioria das vezes um ou mais fosfatos) podem estar presentes em uma ou em ambas as extremidades. 12 13 Por definição, a extremidade 5’ não possui nucleotídeo ligado na posição 5’, e a extremidade 3’ não o possui na posição 3’. Outros grupos (na maioria das vezes um ou mais fosfatos) podem estar presentes em uma ou em ambas as extremidades. 14 15 16 Se a molécula tem 35% de adenina, também terá 35% de timina, totalizando 70%. Assim, sobram 30% para guanina e citosina: 15% de cada. 17 18 19 20 21 22 O rRNA é componente estrutural dos ribossomos, os quais servem de sítio para a tradução da sequencia de mRNA em uma sequência específica de aminoácidos ou cadeia polipeptídica. 23 24 25 Não precisa estudar. É só para conhecerem. 26 A ribonuclease P (RNAse P) é uma endonuclease que processa tRNA de E.coli e pode ser dissociada em dois componentes: um RNA de 375 bases e um polipeptídeo de 20 kD. Sua atividade catalítica reside no RNA, mas o componente protéico é responsável por um grande aumento na velocidade da reação. Os pequenos RNAs de vegetais são moléculas de RNA infecciosas que funcionam como endonucleases em reações de autoclivagem. Os viróides funcionam independentemente, sem necessidade de um capsídeo protéico, enquanto os virusóides (chamados RNAs satélites) são incluídos em capsídeos protéicos juntamente com o genoma viral. 27 RNA fita dupla: ex.: reovírus. DNA fita simples: ex.: fagos, FX174, S13, M13, parvovírus. 28 Sim, pois a hidrólise de moléculas de DNA e RNA libera moléculas de nucleotídeos que se diferenciam pelo tipo de pentose: ribose no RNA e desoxirribose no DNA, ou pelas bases Timina (T) - DNA e Uracila (U) - RNA. 29 Sim, a molécula A é de DNA, pois observamos que nesta as quantidades de guanina e citosina são iguais. 30 31 Experimento de Meselson e Stahl: células de E. coli foram cultivadas em meio nutritivo contendo somente N15 (nitrogênio pesado) durante várias gerações (a). Essas células foram transferidas para outro meio nutritivo contendo apenas N14 (nitrogênio leve): após um único ciclo de replicação (b – moléculas híbridas N15/N14 – padrão de sedimentação intermediário) e após um segundo ciclo de replicação (c – padrão de sedimentação: ½ híbrida e ½ N14 apenas). 32 Enzimas especializadas chamadas iniciases sintetizam os iniciadores quando e onde eles forem requeridos. 33 34 35 A topoisomerase ou DNA topoisomerase é uma enzima que desempenha importante papel nos processo de replicação e empacotamento de DNA. Seria uma nuclease reversível. Ela catalisa uma quebra nas moléculas de DNA, mas usa ligações covalentes para segurar as moléculas de DNA que foram quebradas. Existem dois tipos de topoisomerases: 1) Topoisomerase I: Produz quebras em uma fita do DNA e permite o giro da fita quebrada sobre a fita intacta. A topoisomerase I conserva a energia do rompimento da ligação fosfodiéster, estocando-a na forma de ligação covalente que ocorre entre ela e os grupamentos fosfatos, no ponto de clivagem. Depois ela utiliza essa energia para restaurar a ligação fosfodiéster e selar a quebra. Algumas topoisomerases I podem relaxar superespiralamentos positivos e negativos no DNA. 2) Topoisomerase II Produz quebras nas duas fitas do DNA. Ela quebra as duas fitas de DNA ao mesmo tempo e pode introduzir ou retirar superespiras, duas de cada vez, em um mecanismo que é dependente de ATP. Ela corta as duas fitas de DNA, prendem-se às extremidades através de ligações covalentes, passa a dupla fita através do corte e sela a quebra. 36 37 Fita descontínua: vários iniciadores consecutivos, cada um responsável pela polimerização de um fragmento de DNA. Em bactérias, os fragmentos de Okazaki possuem comprimento de aproximadamente 1.000 a 2.000 nucleotídeos. Nas células eucarióticas, eles possuem de 150 a 200 nucleotídeos de comprimento. 38 39 40 41 42 Os telômeros são complexos DNA-proteína encontrados nas extremidades dos cromossomos lineares, que os protegem da degradação, da recombinação e da fusão, estabilizando-os. Devido à observação de que seu tamanho regride ao longo das duplicações celulares até um tamanho mínimo que interrompe a proliferação celular, criou-se a hipótese de que o telômero funcionaria como um relógio celular e seria um dos fatores responsáveis pela senescência. Na maioria dos organismos, os telômeros são formados por repetições em tandem (agrupadas) de DNA com uma sequência simples. A telomerase é um ribonucleoproteína com atividade polimerase que mantém as terminações teloméricas por adição de repetições TTAGGG do telômero. A enzima consiste de um componente proteico com atividade de transcriptase reversa e um componente RNA que serve como molde para as repetições teloméricas. 43 mtDNA= DNA mitocondrial. 44 45 46 47 48 49 50 51 52 53 54 55 Quanto à cadeia lateral (estacada em negrito), os aminoácidos podem ser classificados em: 1- Aminoácidos apolares: apresentam grupos químicos de hidrocarbonetos apolares ou hidrocarbonetos modificados, exceto a glicina (que possui um átomo de hidrogênio como cadeia lateral). São hidrofóbicos. Glicina: H-CH(NH2)-COOH; Alanina: CH3-CH(NH2)-COOH; Leucina: CH3(CH3)CH2-CH(NH2)-COOH; Valina: CH3-CH(CH3)-CH(NH2)-COOH; Isoleucina: CH3-CH2CH(CH3)-CH(NH2)-COOH; Prolina: -CH2-CH2-CH2- ligando o grupo amino ao carbono alfa; Fenilalanina: C6H5-CH2-H(NH2)-COOH; Triptofano: R aromático-CH(NH2)COOH; Metionina: CH3-S-CH2-CH2-CH(NH2)-COOH 2- Aminoácidos polares neutros: apresentam grupos químicos que tendem a formar ligações de hidrogênio. Serina: OH-CH2-CH(NH2)-COOH; Treonina: OH-CH(CH3)-CH(NH2)-COOH; Cisteina: SH-CH2-CH(NH2)-COOH; Tirosina: OH-C6H4-CH2-CH(NH2)-COOH; Asparagina: NH2-CO-CH2-CH(NH2)-COOH; Glutamina: NH2-CO-CH2-CH2CH(NH2)-COOH 3- Aminoácidos ácidos: apresentam grupos carboxilato. São hidrofílicos. Ácido aspártico: HCOO-CH2-CH(NH2)-COOH; Ácido glutâmico: HCOO-CH2-CH2CH(NH2)-COOH 4- Aminoácidos básicos: apresentam grupos amino. São hidrofílicos. Arginina: HN=C(NH2)-NH-CH2-CH2-CH2-CH(NH2)-COOH; Lisina: NH2-CH2-CH2CH2-CH2-CH(NH2)-COOH; Histidina: H-(C3H2N2)-CH2-CH(NH2)-COOH 56 Código de letras usadas na representação dos aminoácidos: A= Alanina (Ala) C= Cisteína (Cys) D= Aspartato (Asp) E= Ácido glutâmico (Glu) F= Fenilalanina (Phe) G= Glicina (Gly) H= Histidina (His) I= Isoleucina (Ile) K= Lisina (Lys) L= Leucina (Leu) M= Metionina (Met) N= Asparagina (Asn) P= Prolina (Pro) Q= Glutamina (Gln) R= Arginina (Arg) S= Serina (Ser) T= Treonina (Thr) W= Triptofano (Trp) Y= Tirosina (Tyr) V= Valina (Val) 57 Nos procariontes, o primeiro aminoácido carregado é uma forma modificada de metionina (formilmetionina). 58 A presença do códon de iniciação (AUG) é extremamente importante, pois fornece o quadro de leitura em que o mRNA será traduzido. Em eucariontes, o códon iniciador AUG (correspondente à metionina) mais próximo da extremidade cap 5' de um mRNA é geralmente o sinal de início para a síntese de proteínas. Se o ribossomo não identificar o primeiro AUG na sequência, ele poderá seguir até o segundo ou o terceiro. Isto produz proteínas diferentes a partir de um único transcrito, em geral com o mesmo quadro de leitura, mas sem os primeiros aminoácidos, e provavelmente, sem função biológica. 59 60 61 62 63 Três códons sinalizam o fim da tradução (UAA, UAG ou UGA) e são denominados de códons de parada ou terminação, nenhum tRNA reconhece esses códons. Quando um códon de parada atinge o sítio A do ribossomo, proteínas denominadas de fatores de liberação se ligam a esse códon e modificam a atividade da peptidil transferase. A peptidil transferase catalisa então a adição de uma molécula de água a cadeia peptídica ligada ao tRNA. Essa ligação libera a cadeia polipeptídica para o citoplasma e o ribossomo se dissocia e pode iniciar uma nova síntese de proteína. 64 65 66
Download