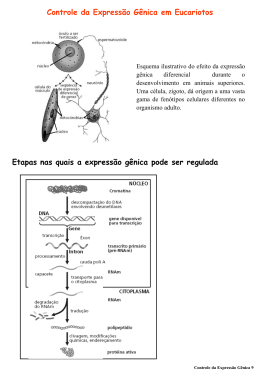
EXPRESSÃO GÊNICA O termo expressão gênica refere-se ao processo em que a informação codificada por um determinado gene é decodificada em uma proteína. Teoricamente, a regulação em qualquer uma das etapas desse processo pode levar a uma expressão gênica diferencial. Expressão gênica 1 Objetivos da regulação da expressão gênica em bactérias e em organismos multicelulares • Nas bactérias o controle da expressão gênica serve principalmente para permitir que as células se ajustem às mudanças nutricionais no ambiente, de forma que o seu crescimento e divisão sejam otimizados. • Em organismos multicelulares a expressão gênica controlada regula um programa genético fundamental para o desenvolvimento embrionário e a diferenciação. Expressão gênica 2 Como uma célula pode controlar as proteínas que ela faz? • Controlando quando e como um determinado gene é transcrito; • Controlando como um transcrito primário de RNA sofre o “splicing” ou é processado; • Selecionando quais mRNAs são traduzidos; • Ativando ou inativando seletivamente as proteínas depois da sua síntese. Expressão gênica 3 Estrutura Gênica e Definição de Termos • Gene: toda a seqüência de ácido nucléico que é necessária para a síntese de um polipeptídeo funcional ou molécula de RNA. • Unidade de Transcrição : segmento de DNA que codifica a seqüência no transcrito primário. • Promotor: a seqüência mínima necessária para que a transcrição se inicie corretamente. • Elementos Regulatórios em Cis: elementos que regulam a iniciação da transcrição. Expressão gênica 4 Expressão gênica 5 Diferenças na Iniciação da Transcrição em Eucariotos e Bactérias • • Enquanto as bactérias contêm um único tipo de RNA-polimerase, as células eucarióticas apresentam três: RNA-polimerase I, RNApolimerase II e RNA-polimerase III. A RNA-polimerase bacteriana é capaz de iniciar a transcrição sem o auxílio de proteínas adicionais. As RNA-polimerases eucarióticas requerem a ajuda de um grande conjunto de proteínas chamadas fatores gerais de transcrição. Expressão gênica 6 Diferenças na Iniciação da Transcrição em Eucariotos e Bactérias • Em eucariotos as proteínas reguladoras da expressão gênica (repressores e ativadores) podem influenciar a iniciação da transcrição, mesmo quando estão ligadas ao DNA a milhares de pares de nucleotídeos distante do promotor. Em bactérias os genes são freqüentemente controlados por uma única seqüência regulatória, tipicamente localizada próxima ao promotor. • A iniciação da transcrição em eucariotos deve levar em consideração a compactação do DNA nos nucleossomos e as formas mais compactas da estrutura da cromatina. Expressão gênica 7 As Três RNA-Polimerases das Células Eucarióticas • • • RNA-polimerase I - transcreve os genes para rRNA. RNA-polimerase II - transcreve todos os genes que codificam proteínas, mais alguns genes que codificam pequenos RNAs (p.ex., aqueles presentes nos “spliceosomes”). RNA-polimerase III – transcreve os genes de tRNAs, rRNA 5S e genes para pequenos RNAs estruturais. Expressão gênica 8 Fatores Gerais de Transcrição Os fatores gerais de transcrição são responsáveis pelo posicionamento correto da RNA-polimerase no promotor, ajudam na separação das fitas de DNA para permitir o início da transcrição, e liberam a RNApolimerase do promotor quando a transcrição se inicia. Expressão gênica 9 Formação do Complexo de Iniciação da Transcrição • • • • TFIID liga-se a região TATA, possibilitando a ligação de TFIIB. Isso é seguido pela ligação de TFIIF e RNApolimerase II. TFIIE, TFIIH e TFIIJ então se juntam ao complexo. TFIIH usa ATP para fosforilar a RNApolimerase II, mudando a sua conformação de forma que a RNA-polimerase é liberada do complexo e é capaz de iniciar a transcrição. Expressão gênica 10 Etapas na formação do complexo de iniciação da transcrição em eucariotos Estrutura tridimensional da Proteína que se liga a TATA (TATA-Binding Protein TBP) Expressão gênica 12 Complexo TBP-DNA Expressão gênica 13 Complexo TBP-TFIIA-DNA Expressão gênica 14 Complexo TBP-TFIIA-TFIIB-DNA Expressão gênica 15 Os Fatores Transcricionais Seletivos Aumentam a Atividade do Promotor • Os promotores isoladamente são geralmente ineficientes. Fatores de transcrição seletivos que se ligam à região “upstream” e a “enhancers” aumentam a iniciação. • Em alguns casos, proteínas adicionais (mediadores, coativadores) são requeridos para estimular a transcrição. • Proteínas que se ligam a seqüências de “enhancer” devem atuar de forma semehante àquelas que se ligam próximas ao promotor. O DNA entre o “enhancer” e o promotor forma uma alça para permitir que as proteínas ativadoras ligadas ao “enhancer” façam contato com as proteínas ligadas ao promotor. Expressão gênica 16 Domínios Funcionais dos Fatores Transcricionais Seletivos • • • • • Domínio de ligação ao DNA - liga a proteína no sítio de ligação do DNA. Seqüências de localização nuclear – requeridas para transporte para dentro do núcleo. Domínio de ativação transcricional - realiza o contato com os fatores gerais de transcrição. Região de dimerização – requerido para formar homo- ou heterodímeros com outras proteínas. Domínio de ligação de ligante – necessário para ligação de composto que pode funcionar como ativador do fator. Expressão gênica 17 Motivos de Ligação a DNA nos Fatores de Transcrição Homeodomínio – consiste de três -hélices adjacentes. A maior parte do contato com as bases do DNA é feita pela hélice 3. Exemplos: proteínas Hox e outras proteínas reguladoras do desenvolvimento. • Dedo de zinco (Zinc finger) - Esse motivo é constituido de uma hélice e uma folha pregueada unidas por um íon zinco. Exemplos: receptores de hormônioo esteróides, Sp1. • Região básica e zíper de leucina (ou bZip) – A região básica serve para o contato com o DNA e o zíper de leucina serve para a formação do dímero. Exemplos: Fos, Jun (complexos Fos-Jun teriam função central na mediação de resposta nuclear a sinais na superfície celular) • Hélice-alça-hélice – Contém um motivo estrutural muito semelhante a b-zip, exceto que uma alça não helicoidal separa as duas -hélices em cada monômero. Exemplo: MyoD (fator regulatório importante na determinação e diferenciação de músculo). • Expressão gênica 18 Genes Eucarióticos São Regulados por Combinação de Proteínas • • A maioria das proteínas reguladoras de genes atuam como parte de um “comitê” de proteínas reguladoras, todas essenciais para a expressão de um determinado gene na célula correta, em reposta a uma dada condição, no tempo certo e no nível requerido. O termo controle combinatorial refere-se a forma como grupos de proteínas trabalham juntas para determinar a expressão de um único gene. Expressão gênica 19 Uma Única Proteína Pode Coordenar a Expressão de Diferentes Genes Embora o controle da expressão gênica em eucariotos seja combinatorial, o efeito de uma única proteína reguladora pode ser decisiva para ligar e desligar, simplesmente completando a combinaçção necessária para ativar ou reprimir um gene. Um exemplo disso em humanos é o caso do receptor de glicocorticóide. Para se ligar aos sítos no DNA o receptor precisa formar um complexo com uma molécula de um hormônio esteróide (p.ex. cortisol). Em resposta aos hormônios glicocorticóides, as células do fígado aumentam a expressão de vários genes. Expressão gênica 20 Efeito de uma Única Proteína Reguladora na Diferenciação • • Estudos com células musculares em diferenciação, em cultura, possibilitaram a identificação de proteínas reguladoras importantes, expressadas somente em céleulas musculares, que coordenam a expressão gênica. Quando o gene que codifica uma dessas proteínas reguladoras, MyoD, é introduzido em fibroblastos, eles passam a se comportar como mioblastos e fundem-se para formar células semelhantes às musculares. Expressão gênica 21 Um Único Gene que Codifica uma Proteína Reguladora Pode Estimular a Formação de um Órgão Inteiro Estudos sobre o desenvolvimento de olho em Drosophila, camundongo e humanos mostraram que um único gene que codifica uma proteína reguladora (Ey em moscas flies e Pax6 em vertebrados) é crucial para o desenvolvimento do olho. Quando expressado num tipo celular apropriado, Ey pode desencadear a formação de um órgão inteiro (olho), composto de diferentes tipos de células, todas corretamente organizadas no espaço tridimensional. Expressão gênica 22 Os processos de iniciação, alongamento, processamento e terminação da transcrição são acoplados em eucariotos Maniatis & Reed NATURE |VOL 416 | 4 APRIL 2002 Expressão gênica 23 Acoplamento das maquinarias envolvidas na transcrição, capping, splicing e poliadenilação Maniatis & Reed NATURE |VOL 416 | 4 APRIL 2002 Expressão gênica 24 Modelo de acoplamento de splicing a exportaçao do mRNA e decaimento mediado por nonsense (NMD) Maniatis & Reed NATURE |VOL 416 | 4 APRIL 2002 Expressão gênica 25 Influência da Estrutura da Cromatina na Transcrição em Eucariotos • A maior parte do DNA em uma célula eucariótica está complexada nos nucleossomos e a estrutura espiralada dificulta o acesso de fatores de transcrição e RNApolimerase. • A iniciação da transcrição depende da remoção dos nucleossomos da região promotora do gene. – – Durante a síntese de DNA, quando os nucleossomos são substituídos, poderia haver competição entre as histonas e os fatores de transcrição (p.ex. TFIID) pelos sítos promotores. A ligação e dirupção dos nucleossomos por ativadores. Expressão gênica 26 Disrupção e Reorganização do Nucleossomo • Complexos poderiam estar envolvidos na disrupção dos nucleossomos: – – – • Participação de fator GAGA e fator de remodelamento de nucleossomo (nucleosome-remodeling factor, NURF) Participação de complexo SW1/SNF Existe uma boa correlação entre acetilação de histona e a atividade transcricional da cromatina. Competição entre histonas e fatores de transcrição poderia estar envolvida no controle da expressão genica. Expressão gênica 27 Regiões Controladoras de Lócus (Locus Control Regions, LCRs) • • Regiões controladoras de lócus (LCRs) são seqüências de DNA essenciais para o estabelecimento de uma configuração “aberta” da cromatina. Elas são capazes de inibir a repressão normal da transcrição sobre áreas relativamente grandes contendo vários genes. Um dos mais bem estudados é o LCR que controla a expressão tecido- específica da família de -globin. Expressão gênica 28 Expressão gênica 29 Expressão gênica 30 Correlações entre a Metilação do Promotor e Inatividade Gênica • • • Em células sangüíneas vermelhas de humanos e galinhas, o DNA envolvido na síntese de globina está completamente (ou quase completamente) não-metilado. O gene de ovalbumina de galinha ñão está metilado nas células do oviduto, mas metilado nos outros tecidos. Nos somitos de camundongo, a demetilação de um “enhancer” de MyoD precede a transcrição de MyoD e é essencial para a especificação dessas células como precursoras de músculo. Expressão gênica 32
Download