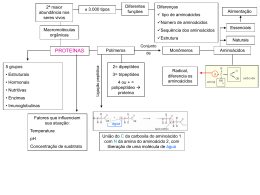
BCM13042 Fundamentos de Análises de Proteínas • Docentes – – – – Célia R Carlini Charley C. Staats Diogo R Demartini Hugo Verli • Súmula – Aminoácidos e peptídeos: estrutura; estereoquímica; propriedades físico-químicas; curva de titulação; ponto isoelétrico; ligação peptídica – Proteínas: arquitetura de proteínas; tipos de estrutura secundária; enovelamento; modificações pós-traducionais; mobilidade eletroforética; flexibilidade; catálise – Métodos para determinação da estrutura de proteínas: RMN; cristalografia de raios-X; Dicroísmo circular – Espectometria de massas: metodologias de ionização; metodologias de análise. – Métodos para determinação da quantidade de proteínas: gravimétrico; absorbância; fluorescência; ninhidrina; biureto; lowry; azul de comassie; acido bicinchônico. – Métodos de separação, purificação se sequenciamento de proteínas, peptídeos e aminoácidos: precipitação; salting in; salting out; eletroforese; cromatografia liquida, ultracentrifugação; química de Edman; métodos de separação de aminoácidos. BCM13042 Fundamentos de Análises de Proteínas • • • • • 22/11 Aminoácidos e peptídeos: Charley 23/11 Estrutura e determinação da estrutura de proteínas: Hugo 24/11 Métodos de quantificação, análise e purificação de proteínas: Célia 25/11 Espectrometria de massas e proteômica 29/11 – 02/12 Seminários – 1 tópico por dia – 5 grupos por tópico • Tópico 1 – Métodos de Análise e Caracterização de Peptídeos 1. 2. 3. 4. 5. Papéis Biológicos de D-Aminoácidos Determinação de sequencia de aminoácidos/peptídeos (Edman, MS-MS, Dansylação) Assinalamento de padrões (bioinformática) Modificações pós-traducionais: tipos, funções e impactos em análises Síntese de peptídeos para avaliação biológica • Tópico 2 – Métodos de Análise e Caracterização de Peptídeos 6. 7. 8. 9. 10. Eletroforeses (1D, 2D, nativa, zimogramas) Cromatografia (troca iônica, hidrofóbica, fase reversa, afinidade) Metodologias de quantificação de proteínas Precipitação fracionada Métodos Imunoquímicos • Tópico 3 – Espectrometria de massas e proteômica 11. 12. 13. 14. 15. MudPit / GELC – MS Proteômica quantitativa comparativa Metaloproteômica Fosfoproteômica Interatoma • Tópico 4 – Determinação de estrutura 16. 17. 18. 19. 20. RMN Dicroísmo circular Fluorometria Cristalografia – Raios X Determinação de motivos estruturais BCM13042 Fundamentos de Análises de Proteínas Avaliação • Apresentação de 1 item referente a cada tópico • Respostas aos questionários aplicados pelos colegas • Cada aluno deverá formular 1 questão sobre o item apresentado • Os demais alunos deverão escolher 2 questões referentes a cada tópico para responder • As questões devem ser embasadas em um artigo científico. Esperase que a questão seja formulada a partir de um gráfico ou tabela presente no artigo científico. • O autor da questão será responsável pela correção. • Aulas serão disponibilizadas junto ao endereço www.ufrgs.br/laprotox Sorteio Amino-Ácidos Estrutura geral dos Aminoácidos Quantos aminoácidos existem em sistemas biológicos? 20 aminoácidos definidos por seu códon no mRNA Grupo R 5 grupos distintos 2 aminoácidos não usuais Inserção depende de um elemento cis no mRNA Polar - aquoso Apolar - lipídico Polar - aquoso Selenocystein Pyrrolysine Hypothetical scheme for the cotranslational insertion of pyrrolysine in response to a contextdependent UAG codon. Ibba M , Söll D Genes Dev. 2004;18:731-738 ©2004 by Cold Spring Harbor Laboratory Press Formação de pontes de enxofre Estereoisomeros a-carbon is a chiral center Two stereoisomers are called enantiomers. The solid wedge-shaped bonds project out of the plane of paper, the dashed bonds behind it. The horizontal bonds project out of the plane of paper, the vertical bonds behind. The Stereochemistry of Amino Acids http://www.imb-jena.de/~rake/Bioinformatics_WEB/gifs/amino_acids_chiral.gif D-aminoácidos existem e são metabolizados Serine-racemase D-amino ácidos em proteínas? Características físico-químicas dos aminoácidos Absorbância na região UV de aminoácidos aromáticos Estudos conformacionais – Try fluorescence Changes in low density lipoprotein receptor intrinsic tryptophan fluorescence upon calcium addition. Dirlam-Schatz K A , Attie A D J. Lipid Res. 1998;39:402-411 ©1998 by American Society for Biochemistry and Molecular Biology Zwitterion = íon híbrido Voet Biochemistry 3e © 2004 John Wiley & Sons, Inc. Efeito sobre o pKa do ambiente químico Henderson/Hasselbach equation and pKa Protonated form HA ↔ Unprotonated form (conjugate base) H+ + A- Ka = [H+] [A-] / [HA] [H+] = Ka [HA]/ [A-] -log [H+] = -log (Ka [HA]/ [A-]) -log [H+] = -log Ka -log ([HA]/ [A-]) pH = p Ka - log ([HA]/ [A-]) Titulação de um aminoácido Curva de titulação Glutamato Curva de titulação> Histidina Anel indólico Peptídeos Formação da ligação peptídica Níveis estruturais das proteínas Como se definem estruturas secundárias? Distâncias interatômicas na ligação peptídicas são menores que as de uma ligação amida comum Ressonância eletrônica Ligação peptídica tem 50% de carácter de dupla ligação rígida e coplanar Ângulos de rotação da cadeia polipeptítica em torno de um carbono alfa Cadeias laterais A B C D A B D C E E F Gráfico de Ramachandran F O que é um domínio protéico? Procura de domínios conservados Localização da proteína pode trazer informações sobre a sua função
Baixar