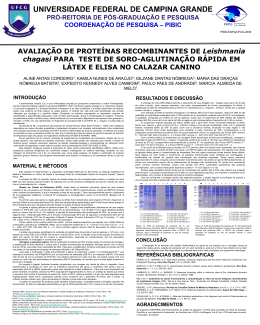
AVALIAÇÃO DE PROTEÍNAS RECOMBINANTES DE Leishmania chagasi PARA TESTE DE SOROAGLUTINAÇÃO RÁPIDA EM LÁTEX E ELISA NO CALAZAR CANINO ALINE ANTAS CORDEIRO1; KAMILA NUNES DE ARAÚJO2; GILZANE DANTAS NÓBREGA3; MARIA DAS GRAÇAS NÓBREGA BATISTA4; EXPEDITO KENNEDY ALVES CAMBOIM5, PAULO PAES DE ANDRADE6, MARCIA ALMEIDA DE MELO7 1Bolsista PIBIC, aluna do Curso de Medicina Veterinária, Unidade Acadêmica de Medicina Veterinária, UFCG, Patos - PB, E-mail: [email protected] 2 Aluna do Curso de Medicina Veterinária, Unidade Acadêmica de Medicina Veterinária, (UAMV) UFCG, Patos - PB, E-mail: [email protected] 3Aluna do Curso de Medicina Veterinária, Unidade Acadêmica de Medicina Veterinária, UFCG, patos - PB, E-mail: [email protected] INTRODUÇÃO A leishmaniose visceral (LV) é uma enfermidade causada por protozoário pertencente à ordem Kinetoplastida, família Trypanosomatidae e gênero Leishmania (RIBEIRO, 1997). No Brasil,o agente etiológico é a Leishmania chagasi, a Lutzomyia longipalpis é o principal flebótomo transmissor e os cães constituem o principal reservatório da doença; sendo uma endemia de forte impacto na saúde pública. O controle tem sido feito pela identificação dos animais infectados por sorologia e o sacrifício destes. Entretanto, os métodos empregados no diagnóstico carecem de sensibilidade e especificidade adequados e são de difícil padronização, devido à complexidade do antígeno. Proteínas recombinantes podem contribuir para o desenvolvimento de novos ensaios diagnósticos mas apenas duas ganharam o mercado, a rK39 e S7, que representam um fragmento da quinesina K39 e da HSP70 de L. chagasi ,e muitas estão já patenteadas. Na década de 80, a importância das proteínas de choque térmico (Heat Shock Proteins – HSP) como antígenos majoritários em várias infecções parasitárias e bacterianas começou a ser revelada. Adicionalmente, ficou evidenciada uma variação importante da expressão das HSP’s durante a diferenciação de diversos parasitas, no estresse provocado no momento em que o parasita se instala no vetor (ou é cultivado em meio de cultura) ou quando é exposto ao aumento de temperatura após inoculação nos hospedeiros vertebrados (FEDER & HOFMAN, 1999). Na era genômica há um número crescente de seqüências de nucleotídeos de Leishmania, obtidos não só no projeto genoma, mas também de trabalhos individuais. Esses dados podem ser utilizados para estudar a função de diversos genes, podendo esclarecer aspectos da relação hospedeiro/parasita e eventualmente ser utilizados como instrumento de diagnóstico e ainda indicar possíveis alvos quimioterápicos (GONTIJO & MELO, 2004). Este trabalho, propõe-se estudar a expressão de vários genes previamente identificados (MELO, 2006) como produzindo proteínas antigênicas de Leishmania chagasi, empregando um sistema heterólogo composto de uma linhagem de Escherichia coli e de um plasmídeo que, numa etapa posterior, irá facilitar a purificação da proteína quimérica gerada. Nenhum dos antígenos selecionados está descrito na literatura ou foi patenteado dentro ou fora do país. RESULTADOS E DISCUSSÃO O emprego do meio NNN bifásico permitiu o isolamento de uma linhagem de L. chagasi, após cerca de 30 dias de cultivo primário. Após repiques semanais, uma maior homogeneidade de formas promastigotas foi obtida. A amplificação do DNA de cinetoplasto pelos primers E e F, específicos pra L. chagasi/ L. infantum, mostra que o parasita isolado é de fato L. chagasi. Com extração do DNA do parasita empregando o kit DNeasy Blood and Tissue (Qiagen), obteve-se DNA de alta qualidade em quantidades adequadas para o PCR partindo de um precipitado contendo cerca de 5X107 promastigotas. A qualidade, averiguada por análise em gel de agarose, mostra que os fragmentos de DNA obtidos são bastante grandes (500 Kb) e que não há um rastro de DNA na região do gel correspondente a fragmentos de 10 Kb ou menos. As seqüências originais descritas por MELO (2006) para o gene SH01 foram novamente blastadas no NCBI. Sendo que, apenas o gene LinJ34.1100 de L. infantum mostrou uma elevada similaridade, tanto com a sequência forward, localizada dentro da ORF do gene, como com a seqüencia reverse, localizada na longa região 3´ não traduzida. Primers foram então desenhados para amplificar a parte 3´terminal da ORF, correspondendo a um polipeptídeo carboxi-terminal da proteína SH01 de aproximadamente 300 aa. As seqüências são: Primer SH01 forward: 5’ - TCG AAC GAT GAG CTG AAC A – 3’ e Primer SH01 reverse: 5’ – AGT GTC GCC TGG CAT ATT TC – 3’. Como controle positivo do experimento de clonagem foram desenhados primers para amplificação, clonagem e expressão do gene da HSP70 de L. chagasi. O segmento escolhido foi a parte 3´ terminal, que codifica um polipeptídeo de cerca de 270 aa, correspondente aproximadamente à proteína S7. As seqüências são: Primer HSP70 forward: 5’ – CAG GCC TTC ATC CTG ACG – 3’ e Primer HSP70 reverse: 5’ – TTA GTC GAC CTC CTC GAC CTT – 3’. O uso do kit de purificação para os produtos de PCR permitiu obter uma banda única consistente, sem dímeros de primers, tanto para o produto de SH01 como para o de HSP70 (dados não apresentados). Em seguida, foram feitas as ligações destes genes e a transformação da hospedeira. Nos dois casos numerosos clones brancos foram obtidos. Após repique em meio líquido e crescimento, as bactérias transformadas foram coletadas por centrifugação e solubilizadas em tampão de amostra para visualização das proteínas expressas. Vários clones cresceram vigorosamente em cultura, enquanto outros mostraram crescimento pobre. Na análise em gel de SDS-poliacrilamida, os clones com crescimento vigoros não se diferenciaram da linhagem não transformada. Os clones com crescimento pobre, contudo, parecem expressar, mesmo sem indução com IPTG, quantidades significativas das proteínas recombinantes, com os pesos moleculares esperados (clones 4, 11 e 20) (figuras 1a e 1b). Embora estes resultados tenham ainda que ser confirmados por western blot e PCR, eles indicam que a ligação pode conduzir, à produção de plasmídeos sem inserto ou com fragmentos pequenos, que impedem a expressão da βgalactosidase, sem conduzir à expressão da proteína quimérica planejada. Uma vez confirmada a clonagem destes dois segmentos gênicos, o projeto seguirá na purificação do produto e uso em testes de ELISA e aglutinação rápida em látex para o sorodiagnóstico do calazar. MATERIAL E MÉTODOS Este trabalho foi desenvolvido no Laboratório de Biologia Molecular do Semi-árido na Unidade Acadêmica de Medicina Veterinária no Centro de Saúde e Tecnologia Rural da Universidade Federal de Campina Grande, Campus Patos - PB. A extração de DNA do parasita, isolado de medula óssea de cão sorologicamente positivo e mantido em meio NNN com fase líquida composta de BHI, foi realizada com um kit comercial, empregando-se para tal o lavado da cultura rica em promastigotas de L. chagasi. Reação em Cadeia da Polimerase (PCR): Nesta etapa os trabalhos enfocaram genes de uma proteína hipotética e de uma proteína com função já comprovada: SH01 e HSP70, respectivamente. O gene da HSP70 foi incluído como controle do experimento, já que a expressão correta da sua porção 3´ deve necessariamente permitir o isolamento de um polipeptídeo antigênico, a proteína S7 empregada no sorodiagnóstico canino no kit ELISA-S7 (Biogene Ind. & Com. LTDA). Os primers para cada gene ou região gênica escolhida foram desenhados pelo programa Primer3, disponível no site http://fokker.wi.mit.edu/primer3/input.htm. Além das características determinadas pelo Primer3 foram feitos ajustes no primer forward para que a seqüencia clonada ficasse no quadro de leitura do plasmídeo. Os genes e regiões gênicas do genoma nuclear de L. chagasi foram amplificados utilizando-se em uma reação: tampão 1X, dNTP 250 μM, 1 μM de cada primer, 2U de Taq polimerase, 5 μL de DNA (1/10) e água destilada ajustada para um volume final de 20 μL. Em seguida, a amostra foi termociclada para amplificação do DNA de acordo com o seguinte ciclo: 1-Desnaturação 95ºC por 3 minutos; 2-Desnaturação 95ºC por 30 segundos; 3-Anelamento 60ºC por 30 segundos; 4-Extensão 72ºC por 30 segundos; 5-Repetir 2ª etapa, 40 vezes; 6-Extensão 72ºC por 10 minutos; 7-1 ciclo 4ºC infinito; 8-Fim (ARAÚJO et al., 2008 dados não publicados). Foram usados como controle positivo do ensaio, primers espécie-específicos que pareiam com o kDNA (DNA de cinetoplasto), descritos por LAMBSON (1999), com as seguintes seqüências: E: 5’ – CCA GTT TCC CGC CCC G – 3’; F: 5’ – GGG GTT GGT GTA AAA TAG G – 3’). Como controle negativo nenhum DNA foi adicionado ao mesmo mix da reação para kDNA. A análise dos produtos amplificados foi realizada por eletroforese horizontal em gel de agarose a 1,2% (p/v), com tampão de corrida TBE 0,5X (0,045M Tris-borato e 1mM de EDTA, pH 8). A imersão do gel numa solução de brometo de etídio (0,5 μg/mL) foi feita por 40 minutos e, posteriormente, observada em transluminador com luz ultravioleta. Alternativamente, foi empregado o corante blue green. Clonagem e expressão gênica: Para a purificação do produto de PCR, primeira etapa no processo de clonagem, a amplificação foi feita utilizando o volume para 5 reações convencionais de avaliação, descritas acima, com o volume final de 100 μL, para obter maior quantidade de DNA. Logo após a amplificação, foi feita a purificação do produto de PCR dos genes ou dos segmentos de genes de interesse utilizando o kit QIAquick® Spin. Logo após a purificação, o DNA pré-amplificado é ligado ao plasmídeo, usando o kit QIAexpressionist™ ,que emprega a Escherichia coli M15[pREP4] com um plasmídeo de expressão pQE-30 e um repressor pREP4. O kit lança mão do fato de que os produtos de PCR têm uma adenina adicional em cada nova fita, que podem parear com uma uracila em cada uma das extremidades do plasmídeo pQE-30 linearizado, de maneira que a enzima ligase presente no kit promove a ligação. Em seguida à ligação, bactérias tornadas competentes, com lavagem em cloreto de cálcio e magnésio, foram transformadas e incubadas em meio com os antibióticos kanamicina (25 g/mL) e ampicilina (100 g/mL), pois os plasmídeos pQE-30 e pREP4 apresentam genes para resistência a estes antibióticos. Para que ocorra expressão dos genes inseridos na bactéria, adiciona-se IPTG (isopropil-β-D-tiogalactosídeo) no meio, um análogo da lactose que inativa o repressor lacZ e induz a transcrição do operon lac. Além do IPTG, utiliza-se também o X-gal, que será degradado pela β-galactosidase. Desta forma, aparecendo colônias azuis, mostra-se que o plasmídeo se fechou sem o inserto. Já colônias brancas indicam que o gene de interesse foi clonado e pode estar sendo expresso. A análise da expressão de proteínas pelas bactérias transformadas, comparativamente à linhagem parental não transformada, foi feita por separação eletroforética em gel de SDS-poliacrilamida a 12%, corado com azul de Coomassie. A análise da expressão de proteínas pelas bactérias transformadas, comparativamente à linhagem parental não transformada, foi feita por separação eletroforética em gel de SDS-poliacrilamida a 12%, corado com azul de Coomassie Realização: Promoção: Apoio: Figura 1a: Perfil da expressão de proteínas por E. coli transformada pelo plasmídeo pQE-30 portando o inserto para o Figura 1b: Perfil da expressão de proteínas por E. coli transformada pelo plasmídeo pQE-30 portanto o inserto para o segmento Figura 1a: Perfil da expressão de proteínas por E. coli transformada pelo plasmídeo pQE-30 portando o inserto para o segmento carboxi-terminal das proteínas SH01 e HSP70. Os números, após a descrição da presença ou não de indução, indicam o clone empregado. Figura 1b: Perfil da expressão de proteínas por E. coli transformada pelo plasmídeo pQE-30 portanto o inserto para o segmento carboxiterminal das proteínas SH01 e HSP70. Os números após a descrição da presença ou não de indução indicam o clone empregado. CONCLUSÃO A estratégia de se associar uma análise bioinformática de genes de um parasita com a clonagem direta de segmentos destes genes, seguida da expressão em um vetor bacteriano, pode ser a forma mais eficiente de se obter antígenos recombinantes para fins diagnósticos ou proteínas para outros usos, produzidas por organismos heterólogos. AGRADECIMENTOS Ao programa CNPq/PIBIC pelo financiamento do projeto de pesquisa, à UFCG pela concessão da bolsa de Iniciação Científica e aos membros do Laboratório de Genômica da UFPE e da Biogene Ind & Com. LTDA pelo apoio durante a execução do trabalho.
Baixar