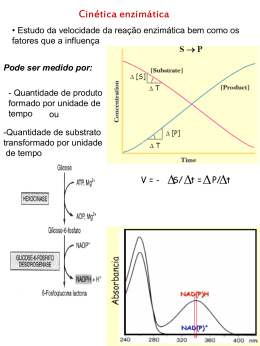
Bioquímica Cinética das reações enzimáticas Energia livre, G Estado de transição Coordenada da reação Cinética das reações Relembrando: A+B B+C V = k[A] [B] S P Se a velocidade é calculada em função de um intervalo de tempo será obtida a velocidade média Cinética das reações A B Para medir a velocidade efetiva proporcional a concentração de reagente: - v0: velocidade inicial Calculada num pequeno intervalo de tempo durante o qual a conversão de A em B tenha sido tão tão reduzida que a concentração de A possa ser considerada constante. - t: tempo inicial Intervalo de tempo do cálculo da v0 Cinética das reações Catálise enzimática ocorre em duas etapas: 1) 2) E + S ES E + S k1 k-1 k2 ES k1 k-1 ES E + P k2 E + P Cinética das reações E + S k1 k-1 ES k2 E + P v1 = k1 [E][S] v-1 = k-1 [ES] v2 = k2 [ES] Michaelis e Menten admitiram: k-1>> k2 Cinética das reações E + S k1 k-1 ES k2 E + P Sendo k-1>> k2: - A primeira reação chega a estabelecer equilíbrio - Velocidade global v2 – sendo esta a etapa mais lenta e limitante do processo Estes pressupostos são válidos para as enzimas michaelianas Formação do complexo transitório Influência da concentração de substrato Variação das concentrações dos componentes em função do tempo Velocidade da reação enzimática • Quando a [S] for muito grande, a concentração de [E] é praticamente nula, estando toda enzima na forma ES: E+S ES ±0% ±100% • A partir desta quantidade de [S] a Velocidade será constante, dessa forma atingimos o Vmax da reação. • Quando 50% [E] e 50% [ES], a concentração de S nesta velocidade será igual a KM Equação de Michaelis-Menten LEONOR MICHAELIS 1875 - 1949 MAUD MENTEN 1879 - 1960 Equação de Michaelis-Menten Para dedução da equação de MichaelisMenten é considerada a reação reversível: E+S k1 k2 ES k3 k4 E+P KM = (k2 + k3)/k1 Obtendo Vmax Variação da velocidade da reação enzimática (v 0) em função da concentração do substrato (S) Velocidade da reação enzimática Fonte: Stryer Enzima Michaeliana Enzima Alostérica Afinidade da enzima Constante de Michaelis-Menten – KM – seu valor indica a afinidade que uma enzima apresenta pelo seu substrato: glicose hexoquinase enzima Açucares de 6 C substrato frutose glicose Necessita 0,15 mM para atingir VMax/2 frutose Necessita 1,5 mM para atingir VMax/2 Afinidade da enzima Variação da velocidade da reação em função do substrato Efeito da variação da enzima A velocidade só é diretamente proporcional em pequenas concentrações de substrato Variação da velocidade da reação (v0) em função da concentração de enzima [E] Dosagem da enzima - É feita pela medida de sua atividade - Incuba-se a enzima com altas concentrações de Enzimas GARANTIR A VMAX - Expressa em unidades internacionais (U) Quantidade de enzima para formar 1 μmol de produto - Medir concentração da atividade de enzimas auxilia no acompanhamento de casos patológicos Dosagem da enzima Transformação de Lineweaver-Burk •Equação descrita em 1934 Hans Lineweaver Dean Burk Químico dos EUA Fisicoquímico dos EUA Determinação de Vmax e KM Fonte: Enzyme Determinação de Vmax e KM KM inclinação = V max Eficiência da catálise enzimática - Constante catalítica: kcat Eficiência máxima – condições de VMAX, em que todas enzimas estão complexadas com o substrato - Relação da eficiência catalítica com a afinidade pelo substrato kcat/ KM Baixo valor → pouca afinidade pelo substrato → baixa eficiência catalítica O critério kcat/KM • Usado para comparar diferentes enzimas • Relaciona a eficiência catalítica da enzima com sua afinidade pelo substrato. • kcat/KM baixo: a enzima tem pouca afinidade pelo Substrato (alto KM); ou porque a eficiência em gerar ES é pequena (baixo kcat); ou por ambos motivos. • Os maiores valores são dados para altas eficiência em gerar ES (alto kcat) e pela alta afinidade pelo substrato (baixo KM) O critério kcat/KM • Exemplos de enzimas de alta eficiência: Fonte: Marzzoco & Torres, 2007 Inibidores enzimáticos - Irreversíveis - Reversíveis - Competitivos - Não Competitivos Exemplo de inibidor irreversível: Grupo acetila Responsável pela 1a reação da via de síntese de prostaglandinas Inibidores Irreversível Fonte: Enzyme Inibidores Irreversível Ex. Compostos organofosforados Formam ligações covalentes com o grupo OH de resíduos de serina Praticamente irreversível Malation – Defensivo agrícola Diminui KM e Vmax Acetil colinesterase Inibidores Irreversível Ex. Carbamatos Carbaryl – Inibe acetil colinesterase (inseticida) Praticamente irreversível Inibidores Irreversível Ex.: Penicilina: Liga-se especificamente a enzimas da via de síntese de parede bacteriana. Iodoacetamida: Reage com o grupo SH de resíduos de cisteína. Inibidores competitivos Fonte: Enzyme Inibidores competitivos Exemplos: Inibidores competitivos Inibidores competitivos Inibidores não competitivos Fonte: Enzyme Inibidores não competitivos Inibidores não competitivos Inibidores incompetitivos - Semelhante ao não competitivo; - O inibidor só consegue se ligar após formação do complexo ES. Fonte: Enzyme Inibidores competitivos e não competitivos Análogos de enzimas - Análogos ou antimetabólitos Tem fórmula estrutural semelhante ao S - Liga-se ao centro ativo e resulta num produto Produto diferente da via “correta” Ex. Espécies análogas de aminoácidos em plantas Azetidina 2-carboxilato Prolina Canavanina Arginina Antimetabólitos Antimetabólitos Regulação - Muitas enzimas tem sua atividade catalítica regulada Ex. A B C D Z Regulação Fonte: Enzyme Regulação – Moduladores alostéricos Fonte: Enzyme Regulação Algumas enzimas são sintetizadas numa forma inativa: ZIMOGÊNIO Ex: Pepsina pepsinogênio Quimiotripsina quimiotripsinogênio Regulação Isozimas - Formas diferentes de uma mesma enzima - Atuam em tecidos diferentes Ex: Lactato desidrogenase Músculo esquelético (MMMM) Coração (HHHH e HHHM) Regulação Ligação covalente Algumas enzimas são reguladas por modificação covalente na molécula Podem ser: - Fosforilação - Adenilação - Uridilação - ADP-ribosilação - Metilação Regulação: Ligação covalente Fosforilação Regulação: Ligação covalente Adenilação Regulação: Ligação covalente Uridilação Regulação: Ligação covalente ADP-ribosilação Regulação: Ligação covalente Metilação
Baixar