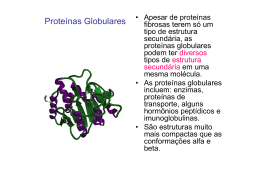
Introdução a Bioquímica: Biomoléculas Níveis estruturais das proteínas Aula 6 Estruturas 3D, domínios motivos Ignez Caracelli Julio Zukerman Schpector BioMat – DF – UNESP/Bauru LaCrEMM – DQ – UFSCar 1º 2º 3º 4º A cadeia polipeptídica se enovela (fold) para gerar uma estrutura 3-D única. Bauru, 24 de setembro de 2007 1. A estrutura 3-D de uma proteína é determinada por sua seqüência de aminoácidos. 2. A função de uma proteína depende de sua estrutura. 3. Cada proteína tem uma estrutura única. As proteínas tem uma conformação específica, fica o arranjo espacial dos átomos. As proteínas na sua conformação funcional e enovelada estão em seu estado nativo. nativo quimotripsina Ângulo diédrico φ φ plano da amida Ângulo diédrico ψ • é o ângulo de torção em torno de N− − Cα : C’ Cα carbono α cadeia N lateral • é a projeção de C’(i-1) –N sobre Cα–C’ [C’(i-1) –N–Cα–C’] N(i+1) ψ é o ângulo de torção em torno de Cα–C’: plano da amida C’ é a projeção de N− − Cα sobre C’– N(i+1) [N− − Cα− −C’– N(i+1)] ψ Cα carbono α cadeia N lateral C’(i-1) plano da amida plano da amida Hélice α átomos Cα C-terminal N-terminal 3,6 aminoácidos por volta passo = 5.4 Å 1 Å = 10-10 m grupos-R → para fora Rotações podem ocorrer em torno do Cα ligações de H entre (1) C=O===H-N (4) COO- Hélice α Hélice α α-hélices na proteínas são hélices de mão direita NH3+ Hélice α - dipolo elétrico Hélice α - estabilidade Devido a 1. repulsão ou atração eletrostática dos aminoácidos adjacentes. dipolo total 2. interação entre os aminoácidos n3 ou n4. 3. ocorrência de resíduos de Pro ou Gly. dipolo em cada interação Hélice α Hélice α - backbone (esqueleto) N nitrogênio C carbono α C carbono carboxila ___ Hélice 310, α, π Hélice 310 3,0 resí resíduos/ volta passo = 6,0 Å mais fina e alongada que α. Hélice α 3,6 resíduos/volta passo = 5,4 Å. ligação de hidrogênio Hélice 310, α, π Hélice π 4,4 resíduos/volta passos = 5,2 Å mais larga e menor que a α. 3,0 resí resíduos/ volta passo = 6,0 Å mais fina e alongada que α. 3,6 resíduos/volta passo = 5,4 Å. 4,4 resíduos/volta passos = 5,2 Å mais larga e menor que a α. Hélice 310, α, π 3,0 resí resíduos/ volta passo = 6,0 Å mais fina e alongada que α. 3,6 resíduos/volta passo = 5,4 Å. 4,4 resíduos/volta passos = 5,2 Å mais larga e menor que a α. Estruturas Beta Anti-paralelas vista de cima vista lateral Hélice 310, α, π 3,0 resí resíduos/ volta passo = 6,0 Å mais fina e alongada que α. 3,6 resíduos/volta passo = 5,4 Å. 4,4 resíduos/volta passos = 5,2 Å mais larga e menor que a α. Estruturas Beta Paralelas vista de cima vista lateral Estruturas “turns” Gráfico de Ramachandran Gráfico de Ramachandran http://www.ebi.ac.uk/thornton-srv/databases/pdbsum/ 150l Pyruvate Kinase 1f0w 1bkv Proteinas globulares e fibrosas 1bkv 1f0w Colágeno Proteinas fibrosas 1bkv Estrutura supersecundária - motivos Há padrões comuns no enovelamento das proteínas chamados de motivos. Os motivos são encontrados em várias proteínas de função diversa. tripla hélice de mão-esquerda Uma proteína pode conter vários motivos estruturais diferentes. A estrutura de cada proteína é totalmente diferente ou há motivos comuns? Estrutura supersecundária - motivos Há padrões comuns de enovelamento da cadeia polipeptídica na maioria das proteínas. Exemplos: α-hélice folhas-β Estrutura supersecundária - motivos Pyruvate Kinase Piruvato Cinase Christine Orengo (Structures, 1997, 5, 1093-1108) Estrutura terciária das proteínas Domínios estruturais As proteínas se enovelam em estruturas globulares e excluem H2O de seu interior. Em geral: Aminoácidos não-polares → no interior Val, Leu, Ile, Met, Phe Aminoácidos carregados → na superfície Arg, Lys, His, Asp, Glu Aminoácidos polares não-carregados → na superfície ou interior Ser, Thr, Asn, Gln, Tyr, Trp Estrutura Quaternária de Proteínas domínio pequeno NADPH domínio grande Estrutura Quaternária de Proteínas Razoes para múltiplas subunidades: Algumas proteínas formam agregados de 2 ou mais subunidades. subunidades 1. cooperatividade exemplo: Hb liga O2 cooperativamente. 2. função catalítica exemplo: HMG-CoA reductase dímero 3. síntese exemplo: groEL chaperonina tem 14 subunidades (a proteína é muito grande para ser sintetizada sozinha. Colágeno 35% Gly 21% Pro + Hidroxiprolina HMG-CoA redutase unidade de repetição: Gly – X – Pro (HyPro) monômero hélice de mão esquerda (não α-hélice) com 3 aa/volta dímero Diagramas - Motivos Diagramas - Motivos helice Motivos Hairpin Motivo Helix-Loop-Helix (H-L-H) Motivo EF-Hand H-L-H B/T- Figure 2.13 Motivo Chave-grega Ligantes Sítio de Ligação Hemoglobina: transporte de O2 no sangue α2β2 Proteínas ligam-se a outras moléculas chamadas ligantes. A interação de uma proteína com um ligante é especifica e reversível. α1 α2 O local de ligação do ligante na proteína é chamado de sítio de ligação. A ligação de um ligante resulta em mudança conformacional na proteína β1 β2 Grupo Heme grupo prostético com: •anel da porfirina •átomo de Fe no estado ferroso (Fe2+) Mioglobina liga O2 nas células musculares. O grupo heme se localiza em um bolso. Heme O Fe2+ é coordenado por 4 nitrogênios pirrolícos Fe2+ X Fe3+ A 5a. posição de coordenação do Fe2+ é ocupada por uma histidina chamada de proximal. A 6a. posição de coordenação é o sítio de ligação do O2. (proximal histidine) Mioglobina Hemoglobina As subunidades da hemoglobina são estruturalmente similares à mioglobina grupo heme Especificidade do ligante no Heme Monóxido de carbono (CO) liga-se 20.000 vezes melhor que O2 Mioglobina liga CO 200 vezes melhor que O2 Interferência estérica da Mioglobina com a ligação do CO ao heme A histidina distal interage com o ligante (CO ou O2). mioglobina subunidade β da hemoglobina Distal His Proximal His Eritrócitos humanos (células vermelhas do sangue) Pulmões pH = 7,6 Tecido pH = 7,2 Hemoglobina libera CO2 nos pulmões e liga O2. O2 O2 CO2 H+ Hemoglobina libera O2 e liga CO2, H+ nas extremidades ERITRÓCITOS Anemia Falciforme normais Glu 6 anemia falciforme Glu 6 Val 6 Val 6 anemia falciforme Hemoglobina: Cooperatividade A hemoglobina é uma proteína alostérica. A ligação de um ligantes afeta a ligação dos ligantes nos outros sítios da proteína. Hb e cooperatividade α1 β1 O sistema imunológico α1 O2 β2 α2 β2 O2 O2 β1 α2 O2 O2 • responde a bactérias, vírus e proteínas nos fluidos corporais. • utiliza anticorpos (imunoglobulinas, Ig), proteinas que se ligam a moléculas estranhas e consideram alvo para destruição. • as moléculas estranhas reconhecidas pelos anticorpos são antigenos. •o anticorpo reconhece uma certa região do chamada de epítopo. P. ex: uma seqüência pequena de aa. •os anticorpos são produzidos pelos linfócitos B. Sistema Imunológico Celular Proteínas MHC (Major Histocompatibility Complex) •destrói células infectadas. são centrais na produção da resposta imunológica). •utiliza macrófagos, células que engolem partículas grandes •e células infectadas. ligam-se a proteínas digeridas na célula. •linfócitos T reconhecem células infectadas através dos receptores da célula T sobre a superficie. proteínas MHC I estão sobre a superfície das células . proteínas MHC II estão sobre a superfície dos macrófagos e células B Proteínas MHC Estrutura da proteína MHC I ligada a um antígeno. O antígeno é um peptídeo derivado do HIV. Anticorpos proteína MHC I O sistema imunologico utiliza anticorpos, ou imunoglobulinas, (Ig). Anticorpos são proteínas que se ligam a moléculas estranhas para destruí-las. Há vários tipos de imunoglobulinas presentes no sangue: peptídeo HIV IgA, IgD, IgE, IgG, IgM. Estrutura do IgG 150.000 D Classes de Ig IgA, IgD, IgE IgG, IgM (≠ na cadeia pesada) Estrutura do IgG Movimento Molecular Há movimento na natureza. O movimento é mediado por proteínas. A energia para o movimento normalmente está na forma de ATP Miosina Filamento fino. Miosina 6 subunidades, PM = 540.000 D Contração do músculo ocorre quando filamentos finos e grossos deslizam uns sobre os outros. relaxado contraído O sistema imunológico hidrólise do ATP ATP O sistema imune é nossa defesa contra invasores. O sistema deve ser capaz de reconhecer o que é próprio ou estranho. A resposta a uma invasão viral ou bacteriana esta baseada na interação proteína-ligante. Leucocitos – células brancas do sangue – células do sistema imune – macrófagos e linfócitos ADP Pi + Enzimas Serino-protease: tríade catalítica Enzimas são proteínas que alteram quimicamente os ligantes que a ela se ligam. Em uma enzima o ligante é chamado de substrato. Os sítios de ligação dos substratos são chamados sítio ativo. Serino-protease: ataque da serina Serino-protease: intermediário Serino-protease: hidrólise Serino-protease: ataque da H2O Enzima GR Ciclo da Reação Catalítica da GR a GSH ES-SE E-S-S-G f Glutationa redutase (GR) H+ humanos e mamíferos em geral GSH NADPH GSSG + NADPH + H+GR → 2 GSH + NADP+ b EH2 + NADP+ enzimas homodiméricas 500 aa / monômero flavoproteínas: grupo prostético → FAD coenzima: NADPH E-S-S-G • GS- e NADP+ c EH2 EH2 • G-S-S-G d GSSG Ignez Caracelli – Departamento de Física – Faculdade de Ciências de Bauru - UNESP
Baixar