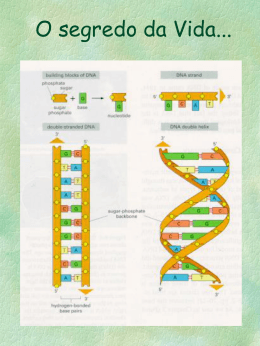
ESTRUTURA E FUNÇÃO DOS GENES E CROMOSSOMOS Abril/2005 CÉLULA HUMANA 46 cromossomos (número diplóide) (autossomos, cromossomos sexuais) 23 paternos 23 pares (número haplóide) (cromossomos homológos) 23 maternos Vários genes GENOMA HUMANO • 30.000 a 35.000 genes – a > codifica proteínas – 1400 genes/cromossomo (em média) • Número de pares de bases: 3x109 pb – <5% codificam proteínas – DNA de cópia única (45%) – DNA repetitivo (55%) TIPOS DE DNA Genoma Humano Nuclear Mitocondrial De Cópia Única (45%) Codificante (<) Repetitivo (55%) Não Codificante (>) Codificante (<) Satélite Genes Pseudogenes Introns Família Gênica Não Codificante (>) Dispersos SINEs LINEs DNA -Satélite Minissatélites Microssatélites espaçador DNA SATÉLITE* Tipo Tamanho repetições (pb) -satélite 171 Minissatélites* 14-500 Microssatélites* 2-13 *Repetições em tandem *polimórficos Número repetições milhões milhares centenas Localização centrômeros telômeros dispersos DNA DISPERSO • Elementos Intercalares Curtos (SINEs): – 90-500 pb – repetições Alu (300 pb). Constituem cerca de 10% de todo o DNA humano. • Elementos Intercalares Longos (LINEs): – até 7000 pb MOLÉCULA DE DNA •Dupla hélice •Duas fitas complementares unidas por pontes de hidrogênio •Duas fitas antiparalelas NUCLEOTÍDEO Unidade básica de repetição da fita única de DNA Desoxirribose + Fosfato + Base NUCLEOSÍDEO Desoxirribose + Fosfato COMPONENTES DA MOLÉCULA DE DNA • Açúcar: desoxirribose • Fosfato externo • Bases hidrofóbicas internas: -mantêm a estrutura da fita dupla; -formam os “degraus” da COMPONENTES DA MOLÉCULA DE DNA • Princípio do pareamento de bases complementares BASES NITROGENADAS PURINAS PIRIMIDINAS Adenina A Timina T Guanina G Citosina C Maior Menor 2 pontes de hidrogênio 3 pontes de hidrogênio: ligação mais estável! MOLÉCULA DE DNA Se o DNA de uma célula fosse esticado ele teria cerca de 2 m de comprimento! Helicoidização do DNA HISTONAS DNA NUCLEOSSOMOS 20 – 60 pb “espaçadoras” 200 pb SOLENÓIDE Cada volta contém 6 nucleossomos CROMATINA – contém até 100,000 pb ou 100 kb Cromossomo: DNA + Proteína REPLICAÇÃO REPLICAÇÃO • FITA MOLDE , FITA COMPLEMENTAR (semi-conservativa) • SEMI-DESCONTÍNUA (fragmentos de Okazaki) • ENZIMAS - DNA POLIMERASE, helicases, topoisomerases, primases, ligases, SSBP REPLICAÇÃO DNA POLIMERASE • Adiciona nucleotídeos complementares ao molde • Revisão/ edição de erro • A nova fita é sintetizada no sentido 5’ 3’ (adiciona nucleotídeos à extremidade 3’ do novo filamento) Origem de Replicação O processo de replicação ocorre em vários pontos simultâneamente Forquilha de replicação Bolha de replicação helicase topoisomerase Relaxa o superenrrolamento Fragmentos de Okasaki Quebra de pontes de hidrgênio síntese descontínua Fita lagging Fita leading síntese contínua TRANSCRIÇÃO Transcrição • RNA: • açúcar: ribose • base: uracil substitui timina (pareia-se com a adenina) • filamento único • tipos: » RNA mensageiro (RNAm) » RNA transportador (RNAt) » RNA ribossomal (RNAr) Transcrição • Cópia em RNA de uma das fitas de DNA - fita codificante ou com sentido. • A síntese é feita por complementaridade a partir da outra fita de DNA - fita molde ou anti-sentido. • RNA polimerase II Transcrição • Embora qualquer um dos filamentos de DNA possa, em princípio, servir como molde para a síntese do RNAm, apenas um deles é escolhido para isso, em uma dada região do cromossomo. A RNA polimerase liga-se ao sítio PROMOTOR do DNA, separando os filamentos RNA polimerase Local de terminação A RNA polimerase move-se no molde de DNA, e vai produzindo um transcrito no sentido 5´-3´. Nucleotídeos são acrescentados no ponta 3´do RNA FIM: quando encontra a seqüência terminal Está formado um RNAm (transcrito primário) Processamento • Promotor: – TATA box: TATAAA ou variante; posição -25 do sítio de início de transcrição. Genes expressos em um estágio específico do ciclo celular ou em um tipo celular específico. – GC box: GGGCGG ou variante. Genes housekeeping. – CAAT box: -80. – Ausência de citosinas metiladas. MODIFICAÇÕES PÓS-TRANSCRICIONAIS • Impedem a degradação do RNA • Adição 7-metilguanosina (5’ CAP) • Adição cauda poliA - sítio de poliadenilação CAP 5´ Clivagem ocorre 15-30 nucleotídeos após CAUDA POLI( A) TRANSCRITO PRIMÁRIO SPLICE (recomposição gênica) - retirada dos introns ATG AG G/A GT EXON 1 AG AG GT EXON 2 G/A AG EXON 3 Transcrito final -Um gene pode ter mais de um promotor (ex: gene da distrofina) -Alguns genes possuem sítios alternativos de corte -Alguns íntrons contêm genes transcritos que aparentemente não são relacionados ao gene no qual estão contidos (intron 26 da NF-1) TRADUÇÃO TRADUÇÃO • Síntese protéica • Envolve o pareamento de bases do RNAm (códon) com o RNAt (anticódon) que traz os AA • Ocorre nos ribossomos TRADUÇÃO Para transformar o RNAm maduro em uma proteína é necessário um CÓDIGO GENÉTICO irá definir quais aminoácidos irão compor o nosso polipeptídeo Código Genético • Nucleotídeos (A,G,C,U) aminoácidos • Cada três nucleotídeos codificam um AA códon (UNIVERSAL) – ex.: AUG - Metionina • 64 códons (4x4x4) para 20 AA e mais de um códon para cada AA (DEGENERADO) • Três códons de terminação fim da proteína: UAA, UGA,UAG Código Genético RNA TRANSPORTADOR Lisina Pareia com o códon – seqüência complementar do RNAm Ribossomo A medida que cada códon é processado, 1 AA é traduzido mRNA Proteínas enzimáticas + RNAr auxiliam na ligação RNAm e RNAt Quando termina a tradução? •Quando o ribossomo chega a um códon finalizador na seqüência de RNAm forma-se um polipeptídeo: NH2 (5’ RNAm) COOH (3’ RNAm) •Modificações pós-traducionais REGULAÇÃO GÊNICA Receptores Neurotransmissores Receptores Actina Miosina Mesmos genes Diferenças na expressão Controle Alguns genes (RAROS) são transcritos em todas as células do corpo genes de manutenção Housekeeping genes importantes para o metabolismo e funcionamento celular. Apesar de todas as células possuírem exatamente a mesma seqüência de DNA, algumas proteínas serão somente sintetizadas em determinados tecidos. EX: receptores de LDL são transcritos somente nos hepatócitos. Controle da Expressão Gênica Interação de mais de 50 proteínas diferentes Enhancer – “acentuadores” – e silenciadores específicos: seqüências localizadas antes ou depois dos genes FATORES DE TRANSCRIÇÃO: Fatores Gerais – ligam-se à RNA polimerase e seqüências específicas de DNA na região do promotor (TATA) Fatores Específicos – ativadores, co-ativadores. Os fatores de transcrição contêm motivos de ligação ao DNA permitem a interação com seqüências específicas do DNA exposição de regiões ativadoras ou silenciadoras. Exemplos: •hélice-giro-hélice •anel de zinco •zíper de leucina • Cromatina transcricionalmente inativa – – – – hetecromatina (não é rica em genes) condensada replicação tardia na fase S ligação forte com histona H1 • Cromatina transcricionalmente ativa – – – – – eucromatina (rica em genes) + aberta replicação precoce na fase S ligação mais fraca com histona H1 acetilação extensa das histonas H2A, H2B, H3, H4
Download