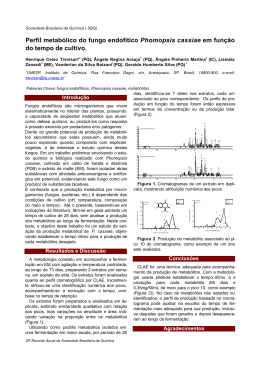
Documentos ISSN 0102-0110 Setembro, 2006 METABOLÔMICA: aplicações e perspectivas 189 ISSN 0102 0110 Setembro, 2006 Recursos Genéticos e Biotecnologia Documentos 189 METABOLÔMICA: aplicações e perspectivas Thales Lima Rocha Raphael Garcia de Sousa Evaristo Luciano Paulino da Silva Djair dos Santos de Lima e Souza Brener Magnabosco Marra Paulo Henrique Alves da Costa José Cesamildo Cruz Magalhães Maria Cristina da Silva Mattar Maria Fátima Grossi-de-Sá Brasília, DF 2006 Exemplares desta edição podem ser adquiridos na Embrapa Recursos Genéticos e Biotecnologia Serviço de Atendimento ao Cidadão Parque Estação Biológica, Av. W/5 Norte (Final) – Brasília, DF CEP 70770-900 – Caixa Postal 02372 PABX: (61) 3348-4739 Fax: (61) 3340-3666 www.cenargen.embrapa.br e.mail:[email protected] Comitê de Publicações Presidente: Sergio Mauro Folle Secretário-Executivo: Maria da Graça Simões Pires Negrão Membros: Arthur da Silva Mariante Maria de Fátima Batista Maurício Machain Franco Regina Maria Dechechi Carneiro Sueli Correa Marques de Mello Vera Tavares de Campos Carneiro Supervisor editorial: Maria da Graça S. P. Negrão Normalização Bibliográfica: Maria Iara Pereira Machado Editoração eletrônica: Maria da Graça S. P. Negrão Fabrício Lopes dos Reis Rodrigues 1ª edição 1ª impressão (2006): M 587 Metabolômica: aplicações e perspectivas. / Thales Lima Rocha ... [et. al.]. – Brasília: Embrapa Recursos Genéticos e Biotecnologia, 2006. 38 p. (Documentos / Embrapa Recursos Genéticos e Biotecnologia, 0102 – 0110; 189) 1. Metabolômica. 2. Ômicas - plataformas tecnológicas. I. Rocha, Thales Lima. II. Série. 620.82 – CDD 21. Autores Thales Lima Rocha Raphael Garcia de Sousa Evaristo Luciano Paulino da Silva Djair dos Santos de Lima e Souza Brener Magnabosco Marra Paulo Henrique Alves da Costa José Cesamildo Cruz Magalhães Maria Cristina da Silva Mattar Maria Fátima Grossi-de-Sá 4 Sumário As Ômicas ................................................................................................................. 6 A Genômica............................................................................................................... 6 A Transcriptômica .................................................................................................... 9 A Proteômica .......................................................................................................... 10 A Metabolômica ...................................................................................................... 11 Os metabólitos........................................................................................................ 13 Metaboloma, Metabolômica, “Metabolic profiling” e Engenharia de metabolismo......................................................................................................... 15 Seqüência de etapas da metabolômica ................................................................ 16 Aplicabilidade da Metabolômica ........................................................................... 21 Farmacologia .......................................................................................................... 22 Nutrição ................................................................................................................... 24 Ecologia e Evolução............................................................................................... 25 Perspectivas para a metabolômica....................................................................... 27 Considerações Finais ............................................................................................ 28 Referências Bibliográficas..................................................................................... 31 5 METABOLÔMICA – Aplicações e Perspectivas Thales Lima Rocha Raphael Garcia de Sousa Evaristo Luciano Paulino da Silva Djair dos Santos de Lima e Souza Brener Magnabosco Marra Paulo Henrique Alves da Costa Maria Fátima Grossi-de-Sá As Ômicas As plataformas tecnológicas conhecidas como “ômicas” são aquelas que têm o objetivo de isolar e caracterizar o maior número possível de biomoléculas de um mesmo grupo, como Ácidos Desoxirribonucléicos (DNA), Ácidos Ribonucléicos (RNA), proteínas ou metabólitos. A primeira ômica a ser denominada como tal foi a genômica. Esse termo vem da palavra Genoma, que por sua vez deriva da junção das palavras Gene e Cromossoma. Graças a grande aceitação desses termos, outros como transcriptoma, proteoma e metaboloma (assim como suas respectivas ômicas) foram também surgindo. Entretanto as suas origens etimológicas são ainda mais confusas (ROBERTSON, 2005). Nos últimos anos têm surgido várias ômicas. Entretanto, aquelas mais conhecidas são: a genômica, a transcriptômica, a proteômica e a metabolômica. Elas têm como objetivo isolar e caracterizar, respectivamente, o DNA, o RNA, as proteínas e os metabólitos (BINNECK, 2004). A integração das informações obtidas por essas plataformas citadas acima são o objetivo principal da Biologia de Sistemas, também conhecida como Genômica Funcional (ROCHFORT, 2005). A Genômica Nos últimos anos, é crescente o número de seqüências gênicas conhecidas, assim como a quantidade de dados disponíveis para acesso público (BINNECK, 2004). Isso certamente está relacionado à alta estabilidade das moléculas de DNA, quando comparadas às moléculas de RNA ou proteínas (PODGORNICK, 2004). Outro fator que contribuiu bastante para esse aumento foi o grande investimento na área da genômica, sobretudo na última década (BINNECK, 2004). 6 Além do crescimento do número de dados, ocorre também um maior acesso aos mesmos. A grande facilidade em se explorar os dados de seqüências reside nas ferramentas de bioinformática. Essas ferramentas agrupam uma grande quantidade de informação e permitem a busca de seqüências similares no banco de dados, ou mesmo a comparação de dados de diferentes genomas (BINNECK, 2004). Entretanto, tem-se criado uma expectativa excessiva quanto à aplicação dos dados obtidos de seqüências gênicas no que tange a inferências biológicas (BINNECK, 2004). Conhecer essas seqüências não garante que a proteína codificada seja facilmente determinada (HÜTTENHOFER e VOGEL, 2006). As predições computacionais efetuadas a partir de dados de seqüências são complexas e nem sempre garantem resultados aceitáveis, principalmente quando se trata de genomas complexos, como o humano. A razão dessa incerteza reside no fato de que as predições são derivadas de diferentes métodos (BINNECK, 2004). Alguns programas determinam genes pela identificação de parâmetros que definem o início e o fim de um gene (predição “ab inittio”). Já outros programas buscam genes pela comparação de fragmentos de seqüências com homologia a genes conhecidos (predição comparativa). Dessa forma, a predição ab inittio tende a superestimar o número de genes por identificar qualquer fragmento de seqüência que pareça um gene. Já a predição comparativa tende a subestimar o número de genes porque só identifica os genes homólogos aos já conhecidos. Portanto, com as informações obtidas até hoje, apenas os métodos computacionais não são suficientes para gerar o número real de genes de um genoma eucariótico complexo (BINNECK, 2004). Se não bastasse essa incerteza no número total de genes em um genoma específico, a identificação de sua função também não é fácil (HÜTTENHOFER e VOGEL, 2006). O tamanho do genoma por si só não define a complexidade de um organismo. A tabela 1 mostra que organismos fenotipicamente bem diferentes, como o nematóide Caenorhabditis elegans e a planta Arabidopsis thaliana, possuem genomas de tamanho próximo. Além disso, só pela seqüência de nucleotídeos apresentada por gene não é possível inferir com certeza a proteína que ele codifica (RIGOUTSOS et al., 2006). Isso é devido à ocorrência de várias modificações pós transcricionais e pós traducionais (ZANCAN, 2002; BINNECK, 2004). 7 Tabela 1: Tamanho do Genoma e número estimado de genes de vários organismos Tamanho do Genoma Número estimado de Organismo (pares de base) Genes Homo sapiens 3 bilhões 30.000 Mus musculus 2,6 bilhões 30.000 Arabidopsis thaliana 100 milhões 25.000 Caenorhabditis elegans 97 milhões 19.000 Drosophila melanogaster 137 milhões 13.000 12,1 milhões 6.000 Escherichia coli 4,6 milhões 3.200 Vírus da AIDS (HIV) 9700 9 Saccharomyces cerevisiae (Adaptado de BINNECK, 2004). Sendo assim, a explicação para tantas diferenças entre os fenótipos das espécies em questão é depositada nos mecanismos de modificações pós transcricionais como o “splicing” alternativo, nas inúmeras transformações pós traducionais que ocorrem com as diferentes proteínas, na produção e utilização dos inúmeros metabólitos como cofatores enzimáticos e nos mecanismos que regulam esses processos (ZANCAN, 2002; FIEHN, 2002; RIGOUTSOS et al., 2006). O “splicing” alternativo consiste em produzir um RNA mensageiro (RNAm) diferente do esperado a partir de um RNA nuclear heterogêneo (RNAnh) comum. Para tanto, pode ocorrer de se eliminar um dos éxons ou mesmo manter um dos íntrons. É dessa forma que a D. melanogaster produz suas três diferentes cadeias pesadas de miosina, partindo de apenas um RNAnh (MACHUCA-TZILI et al., 2006). As modificações pós traducionais são alterações na seqüência de aminoácidos recém traduzida (clivagens proteolíticas) ou a inserção de grupamentos acetil, fosforil, metil, entre outros, além da adição de grupos prostéticos, como por exemplo, o grupo Heme na hemoglobina (MÄDER et al., 2002; ZANCAN, 2002). 8 Com o intuito de se relacionar as informações obtidas pela genômica com o fenótipo apresentado pelo indivíduo em questão, outras tecnologias estão se desenvolvendo e ganhando destaque no cenário científico. Dentre essas tecnologias, ganham destaque a transcriptômica, a proteômica e, ultimamente, a metabolômica (MÄDER et al., 2002; FRANK e HARGREAVES, 2003; VIVANCO et al., 2003). A Transcriptômica Transcriptoma é o termo dado ao produto inicial da expressão gênica de um organismo. Em outras palavras, é o conjunto de RNAs mensageiros (RNAm) que codificam as informações que a célula precisa em um determinado momento (NAKANISHI e NUREKI, 2005). Qualquer célula recebe seu transcriptoma da célula mãe que lhe deu origem por divisão celular. Logo, a transcrição de seus genes não resulta na síntese de seu transcriptoma, mas sim na manutenção do mesmo e na mudança de sua composição à medida que genes específicos são transcritos ou não (BINNECK, 2004). O estudo de RNAm tem se beneficiado muito com as tecnologias baseadas na Reação de Cadeia de Polimerase (PCR). Dessa forma, vários genes podem ser estudados paralelamente, ao invés de um só de cada vez. As técnicas “Differential Display” (DD), “Serial Analylis of Gene Expression” (SAGE) e microarranjo de DNA apresentam muitas vantagens quando comparadas ao “Northern Blotting”, tanto em sensibilidade quanto em número de ensaios (BINNECK, 2004). Dentre essas técnicas, a que vem sendo mais utilizada tanto no estudo da composição de transcriptomas quanto na comparação entre transcriptomas diferentes é o microarranjo de DNA (FRANK e HARGREAVES, 2003; NAKANISHI e NUREKI, 2005; RIGOUTSOS et al., 2006). O microarranjo de DNA consiste em uma coleção de vários fragmentos pequenos de genes que são dispostos em locais específicos de uma lâmina de vidro especial de microscopia ou uma lâmina de sílica. A técnica se alicerça da habilidade das moléculas de RNAm de se ligarem especificamente (hibridizar) ao DNA complementar do qual foram transcritas. Utilizando uma placa com milhares de fragmentos de DNA, é possível saber, em um único experimento, o nível de 9 expressão de milhares de genes em uma célula com base na quantidade de RNA hibridizados em cada sítio (BINNECK, 2004). A quantificação dos RNA de cada sítio é efetuada com o auxílio de programas computacionais (por exemplo, ArrayVision software), gerando um perfil de expressão gênica da célula (MÄDER et al., 2002). Apesar de informarem que genes estão sendo expressos em maior quantidade, os dados obtidos pela quantificação do RNAm de uma célula nem sempre se correlacionam com a expressão das proteínas da mesma (CALSA JUNIOR et al., 2004). Isso porque os RNA menos abundantes, como aqueles que codificam proteínas responsáveis pela maturação de leucócitos são mais difíceis de serem quantificados do que aqueles RNAm mais abundantes (PATTON et al., 2006). Informações mais precisas sobre o nível de expressão protéica de um organismo são obtidas pela utilização da tecnologia proteômica (BINNECK, 2004). A Proteômica O proteoma de um organismo é o conjunto de proteínas expressas por um organismo ou tecido em um determinado momento e sob uma condição específica (JAMES, 1997; ROCHA et al., 2005). O número de proteínas produzidas por um organismo excede e muito o número de genes que o mesmo possui. Isso novamente se explica pelas inúmeras modificações pós transcricionais e pós traducionais (BINNECK, 2004). O conjunto de tecnologias criadas para analisar a expressão de proteínas é conhecido como proteômica (ROCHA et al., 2005; VIVANCO et al., 2003). A proteômica vem se desenvolvendo com o intuito de implementar novas estratégias para a análise de proteínas em escala genômica. Entretanto, a análise das proteínas de um organismo depende da extração, purificação, visualização, identificação e quantificação dessas, e em cada uma das etapas há uma série de limitações. Dessa forma, para se conhecer o proteoma completo de um organismo, se faz necessária a utilização de diversas estratégias, como: a utilização de técnicas como cromatografias associadas à eletroforese, o pré-fracionamento das amostras por meio de extrações seqüenciais, a análise do proteoma organelar, entre outras. (ROCHA et al., 2005). Atualmente, as investigações proteômicas se iniciam principalmente com a eletroforese bidimensional (2DE). Essa técnica permite separar as proteínas de uma 10 amostra pelos seus respectivos pontos isoelétricos e pesos moleculares (O’FARREL, 1975; KLOSE, 1975). As proteínas separadas por essa técnica são, geralmente, digeridas por proteases e analisadas em espectrômetros de massa. Os perfis obtidos pelo espectrômetro de massa, contendo todos os fragmentos resultantes da digestão enzimática que foram detectados pelo equipamento, são denominados “fingerprinting” (MÄDER et al., 2002). Dependendo da qualidade da amostra analisada, pode-se chegar ao seqüenciamento parcial ou mesmo total da proteína em questão. A partir da comparação das seqüências obtidas em um banco de dados, pode-se identificar a proteína isolada (BINNECK, 2004). A eletroforese bidimensional apresenta as mesmas limitações observadas nas técnicas transcriptômicas acima descritas. As proteínas expressas em baixa quantidade, ou em um curto período, acabam por não serem, às vezes, sequer visualizadas (MÄDER et al., 2002). Essas proteínas podem estar sendo expressas para iniciar a resposta a algum estímulo importante, como aquelas relacionadas à cascata de ativação da proteína Kinase C, que tem um importante papel na prevenção da isquemia no miocárdio (VIVANCO et al., 2003; BINNECK, 2004). A Metabolômica Não há dúvidas de que conhecer as proteínas de um organismo é importante. Mas existe um universo de pequenas moléculas orgânicas que interagem diretamente com as proteínas e outras macromoléculas (HALL, 2006). Essas moléculas atuam como substratos, inibidores ou ativadores alostéricos de uma enzima. Podem ser precursores de alguma molécula importante nas inúmeras rotas metabólicas celulares, ou até mesmo um resíduo metabólico de alguma via de síntese ou degradação de macromoléculas que por algum motivo é estocado e utilizado como defesa química contra algum patógeno (ENGEL et al., 2002; DEMAIN e VAISHNAV, 2006). Essas pequenas, porém, importantes moléculas são conhecidas como metabólitos (DIXON, 2001). Esses metabólitos são divididos em dois grandes grupos: os metabólitos primários e os secundários. Os primários são aqueles que estão diretamente envolvidos nas rotas de síntese e degradação das macromoléculas em qualquer ser vivo (DIXON, 2001). Já os secundários são mais comuns em plantas e fungos (CHALLIS e HOPWOOD, 2003; HALL, 2006). Eles atuam como componentes 11 estruturais, defesas contra herbívoros e patógenos, atrativos para polinizadores e agentes nas interações interespecíficas, como a alelopatia (ENGEL et al., 2002; CHALLIS e HOPWOOD, 2003). Segundo Challis e Hopwood (2003), o estudo desses metabólitos visando à obtenção de produtos efetivos contra diversas doenças e passíveis de comercialização já vem sendo feito desde o início do século XX. Entretanto, a idéia de se analisar todo o conjunto de metabólitos em qualquer nível de complexidade (organismo, órgão, tecido, célula ou mesmo compartimentos celulares) só começou a ganhar destaque nos últimos anos, conforme pode ser observado na figura 1 (HORNING e HORNING, 1971; NICHOLSON et al., 1999; FIEHN, 2002; REO, 2002; LINDON et al., 2003, 2004a, b; NICHOLSON e WILSON, 2003; WECKWERTH, 2003; BINO et al., 2004; FERNIE et al., 2004; GOODACRE et al., 2004; FUKUSAKI e KOBAYASHI, 2005; MUNGUR et al., 2005; ROBERTSON, 2005; ROCHFORT, 2005; WECKWERTH e MORGENTHAL, 2005). O nome dado a todas as tecnologias envolvidas na análise desses metabólitos foi metabolômica. 250 Nº de artigos publicados 200 150 100 50 0 1975 - 1994 1995 - 1999 2000 2001 2002 2003 2004 2005* Ano Figura 1: Número de artigos relacionados à metabolômica publicados desde 1975 até Setembro de 2005. Nota-se um grande aumento nas publicações sobre esse tema nos últimos cinco anos. (Adaptado de ROCHFORT, 2005). 12 Nos últimos anos, a metabolômica tem sido empregada em diversas áreas e com inúmeros propósitos. Todavia, em qualquer âmbito existem várias dificuldades a serem superadas tanto na extração e separação das amostras quanto na análise dos resultados (ROCHFORT, 2005; WECKWERTH e MORGENTHAL, 2005; GIBNEY et al., 2005; FUKUSAKI e KOBAYASHI, 2005; ROBERTSON, 2005; CHALLIS e HOPWOOD, 2003). Contudo, é de senso comum que os resultados obtidos pela metabolômica facilitarão o entendimento de como o genótipo de um indivíduo está relacionado como o seu fenótipo (FUKUSAKI e KOBAYASHI, 2005; BINNECK, 2004). O objetivo principal desse estudo é mostrar a metabolômica hoje em dia. Isso inclui todos os avanços já realizados, assim como as dificuldades ainda enfrentadas. As informações abordadas aqui poderão ser utilizadas em uma possível execução de projetos de pesquisa envolvendo metabolômica e engenharia de metabolismo na Embrapa Recursos Genéticos e Biotecnologia. Como objetivos específicos, podem ser citados: - Esclarecer os conceitos básicos da metabolômica; - Citar todas as etapas básicas para a análise de uma amostra, enfatizando as dificuldades existentes; - Apontar as inovações tecnológicas geradas ou aplicadas na metabolômica; - Discutir a aplicabilidade dessa plataforma tecnológica em várias áreas de conhecimento, como a farmacologia, a nutrição, a ecologia e a evolução; - Comentar sobre as perspectivas dessa plataforma, ressaltando as principais dificuldades e apontando os benefícios da interação com outras plataformas. Os metabólitos: Encontram-se dissolvidas no citosol de qualquer célula milhares de pequenas moléculas conhecidas como metabólitos primários. Essas moléculas estão envolvidas nas principais rotas metabólicas que ocorrem nas células (DIXON, 2001). Dentre esses metabólitos estão: os aminoácidos, nucleotídeos, lipídeos, carboidratos e seus derivados fosforilados, além de um grande número de ácidos mono-, di- e tricarboxílicos. Esses metabólitos são altamente polares ou carregados eletricamente, solúveis em água e presentes nas células em concentrações variadas (entre milimolar e micromolar). Sua retenção dentro das células se deve ao fato de a 13 membrana plasmática ser impermeável a eles, embora existam transportadores nas membranas celulares que possibilitem a entrada e/ou saída desses compostos na célula, ou em compartimentos subcelulares (células eucarióticas). A presença bastante similar dos mesmos grupos de metabólitos em qualquer célula viva demonstra a conservação evolucionária dessas rotas metabólicas principais que surgiram nos organismos mais primitivos e permanecem até os dias de hoje (SMEDSGAARD e NIELSEN, 2005). Existe um outro grupo de pequenas biomoléculas que, ao contrário dos metabólitos primários, são específicas de certos tipos de células ou organismos. Essas moléculas são comumente chamadas de metabólitos secundários (DIXON, 2001). Os metabólitos secundários são moléculas que geralmente não estão envolvidas em funções vitais, geralmente não fazem parte do metabolismo básico e possuem características químicas muito variadas e, às vezes, bem complexas (HALL, 2006). Durante muito tempo acreditou-se que os metabólitos secundários fossem produzidos sem uma função específica, simplesmente como produtos finais das reações, chegando a ser considerados até como anomalias (MARASCHIN e VERPOORTE, 1999). Essa visão mudou radicalmente e a cada dia descobre-se um pouco mais sobre a função destas substâncias, sua utilidade para o desenvolvimento fisiológico das plantas e seu papel como mediadores das interações entre as plantas e outros organismos (MARASCHIN e VERPOORTE, 1999; NEWMAN et al., 2003; ZHANG et al., 2003). Os principais grupos de metabólitos secundários são os alcalóides, terpenos, taninos, flavonóides e glicosídeos (HALL, 2006). Os alcalóides são bases nitrogenadas com grande variação na estrutura molecular. Possuem sabor amargo e pH alcalino quando em solução. Nas plantas têm função de regulação do crescimento e de proteção. Os terpenos são moléculas de estrutura cíclica, também conhecidos como óleos essenciais. Eles são voláteis e por isso são responsáveis pelos odores dos vegetais. Podem ser classificados quanto ao número de átomos de carbono em: mono, sesqui, di e triterpenos. Nas plantas possuem funções na polinização, proteção, etc. Os taninos são compostos fenólicos (poli fenóis) de estrutura variável. Apresentam atividade adstringente, ou seja, precipitam proteínas. Nos vegetais, têm função de proteção. Os flavonóides são heterosídeos, ou seja, 14 contém uma porção açúcar (diferente da glicose) ligada a uma porção não-glicídica (aglicônio), que neste caso é um pigmento. Esses compostos possuem cores de acordo com o tipo de pigmento presente na molécula, por exemplo, as flavonas são amarelas e os flavonóides antociânicos são azuis. Nas plantas atuam na coloração dos órgãos vegetais e na diferenciação celular. Por fim, os glicosídeos são compostos que contém um açúcar ligado a um aglicônio, sendo esse a porção ativa da molécula. Possuem gosto muito amargo, e são classificados de acordo com o tipo de aglicônio. Por exemplo: glicosídeos alcoólicos (o aglicônio é um álcool); glicosídeos cianogênicos (liberam ácido cianídrico quando hidrolisados); glicosídeos esteróides (o aglicônio é um esteróide); glicosídeos antraquinônicos (o aglicônio é uma antraquinona). Nos vegetais, a função varia de acordo com a porção nãoglicídica (MARASCHIN e VERPOORTE, 1999; ZHANG et al., 2003). Metaboloma, Metabolômica, “Metabolic profiling” e Engenharia de metabolismo O conjunto de todas essas pequenas moléculas (metabólitos primários e secundários) de uma dada célula, fluido corporal ou organismo é conhecido como metaboloma (GOODACRE, 2005). Uma vez conhecida a composição do metaboloma, pode-se prever que genes e rotas metabólicas estão ativos naquele momento na célula (GOODACRE, 2005; ROBERTSON, 2005). O interesse em se conhecer melhor os metabólitos (primários ou secundários), assim como suas vias de síntese e a regulação das mesmas tem crescido muito nos últimos anos (ROCHFORT, 2005). Dessa forma, a Metabolômica, ou Metabonômica, já figura entre as grandes plataformas tecnológicas para o estudo de sistemas biológicos, como a Genômica, Transcriptômica e Proteômica (BINNECK, 2004). Os dois termos (Metabolômica e Metabonômica) estão corretos. O que os diferencia são apenas as suas origens etimológicas (ROBERTSON, 2005). Para todos os fins, nesse trabalho será utilizado somente o termo Metabolômica. A metabolômica tem como objetivo isolar e caracterizar todos os metabólitos de uma dada amostra biológica em condições normais e sob algum tipo de estresse (ROCHFORT, 2005; WECKWERTH e MORGENTHAL, 2005). Mesmo utilizando as mesmas técnicas, ela se difere da análise de perfil metabólico (Metabolic profiling), 15 que busca o isolamento de moléculas alvo e a caracterização das suas vias de síntese (WECKWERTH e MORGENTHAL, 2005). A linha que divide essas duas plataformas geralmente é muito tênue. Técnicas como a Ressonância Magnética Nuclear de biofluidos ou a Espectrometria de Massa de biofluidos são utilizadas para os dois propósitos. No entanto, o que se sabe a cerca do biofluido e o conhecimento prévio de parâmetros a serem analisados é que vai definir se a análise se enquadra na metabolômica ou no “metabolic profiling” (ROBERTSON, 2005). A metabolômica, devido ao seu objetivo, não utiliza parâmetros, mas sim os determina. Após serem determinados, o “metabolic profiling” os utiliza (ROBERTSON, 2005; WECKWERTH e MORGENTHAL, 2005). Após a descoberta dos metabólitos de uma amostra qualquer e a identificação de algum composto de interesse (farmacêutico, por exemplo), é possível iniciar os estudos de engenharia de metabolismo. Tais estudos primam por identificar os genes e as rotas metabólicas desses compostos, visando aumentar ou diminuir a sua produção. Além disso, a engenharia de metabolismo pode ser aplicada na modificação genética de um outro organismo, para que este possa produzir o metabólito em questão (AHARONI et al., 2005). Seqüência de etapas da metabolômica A metabolômica, aplicada em qualquer área da ciência, possui uma série de etapas. Em cada uma dessas etapas, podem ocorrer erros experimentais (FUKUSAKI e KOBAYASHI, 2005). A primeira etapa é a seleção do organismo, célula, tecido ou biofluido a ser analisado. Nesse ponto, o mais difícil é manter a uniformidade da amostra. A variação na idade ou tamanho destas amostras, assim como o tratamento dado ao organismo de origem (dieta, adubação, exposição à luz, etc.) ou mesmo o horário da coleta interfere substancialmente na composição da amostra (BOLLARD et al., 2001; FIEHN, 2002; FUKUSAKI e KOBAYASHI, 2005; ROBERTSON, 2005; ROCHFORT, 2005). Após a seleção do organismo, é realizado o preparo da amostra. Essa etapa é uma das mais importantes, uma vez que qualquer displicência pode interferir drasticamente no resultado da análise (FUKUSAKI e KOBAYASHI, 2005). Para se 16 evitar grandes variações experimentais, a mesma quantidade da amostra, avaliandose o peso seco do mesmo tipo de órgão deve ser coletada. E tudo isto deve ser feito no mesmo horário (ROBERTSON, 2005). O processo de extração é o ponto mais crítico. A maceração, quando necessária, deve ser realizada obedecendo a rigorosos padrões (FIEHN, 2002; FUKUSAKI e KOBAYASHI, 2005). Se a análise for feita com biofluidos, essa etapa é mais simples. No entanto, esse é um tipo de amostra muito sensível às variações citadas anteriormente, exigindo bastante cuidado dos responsáveis por essa etapa (ROBERTSON, 2005; ROCHFORT, 2005). Muitas das amostras, antes de serem analisadas, necessitam de um préfracionamento. Isso vai depender, principalmente, dos equipamentos a serem utilizados no fracionamento (FIEHN, 2002; FUKUSAKI e KOBAYASHI, 2005; ROBERTSON, 2005; ROCHFORT, 2005; WECKWERTH e MORGENTHAL, 2005;). Esse pré-fracionamento serve para agrupar os compostos presentes na amostra de acordo com suas características moleculares utilizando diferentes solventes (WANT et al., 2006). Dessa forma, pode-se definir qual ferramenta será utilizada (FUKUSAKI e KOBAYASHI, 2005; ROBERTSON, 2005; WECKWERTH e MORGENTHAL, 2005). O ponto mais crítico desse pré-fracionamento é a escolha do solvente usado para a separação (FUKUSAKI e KOBAYASHI, 2005). Deve-se prestar muita atenção nas possíveis interferências que esse solvente pode causar nos equipamentos de análise, assim como nas possíveis reações indesejadas que podem ocorrer entre o solvente e a amostra, produzindo novos compostos (ROBERTSON et al., 2000; BECKWITH-HALL et al., 2002; ROBERTSON, 2005; WANT et al., 2006). Outras considerações devem ser feitas acerca da utilização da metabolômica no que tange estudos com animais (ROBERTSON et al., 2002). Uma vez que essa tecnologia é extremamente sensível, variações fisiológicas ou ambientais que o animal pode sofrer durante o teste serão detectadas e podem mascarar os resultados. Por exemplo, uma diferença de apenas quatro semanas na idade pode resultar em uma notória discrepância no perfil metabólico de ratos (ROBERTSON et al., 2000). O fracionamento da amostra é a etapa mais importante da metabolômica (FIEHN, 2002; GERMAN et al., 2005). Embora qualquer ferramenta que permita 17 mensurar os metabólitos possa ser usada pela metabolômica, a Espectrometria de Massa (EM) acoplada a Cromatografia líquida, Cromatografia Gasosa ou Eletroforese Capilar, juntamente com a Ressonância Magnética Nuclear (RMN) dominam a literatura (NICHOLSON et al., 1999; FIEHN, 2002; REO, 2002; LINDON et al., 2003, 2004a; NICHOLSON e WILSON, 2003; WECKWERTH, 2003; BINO et al., 2004; FERNIE et al., 2004; GOODACRE et al., 2004; ROBERTSON, 2005; WECKWERTH e MORGENTHAL, 2005). No fracionamento de amostras para os estudos de metabolômica, o ideal é que se tenha uma alta resolução dos diferentes compostos presentes. Essa resolução depende, sobretudo, da ferramenta utilizada para o fracionamento (FUKUSAKI e KOBAYASHI, 2005; WILSON et al., 2005). A EM é muito mais sensível do que a RMN, muito embora isso nem sempre represente uma vantagem (ROBERTSON, 2005). Essa maior sensibilidade implica em uma menor reprodutibilidade da análise no mesmo equipamento ou em equipamentos equivalentes (FUKUSAKI e KOBAYASHI, 2005; ROCHFORT, 2005; WECKWERTH e MORGENTHAL, 2005; WILSON et al., 2005). A tabela 2 (ROBERTSON, 2005) mostra um comparativo entre essas duas ferramentas com relação à aplicabilidade das mesmas na metabolômica. Equipamentos como o “Fourier Transform Ion Ciclotron Ressonance Mass Spectrometry” (FT-ICR-MS) combinam a RMN e a EM, possibilitando a detecção de um maior número de compostos diferentes. Entretanto, esse tipo de equipamento ainda carece do desenvolvimento de algoritmos mais efetivos para analisar os resultados obtidos com ele (MUNGUR et al., 2005). Uma variação da RMN, conhecida como “Magic Angle Spinning” (MAS), permite a análise direta de tecidos, não necessitando das etapas de extração e pré-fracionamento. Entretanto essa técnica necessita de equipamentos especiais e recursos humanos altamente qualificados, acabando por limitar seu uso (STRAUS, 2004). Devido a essas peculiaridades, a maioria dos grupos de pesquisa voltados aos estudos de metabolômica estão usando tanto os equipamentos associados a EM quanto os associados a RMN (ROBERTSON, 2005). 18 Tabela 2: Comparação entre RMN e EM para a aplicação na metabolômica. RMN EM Logística Custo total sem vantagem Custos para sem vantagem utilização rotineira Manutenção vantagem Custo por amostra sem vantagem Habilidade técnica vantagem requeridaa Durabilidade sem vantagem Considerações Analíticas Sensibilidade muita vantagem Reprodutibilidade (em outros muita vantagem laboratórios) Velocidade de sem vantagem análise Capacidade sem vantagem (amostras/dia) Preparo da amostra vantagem Automatização da vantagem análise Versatilidadeb vantagem Seletividadec vantagem Metabolômica Resolução de muita vantagem estrutura molecular Identificação de moléculas vantagem desconhecidas Polarizaçãod da muita vantagem amostra Automatização da análise dos vantagem resultados a. O número de analitos qualificados por RMN é muito menor do que por EM. b. Geralmente, qualquer equipamento de RMN pode ser configurado para a maioria das aplicações, enquanto diferentes equipamentos de EM podem ser necessários para aplicações específicas. c. A EM prima pela identificação seletiva da identidade molecular, enquanto a RMN prima pela identificação de todos os átomos que contêm prótons na amostra. Entretanto, a seletividade pode ser uma vantagem ou uma desvantagem, dependendo da aplicação. d. Potencial para gerar séries de dados incompletos ou com baixa reprodutibilidade devido à polarização inerente a tecnologia (ex: supressão de íons na EM). (Adaptado de ROBERTSON, 2005). 19 Existem três requisitos básicos para a utilização de qualquer uma das ferramentas listadas. São eles: o capital de investimento para a compra dos equipamentos e reagentes necessários, uma vez que não existam equipamentos aptos ao uso; o espaço físico, já que esses equipamentos não são pequenos e necessitam de temperaturas controladas; e, principalmente, pessoas qualificadas para manipularem-nos (LINDON et al., 2004b; ROBERTSON, 2005). Como em grandes instituições de pesquisas é comum que existam os três pré-requisitos, geralmente, a disponibilidade dos equipamentos e de profissionais capacitados para operá-los é que irá determinar a ferramenta que será usada (ROBERTSON, 2005). Após todos os passos de fracionamento, se faz necessária a conversão dos valores obtidos por diferentes equipamentos para uma unidade padrão. Contudo, isso nem sempre é fácil, ainda mais se forem utilizados muitos passos para o fracionamento (FUKUSAKI e KOBAYASHI, 2005; ROBERTSON, 2005; ROCHFORT, 2005). A conversão de dados obtidos por cromatografias, eletroforeses ou mesmo RMN necessitam de ser convertidos em uma linguagem digital que possa ser utilizada em análises multivariadas utilizando ferramentas de estatística (FIEHN, 2002; DURAN et al., 2003; STEUER et al., 2003; FUKUSAKI e KOBAYASHI, 2005; MUNGUR et al., 2005; ROBERTSON, 2005; ROCHFORT, 2005; WECKWERTH e MORGENTHAL, 2005). As ferramentas de estatística são muito importantes para a metabolômica (DURAN et al., 2003; FUKUSAKI e KOBAYASHI, 2005; ROBERTSON, 2005; WECKWERTH e MORGENTHAL, 2005). Um estudo de metabolômica qualquer pode gerar milhares de espectros de RMN para serem analisados, o que seria uma tarefa no mínimo exaustiva, mesmo para um espectroscopista experiente (ROBERTSON, 2005). Dessa forma, muitas ferramentas têm sido desenvolvidas ou adaptadas de outros estudos para analisar um grande número de dados rapidamente (FUKUSAKI e KOBAYASHI, 2005; ROBERTSON, 2005; WECKWERTH e MORGENTHAL, 2005). Entretanto, assim como nos outros passos dos estudos de metabolômica, as ferramentas de análise dos dados obtidos ainda são limitadas (FIEHN, 2002; STEUER et al., 2003; FUKUSAKI e KOBAYASHI, 2005; MUNGUR et al., 2005; WECKWERTH e MORGENTHAL, 2005). 20 A escolha do tipo de análise estatística a ser utilizada nos dados obtidos pelas técnicas de fracionamento varia de acordo com a estrutura dos dados e da intenção da análise. Entretanto, a Análise de Componente Principal (ACP) é a mais utilizada na metabolômica (NICHOLLS et al., 2001; NICHOLSON et al., 2002). A ACP é uma análise muito útil para se reduzir grupos de dados muito grandes e identificar sinais importantes nesses grupos, sobretudo se o fracionamento não foi bem sucedido. A partir de uma série de cálculos conhecidos por análise dos vetores de Eigen, a ACP permite identificar os compostos mais abundantes e, a partir disto, visualizar as diferenças dentro da amostra e agrupar os compostos hierarquicamente (FUKUSAKI e KOBAYASHI, 2005). Associando-se todo esse conhecimento gerado pela metabolômica àqueles obtidos pelas outras ômicas, é possível determinar quais são as rotas metabólicas e os genes que possivelmente estejam envolvidos na produção dos compostos de maior destaque (FUKUSAKI e KOBAYASHI, 2005). Essa integração entre as diferentes plataformas tecnológicas é chamada de Biologia de Sistemas (ROCHFORT, 2005). A figura 2 mostra um esquema típico dos passos a serem seguidos em análises metabolômicas utilizando plantas, da seleção da amostra até a determinação dos genes envolvidos na produção dos compostos mais relevantes (FUKUSAKI e KOBAYASHI, 2005). Aplicabilidade da Metabolômica As informações obtidas pela metabolômica, assim como aquelas obtidas pelo “metabolic profiling”, têm sido freqüentemente usadas em pesquisas de diversas áreas. Exemplos específicos para indicar o alcance dessa tecnologia incluem a correlação entre perfis metabólicos dos biofluidos e doenças em animais, na influência da dieta no padrão metabólico do indivíduo, na investigação dos efeitos causados pela inserção de um gene em um organismo transgênico, entre outras (BRINDLE et al., 2003; FUKUSAKI e KOBAYASHI, 2005; GIBNEY et al., 2005; ROBERTSON, 2005; ROCHFORT, 2005; WECKWERTH e MORGENTHAL, 2005). 21 Escolha do organismo Fracionamento Conversão de Preparo da amostra Préfracionamento Análise estatística Identificação dos genes e rotas metabólicas envolvidos na produção de metabólitos específicos Figura 2: Esquema típico de análises metabolômicas utilizando plantas. 1- escolha do organismo a ser estudado; 2- preparo da amostra; 3- pré-fracionamento; 4- fracionamento; 5- conversão dos resultados para uma unidade comum; 6- análise estatística dos dados; 7elucidação funcional de genes. (Adaptado de FUKUSAKI e KOBAYASHI, 2005). Farmacologia A metabolômica tem uma gama de aplicações na área farmacêutica (figura 3), tendo grande destaque as pesquisas para seleção de fármacos efetivos e seguros, a identificação de marcadores biológicos de toxicidade e doenças e o entendimento dos mecanismos de toxicidade (ROBERTSON, 2005). Na seleção de fármacos efetivos e seguros, tem-se utilizado bastante os ensaios “in vitro”. Isso porque este tipo de ensaio é mais rápido e mais fácil de ser executado. Entretanto, por mais que se use no ensaio células do mesmo tecido onde o fármaco irá atuar, essas linhagens celulares cultivadas não respondem da mesma maneira do que as células que estão no organismo. Com isso, muitos dos fármacos bem sucedidos nos ensaios in vitro acabam por fracassar nos ensaios in vivo por provocarem efeitos colaterais indesejados, não observados anteriormente (ROBERTSON, 2005). 22 Seleção Ensaios de toxicidade in vitro e in vivo Síntese Industrialização Busca de marcadores de toxicidade Mecanismos de toxicidade Figura 3: Esquema padrão de desenvolvimento de fármacos indicando as principais etapas seguidas. Em cada etapa estão indicados os locais onde a metabolômica pode ser aplicada. (Adaptado de ROBERTSON, 2005). Os ensaios “in vivo” certamente trazem resultados mais concisos quanto à eficiência e à segurança dos fármacos. Entretanto, a sua utilização como método primário para a identificação de fármacos promissores ainda é uma esperança para um futuro próximo (ROBERTSON, 2005). Seguindo essa idéia, espera-se utilizar a metabolômica como uma técnica padrão para diferenciar os efeitos colaterais causados pelo fármaco daqueles causados por outros fatores. Outra vantagem reside no fato de que a metabolômica permite a detecção de problemas causados por fármacos de uma forma não invasiva – através da utilização de biofluidos como a urina e sangue – facilitando a sua aceitação (ROBERTSON et al., 2002). A identificação de marcadores biológicos de toxicidade utilizando-se a metabolômica tem mostrado bons resultados (ROBERTSON, 2005; ROCHFORT, 2005). Essa aplicação teve seu início com a busca de marcadores biológicos para doenças renais e hepáticas (ROBERTSON et al., 2000; NICHOLLS et al., 2001). À medida que esses estudos foram avançando, foi constatado que uma série de compostos sempre variavam, não importando a natureza do composto tóxico, seu mecanismo de ação, ou mesmo seu alvo (ROBERTSON, 2005). Entretanto, a busca por marcadores biológicos utilizando a metabolômica vai além desses compostos comuns. A partir de análises comparativas foram detectados uma série de compostos que estão relacionados à toxicidade renal e nervosa (ROBERTSON, 2005; ROCHFORT, 2005). Certamente, a maior parte do esforço em prol da utilização da metabolômica para esse fim é realizada pelo COMET (Consórcio de Metabolômica aplicada a Toxicologia). Esse consórcio visa montar um banco de 23 dados com milhares de espectros de RMN utilizando biofluidos de animais tratados com toxinas modelo (ROBERTSON, 2005). Esse grupo é formado pela Imperial College de Londres e mais seis companhias farmacêuticas que investem milhões de dólares na seleção e produção de fármacos (ROCHFORT, 2005). A outra grande aplicação da metabolômica dentro da farmacologia, senão a maior, é no entendimento dos mecanismos de toxicidade, uma vez que compreendendo esse mecanismo, os marcadores biológicos serão identificados (ROBERTSON, 2005). Todavia, para a identificação de um marcador biológico, não é necessário conhecer o mecanismo de toxicidade do fármaco. A maioria dos trabalhos de metabolômica relacionados aos mecanismos de toxicidade tem comparado os perfis obtidos pelas análises de biofluidos de animais submetidos à utilização da toxina estudada aos perfis obtidos de animais não submetidos, observando os efeitos provocados por essa toxicidade (HOLMES et al., 1998; ROBERTSON et al., 2000; NICHOLSON et al., 2002; LENZ et al., 2003). Nutrição Assim como na farmácia, a metabolômica tem varias aplicações na área nutricional. Como a dieta influencia diretamente no desenvolvimento do indivíduo, a metabolômica possibilita conhecer os arranjos e desarranjos metabólicos causados pela alimentação (GERMAN et al., 2005). Dessa forma, o papel que os componentes alimentares desempenham na saúde e na doença, assim como os efeitos causados pela falta ou excesso de determinados nutrientes podem muito bem ser conhecidos pela metabolômica (WATKINS et al., 2001; GERMAN et al., 2003). Nos alimentos, o número de compostos que não são nutrientes é muito maior do que o número daqueles que são. Muitos desses compostos não nutrientes são aqueles já descritos acima, os chamados metabólitos secundários (GIBNEY et al., 2005). Além dos compostos advindos dos alimentos, deve-se considerar ainda àqueles produzidos pela microbiota intestinal e àqueles produzidos ou adicionados aos alimentos durante o cultivo ou o preparo (GERMAN et al., 2005; GIBNEY et al., 2005). Essas moléculas possuem diversas atividades biológicas e, sem dúvida, interagem de diversas formas dentro do organismo, alterando o metaboloma desse indivíduo (ROCHFORT, 2005). Exemplos simples disso são: a detecção de vários metabólitos oriundos do café em alta concentração na urina seis horas após a 24 ingestão; o aparecimento de vinte e três tipos de quercitinas (flavonóides) no sangue após a ingestão de cebolas fritas, sendo que dezoito delas já são detectadas em menos de quatro horas após a ingestão; entre outros (AHARONI et al., 2005; GIBNEY et al., 2005). Muitas dessas moléculas possuem efeitos positivos ou negativos sobre algumas doenças importantes, como o câncer (GIBNEY et al., 2005). Como exemplos disso, podem ser citados: o álcool de perila, um monoterpeno extraído do morango (Fragaria sp.), que tem atividade anticarcinogênica e alguns análogos do estrógeno encontrados em leguminosas que podem interferir no tratamento do cancer de mama (CASSIDY, 2003; AHARONI et al., 2005). A figura 4 mostra a distribuição dos compostos oriundos dos alimentos em nutrientes e não-nutrientes e suas respectivas subdivisões. De posse dessas informações acerca de como o organismo reage, na esfera metabólica, aos diferentes compostos adquiridos pelo consumo dos alimentos, em um futuro próximo será possível determinar a quantidade correta a ser ingerida de cada alimento, sem incorrer em problemas causados pela falta ou excesso dos diversos componentes que esses alimentos possuem (ASZTALOS et al., 2000; PARKS e HELLERSTEIN, 2000; GERMAN et al., 2005). Alimento Nutrientes Macro-nutrientes Não-nutrientes Micro-nutrientes Contaminantes Insumos agrícolas Fitoquímicos Defensivos Figura 4: Esquema da divisão dos compostos oriundos dos alimentos em nutrientes e nãonutrientes, assim como suas respectivas subdivisões. (Adaptado de GIBNEY et al., 2005). Ecologia e Evolução Outro campo de notória aplicabilidade da metabolômica é a ecologia (ROBERTSON, 2005; ROCHFORT, 2005). A utilização dessa plataforma nos estudos com vegetais tem sido constante nos últimos anos (FIEHN et al., 2000; 25 ROBERTS, 2000; FIEHN, 2002; VIANT et al., 2003; WECHWERTH, 2003; FUKUSAKI e KOBAYASHI, 2005; RISCHER e OKSMAN-CALDENTEY, 2006). A realização desses estudos visa entender, sobretudo, o mecanismo de ação de compostos bioativos e a diferenciação de variedades selvagens daquelas modificadas geneticamente (OTT et al., 2003; FUKUSAKI e KOBAYASHI, 2005; MUNGUR et al., 2005; RISCHER e OKSMAN-CALDENTEY, 2006). A metabolômica permite identificar os efeitos de compostos bioativos, como os herbicidas, em plantas de interesse agronômico (ARANIBAR et al., 2001; OTT et al., 2003). A partir de espectros de RMN realizados com extratos obtidos de exemplares de milho (Zea mays) tratados com diversos compostos bioativos, como o Glyphosate, entre outros, demonstraram a interferência desses compostos em processos biológicos importantes, como a mitose, a fosforilação oxidativa e o transporte de auxina (OTT et al., 2003). A análise de organismos geneticamente modificados (OGM) pela metabolômica tem sido extensamente utilizada (MUNGUR et al., 2005; KRISHNAN et al., 2005; RISCHER e OKSMAN-CALDENTEY, 2006). Esse tipo de estudo prima por detectar as diferenças imprevistas existentes entre a cultivar selvagem e a transgênica. Essas diferenças imprevistas são explicadas pelas interações randômicas ou desconhecidas que ocorrem entre os genes, assim como a participação dos metabólitos em diversas rotas metabólicas (RISCHER e OKSMANCALDENTEY, 2006). A inserção de genes pode provocar um número elevado de modificações no perfil metabólico de uma planta. De acordo com Mungur et al. (2005), a inserção do gene oriundo da bactéria Escherichia coli que codifica a enzima Glutamato desidrogenase em plantas de tabaco (Nicotiana tabacum) alterou o perfil metabólico de duzentos e oitenta e três metabólitos nas raízes e noventa e oito nas folhas da planta. Nesse estudo foi utilizada a ferramenta de fracionamento FT-ICR-MS. Mesmo com a confirmação dos metabólitos correspondentes a apenas 42% dos íons detectados, foi comprovado o aumento da concentração de: alguns aminoácidos, como a arginina, fenilalanina, triptofano, asparagina, glutamina e histidina; alguns açúcares e derivados, sobretudo alguns intermediários na regeneração da ribulose-5-fosfato no ciclo de Calvin; alguns metabólitos secundários e compostos nitrogenados, como fenóis, alcalóides e aminas; entre outros. Além 26 disso, foi detectada a presença de cerca de trinta e dois compostos de importância farmacêutica, entre eles nove compostos carcinogênicos e quatorze fármacos em potencial (MUNGUR et al., 2005). Outra aplicação da metabolômica na botânica reside no entendimento da relação planta-patógeno. Ao serem parasitadas por insetos ou nematóides, por exemplo, as plantas produzem e liberam uma série de compostos como fenóis, terpenos ou alcalóides. Esse aumento de produção é induzido pelo contato com sinalizadores químicos oriundos dos patógenos. Contudo, apesar de todas as plantas reagirem ao parasitismo, nem todas produzem os metabólitos necessários para o controle do patógeno. Dessa forma, a metabolômica pode ser aplicada na identificação dos metabólitos diferenciais entre variedades resistentes e susceptíveis a uma determinada praga. Esse tipo de trabalho já é largamente efetuado a nível transcriptômico e proteômico. Entretanto, em se tratando de metabolômica, isso já é bem mais incomum (FORST, 2006). Adicionalmente, a utilização da metabolômica para a diferenciação de organismos também vem sendo usada fora da botânica. Challis e Hopwood (2003) compararam algumas espécies do gênero Streptomyces, organizando-as evolutivamente a partir da presença de metabólitos secundários com efeito antibiótico. Em outro exemplo, Viant et al. (2003) conseguiram diferenciar os mariscos (Haliotis rufescens) normais dos portadores da síndrome de “withering” utilizando espectros de RMN de diversos biofluidos. Perspectivas para a metabolômica Recebe o nome de repetitivo aquele resultado que é obtido mais de uma vez, sob as mesmas condições, com os mesmos equipamentos e no mesmo laboratório. É chamado de reproduzível aquele resultado que é obtido em condições parecidas, com equipamentos similares e em um outro laboratório (FUKUSAKI e KOBAYASHI, 2005). A metabolômica atualmente tropeça em um problema que, de certa forma, é comum a outras plataformas tecnológicas, como a proteômica ou a transcriptômica. Esse problema é a falta de um protocolo padrão (ROCHFORT, 2005). Como cada laboratório utiliza uma determinada seqüência de procedimentos com seus próprios equipamentos, fica difícil de manter a reprodutibilidade, ou até mesmo a repetitividade (ROBERTSON, 2005). 27 A falta de um protocolo padrão existe hoje porque as próprias ferramentas de fracionamento da amostra e análise dos resultados necessitam ser melhoradas (FUKUSAKI e KOBAYASHI, 2005; WECKWERTH e MORGENTAL, 2005). No fracionamento, as ferramentas de maior sensibilidade esbarram na baixa resolução dos resultados, enquanto aquelas que primam pela resolução, exigem uma grande quantidade de amostra, limitando a sua utilização (ROBERTSON, 2005). Contudo, o principal desafio a ser enfrentado pela metabolômica é desenvolver ferramentas para comparar, entender e aplicar todo o conhecimento gerado por ela. Uma boa parte dos dados obtidos é simplesmente rejeitada pela falta de uma tecnologia que o entenda (MUNGUR et al., 2005; KRISHNAN et al., 2005; ROBERTSON, 2005). Conhecer o perfil metabólico de um organismo por si só é importante, mas não é suficiente. Faz-se necessária a integração das informações obtidas pela metabolômica, proteômica, transcriptômica e genômica (ROBERTSON, 2005; ROCHFORT, 2005, KLENO et al., 2004). Dessa forma seria possível prever como uma supressão ou uma super expressão gênica influenciaria no perfil protéico e metabólico, assim como também prever a influência de metabólitos inseridos no organismo (alimentos e remédios, principalmente) na expressão gênica (FUKUSAKI e KOBAYASHI, 2005; GIBNEY et al., 2005; ROBERTSON, 2005). Essa união de esforços é o objetivo da Biologia de sistemas (ROCHFORT, 2005). Considerações Finais O desenvolvimento destas plataformas tecnológicas conhecidas como “ômicas” tem trazido uma série de conhecimentos para a ciência. Sem dúvida, a genômica é a mais desenvolvida. Isso porque ela já tem protocolos determinados e ferramentas de fracionamento e análise dos resultados bem desenvolvidos. Entretanto, ainda há muito que se descobrir e melhorar na genômica (BINNECK, 2004). Contudo, muitas das reações e interações que definem um processo biológico, a diferenciação de um organismo ou mesmo a interação entre vários seres está fora da esfera genômica (GOODACRE, 2005; HÜTTENHOFER e VOGEL, 2006). Por isto que existe a necessidade de se desenvolver as outras plataformas (HÜTTENHOFER e VOGEL, 2006; BINNECK, 2004). 28 Nem todos os fragmentos de DNA de uma célula representam um gene, assim como nem todo gene vai dar origem a apenas um RNAm. A transcriptômica tem a função de descobrir que genes estão sendo transcritos sob uma determinada condição (NAKANISHI e NUREKI, 2005). Entretanto, as técnicas utilizadas para esse fim não conseguem detectar todos os RNAm, deixando de fora aqueles encontrados em concentrações muito baixas. Muitos desses podem estar relacionados a processos regulatórios importantes, sendo responsáveis por codificar as proteínas que iniciarão alguma importante modificação fisiológica da célula (RIGOUTSOS et al., 2006; NAKANISHI e NUREKI, 2005; FRANK e HARGREAVES, 2003). Por isso os dados obtidos pela transcriptômica, apesar de muito importantes e úteis, nem sempre refletem a expressão protéica da célula (BINNECK, 2004; FEDER e WALSER, 2005). O número de proteínas diferentes produzidas por um organismo excede e muito o número de genes do mesmo. Isso se deve às várias modificações pós transcricionais e traducionais que ocorrem com o RNAm e a seqüência de aminoácidos produzida, respectivamente. Devido a esse aumento das variações possíveis, torna-se mais trabalhoso conhecer o proteoma completo de um organismo (VIVANCO et al., 2003). As técnicas utilizadas na proteômica, pelo mesmo motivo, acabam por Ter problemas similares àquelas da transcriptômica. As baixas concentrações de proteínas regulatórias e iniciadoras de processos biológicos impedem, às vezes, a detecção destas (ROCHA et al., 2005; VIVANCO et al., 2003). Mesmo com o desenvolvimento de novas estratégias, como por exemplo, o pré-fracionamento da amostra ou a utilização de corantes altamente específicos, ainda é muito difícil detectar tais proteínas (ROCHA et al., 2005; MÄDER, 2002). A metabolômica foi criada para analisar um outro grupo de moléculas ainda mais diversificado que o das proteínas. Estas moléculas são chamadas de metabólitos (ROBERTSON, 2005; FIEHN, 2002). De acordo com a sua presença em diferentes organismos, assim como a sua participação em rotas metabólicas importantes, esses metabólitos são classificados em primários e secundários (CHALLIS e HOPWOOD, 2003; HALL, 2006). Os primários são aqueles comuns a qualquer célula e que participam de processos celulares importantes e que se conservaram ao longo da evolução (SMEDSGAARD e NIELSEN, 2005). Os 29 secundários são aqueles encontrados somente em algumas famílias ou gêneros de plantas e fungos e que estão relacionados às processos específicos, como a defesa contra patógenos, ou mesmo à estrutura da célula (DIXON, 2001; CHALLIS e HOPWOOD, 2003). As dificuldades para se conhecer todo o metaboloma de um organismo, célula ou biofluido é ainda maior do que àquelas encontradas nas análises de RNAm ou proteínas. Os metabólitos são estruturalmente muito diversos, por isso necessitam da utilização de vários equipamentos de fracionamento (FIEHN, 2002). Além disto, eles são encontrados em concentrações muito variadas entre si, tornando a detecção e a resolução de suas estruturas um processo muito difícil, mesmo com equipamentos muito sensíveis (MUNGUR et al., 2005). Por fim, a concentração deles é muito sensível a qualquer modificação fisiológica ou ambiental. Isso faz com que os protocolos de preparo e fracionamento das amostras possuam baixa reprodutibilidade, ou mesmo, repetibilidade (FUKUSAKI e KOBAYASHI, 2005). Assim como as outras plataformas referidas anteriormente, a metabolômica possui uma série de etapas. Em todas elas, uma série de fatores pode causar erros experimentais que afetariam diretamente o resultado da pesquisa. Além disto, os trabalhos de metabolômica exigem uma série de equipamentos muito caros e, principalmente, profissionais altamente capacitados para manipulá-los (FUKUSAKI e KOBAYASHI, 2005; ROCHFORT, 2005; WECKWERTH e MORGENTHAL, 2005). Estes fatores limitam os trabalhos de metabolômica aos grandes centros de pesquisa, onde já existem esses pré-requisitos (ROBERTSON, 2005). A metabolômica tem sido empregada em várias áreas da ciência e para diversos fins. Na farmacêutica, sobretudo na parte de toxicologia e desenvolvimento de fármacos, a metabolômica está sendo empregada para se identificar marcadores biológicos de toxicidade ou doenças, no entendimento do mecanismo de ação de várias toxinas e na seleção de fármacos efetivos e seguros (ROBERTSON, 2005; WECKWERTH e MORGENTHAL, 2005). Na área nutricional, a metabolômica pode ser aplicada para se conhecer os efeitos que os compostos não nutrientes que estão presentes no alimento causam no indivíduo, assim como entender os efeitos causados pela falta ou excesso de determinado nutriente na manutenção da saúde ou no desenvolvimento de doenças (GERMAN et al., 2003; GIBNEY et al., 2005; WATKINS et al., 2001). Na área ecológica, a metabolômica tem sido aplicada, 30 sobretudo, na detecção de alterações imprevistas em OGM, demonstrando que a inserção de apenas um gene pode alterar a concentração de centenas de metabólitos, assim como levar à produção de dezenas de compostos de importância farmacêutica (MUNGUR et al., 2005). Além disso, essa plataforma tecnológica pode ser aplicada na identificação dos metabólitos que diferenciam variedades de uma planta de interesse agronômico que são resistentes a uma determinada praga daquelas que são susceptíveis. Entretanto, a aplicação da metabolômica na ecologia ainda abrange outros campos, como por exemplo, a diferenciação de espécimes normais daqueles portadores de doenças, de uma forma não invasiva (CHALLIS e HOPWOOD, 2003) ou o efeito de compostos bioativos em plantas de interesse agronômico (OTT et al., 2003; ARANIBAR et al., 2001). Na evolução, a metabolômica pode ser aplicada na organização evolutiva de diferentes espécies de um mesmo gênero a partir do perfil metabólico (VIANT et al., 2003). Na metabolômica ainda há muito que se desenvolver. A primeira coisa a ser feita é estabelecer um protocolo padrão (FUKUSAKI e KOBAYASHI, 2005). Uma vez que cada laboratório desenvolve seus projetos a sua maneira, fica complicado reproduzi-los. Porém, o mais importante a ser melhorado, tanto para a metabolômica quanto para as outras plataformas tecnológicas, são as ferramentas de fracionamento e análise das amostras, principalmente as do segundo grupo (ROBERTSON, 2005). As informações obtidas pela metabolômica são de grande valia em qualquer das áreas já citadas. Entretanto, a integração dessas informações com aquelas obtidas pelas outras plataformas tecnológicas (proteômica, transcriptômica e genômica, principalmente) trará muito mais avanços no que tange relacionar o genótipo e o fenótipo dos organismos (ROCHFORT, 2005; BINNECK, 2004). Referências Bibliográficas AHARONI, A.; JONGSMA, M. A.; BOUWMEESTER, H. J. Volatile science? Metabolic engineering of terpenoids in plants. Trends in Plant Science, Oxford, v. 10, n. 12, p. 594-602, 2005. 31 ARANIBAR, N.; SINGH, B. K.; STOCKTON, G. W.; OTT, K. H. Automated mode-ofaction detection by metabolic profiling. Biochemical and Biophysical Research Communications, San Diego, v. 286, n. 1, p. 150-155, 2001. ASZTALOS, B.; LEFEVRE, M.; WONG, L.; FOSTER, T. A.; TULLEY, R.; WINDHAUSER, M.; ZHANG, W.; ROHEIM, P. S. Differential response to low-fat diet between low and normal HDL-cholesterol subjects. Journal of Lipid Research, Bethesda, USA, v. 41, p. 321-328, 2000. BECKWITH-HALL, B. M.; HOLMES, E.; LINDON, J. C.; GOUNARIDES, J.; VICKERS, A.; SHAPIRO, M.; NICHOLSON, J. K. NMR-based metabonomic studies on the biochemical effects of commonly used drug carrier vehicles in the rat. Chemical Research in Toxicology, Washington, v. 15, p. 1136-1141, 2002. BINNECK, E. As ômicas: integrando a bioinformação. Biotecnologia Ciência e Desenvolvimento, Uberlândia, v. 1, n. 32, p. 28-37, 2004. BINO, R. J.; HALL, R. D.; FIEHN, O.; KOPKA, J.; SAITO, K.; DRAPER, J.; NIKOLAU, B. J.; MENDES, P.; ROESSNER-TUNALI, U.; BEALE, M. H. Potential of metabolomics as a functional genomics tool. Trends in Plant Science, Oxford, v. 9, p. 418-425, 2004. BOLLARD, M. E.; HOLMES, E.; LINDON, J. C.; MITCHELL, S. C.; BRANSTETTER, D.; ZHANG, W.; NICHOLSON, J. K. Investigations into biochemical changes due to diurnal variation and estrus cycle in female rats using high resolution (1) H NMR spectroscopy of urine and pattern recognition. Analytical Biochemistry, Orlando, v. 295, p. 194-202, 2001. BRINDLE, J. T.; NICHOLSON, J. K.; SCHOFIELD, P. M.; GRAINGER, D. J. HOLMES, E. Application of chemometrics to 1H NMR spectroscopic data to investigate a relationship between human serum metabolic profiles and hypertension. The Analyst, Cambridge, v. 128, p. 32-36, 2003. CALSA JUNIOR, T.; BENEDITO, V. A.; FIGUEIRA, A. V. O. Análise serial da expressão gênica. Biotecnologia Ciência e Desenvolvimento, Uberlândia, v. 33, p. 88-100, 2004. CASSIDY, A. Are herbal remedies and dietary supplements safe and effective for breast cancer patients? Breast Cancer Research, London, v. 5, n. 6, p. 300-302, 2003. CHALLIS, G. L.; HOPWOOD, D. A. Synergy and contingency as driving forces for the evolution of multiple secondary metabolite production by Streptomyces species. National Academy of Science of the United States, Washington, v. 100, p. 1455514561, 2003. DEMAIN, A. L. ; VAISHNAV, P. Involvement of nitrogen-containing compounds in beta-lactam biosynthesis and its control. Critical Reviews in Biotechnology, v. 26, n. 2, p. 67-82, 2006. 32 DIXON, R. A. Natural products and plant disease resistance. Nature, London, v. 411, p. 843-847, 2001. DURAN, A. L.; YANG, J.; WANG, L.; SUMNER, L. W. Metabolomics spectral formatting, alignment and conversion tools (MSFACTs). Bioinformatics, Oxford, v. 19, p. 2283-2293, 2003. ENGEL, S.; JENSEN, P. R.; FENICAL, W. Chemical ecology of marine microbial defense. Journal of Chemical Ecology, Dordrecht, Netherlands, v. 28, n. 10, p. 1971-1985, 2002. FEDER, M. E.; WALSER, J. C. The biological limitations of transcriptomics in elucidating stress and stress responses. Journal of Evolutionary Biology. Oxford, v. 18, n. 4, p. 901-910, 2005. FERNIE, A. R.; TRETHEWEY, R. N.; KROTZKY, A. J.; WILLMITZER, L. Metabolite profiling: From diagnostics to systems biology. Natural Review of Molecular Cell Biology, v. 5, p. 763-769, 2004. FIEHN, O. Metabolomics–the link between genotypes and phenotypes. Plant Molecular Biology, Dordrecht, Netherlands, v. 48, p. 155-171, 2002. FIEHN, O.; KOPKA, J.; DORMANN, P.; ALTMANN, T.; TRETHEWEY, R. N.; WILLMITZER, L. Metabolite profiling for plant functional genomics. Nature Biotechnology, New York, v. 18, p. 1157-1161, 2000. FORST, C. V. Host–pathogen systems biology. Drug Discovery Today, New Jersey, USA, v. 11, n 6, p. 220-227, 2006. FRANK, R.; HARGREAVES, R. Clinical biomarkers in drug discovery and development. Natural Review Drug Discovery, v. 2, p. 566-580, 2003. FUKUSAKY, E.; KOBAYASHI, A. Plant metabolomics: potential for practical operation. Journal of Bioscience and Bioengineering, Osaka, Japan, v. 100, n. 4, p. 347-354, 2005. GERMAN, J. B.; ROBERTS, M. A.; WATKINS, S. M. Genomics and metabolomics as markers for the interaction of diet and health: Lessons from lipids. Journal of Nutrition. Bethesda, USA, v. 133, p. 2078-2083, 2003. GERMAN, J. B.; WATKINS, S. M.; Fay, L. B. Metabolomics in practice: Emerging knowledge to guide future dietetic advice toward individualized health. Journal of the American Dietetic Association, Chicago, v. 105, p. 1425-1432, 2005. GIBNEY, M. J.; WALSH, M.; BRENNAN, L.; ROCHE, H. M.; GERMAN, B.; VAN OMMEN, B. Metabolomics in human nutrition: opportunities and challenges. The American Journal of Clinical Nutrition, Bethesda, USA, v. 82, p. 497-503, 2005. 33 GOODACRE, R. Making sense of the metabolome using evolutionary computation: seeing the wood with the trees. Journal of Experimental Botany, Oxford, v. 56, n. 410, p. 245-254, 2005. GOODACRE, R.; VAIDYANATHAN, S.; DUNN, W. B.; HARRIGAN, G. G.; KELL, D. B. Metabolomics by numbers: Acquiring and understanding global metabolite data. Trends in Biotechnology, Oxford, v. 22, p. 245-252, 2004. HALL, R. D. Plant metabolomics: from holistic hope, to hype, to hot topic. New Phytologist, Oxford, v. 169, p. 453-468, 2006. HOLMES, E.; NICHOLLS, A. W.; LINDON, J. C.; RAMOS, S.; SPRAUL, M.; NEIDIG, P.; CONNOR, S. C.; CONNELLY, J.; DAMMENT, S. J.; HASELDEN, J. Development of a model for classification of toxin-induced lesions using 1H NMR spectroscopy of urine combined with pattern recognition. NMR in Biomedicine, v. 11, p. 235-244, 1998. HORNING, E. C.; HORNING, M. G. Metabolic Profiles - Gas-Phase Methods for Analysis of Metabolites. Clinic Chemistry, v. 17, p. 802-809, 1971. HUTTENHOFER, A.; VOGEL, J. Experimental approaches to identify non-coding RNAs. Nucleic Acid Research, v. 34, n. 2, p. 635-646, 2006. JAMES, P. Protein identification in the post-genomic era: the rapid rise of proteomics. Quarterly Reviews of Biophysics, Cambridge, v. 30, n. 4, p. 279-331, 1997. KLENO, T. G.; KIEHR, B.; BAUNSGAARD, D.; SIDELMANN, U. G. Combination of ‘omics’ data to investigate the mechanism(s) of hydrazineinduced hepatotoxicity in rats and to identify potential biomarkers. Biomarkers, London, v. 9, p. 116-138, 2004. KLOSE, J. Protein mapping by combined isoeletric focusing and electrophoresis of mouse tissues. A novel approach to test for induced point mutations in mammals. Archives of Toxicology, Heidelberg, Germany, v. 26, p. 231-243, 1975. KRISHNAN, P.; KRUGER, N. J.; RATCLIFFE, R. G. Metabolite fingerprinting and profiling in plants using NMR. Journal of Experimental Botany, Oxford, v. 56, n. 410, p. 255-265, 2005. LENZ, E. M.; BRIGHT, J.; WILSON, I. D.; MORGAN, S. R.; NASH, A. F. A 1H NMRbased metabonomic study of urine and plasma samples obtained from healthy human subjects. Journal of Pharmaceutical and Biomedical Analysis, Amsterdam, v. 33, p. 1103-1115, 2003. LINDON, J. C.; HOLMES, E.; NICHOLSON, J. K. Metabonomics and its role in drug development and disease diagnosis. Expert Review of Molecular Diagnosis, v. 4, p. 189-199, 2004a. 34 LINDON, J. C.; HOLMES, E.; NICHOLSON, J. K. Metabonomics: Systems biology in pharmaceutical research and development. Current Opinnion Molecular Therapy, v. 6, p. 265-272, 2004b. LINDON, J. C.; HOLMES, E.; NICHOLSON, J. K. So what’s the deal with metabonomics? Analitical Chemistry, v. 75, p. 384-391, 2003. MACHUCA-TZILI, L.; THORPE, H.; ROBINSON, T. E.; SEWRY, C.; BROOK, J. D. Flies deficient in Muscleblind protein model features of myotonic dystrophy with altered splice forms of Z-band associated transcripts. Human genetics, v. 30, p. 113, 2006. MÄDER, U.; HOMUTH, G.; SCHARF, C.; BÜTTNER, K.; BODE, R.; HECKER, M. Transcriptome and Proteome Analysis of Bacillus subtilis Gene Expression Modulated by Amino Acid Availability. Journal of Bacteriology, Washington, v. 184, n. 15, p. 4288-4295, 2002. MARASCHIN, M.; VERPOORTE, R. Engenharia do metabolismo secundário. Biotecnologia Ciência & Desenvolvimento, Brasília, v. 23, p. 24-28, 1999. MUNGUR, R.; GLASS, A. D. M.; GOODENOW, D. B.; LIGHTFOOT, D. A. Metabolite fingerprinting in Nicotiana tabacum altered by the Escherichia coli glutamate desidrogenase gene. Journal of Biomedicine and Biotechnology, Sylvania, v. 2, p. 198-214, 2005. NAKANISHI, K.; NUREKI, O. Recent progress of structural biology of tRNA processing and modification. Molecules and Cells, Kowloon, Hong Kong, v. 19, n. 2, p. 157-166, 2005. NEWMAN, D. J.; CRAGG, G. M.; SNADER, K. M. Natural products as a source of new drugs over the period 1981–2002. Journal of Natural Products, Washington, v. 66, p. 1022-1037, 2003. NICHOLLS, A. W.; HOLMES, E.; LINDON, J. C. SHOCKCOR, J. P.; FARRANT, R. D.; HASELDEN, J. N.; DAMMENT, S. J.; WATERFIELD, C. J.; NICHOLSON, J. K. Metabonomic investigations into hydrazine toxicity in the rat. Chemical Research in Toxicology, Washington, v. 14, p. 975-987, 2001. NICHOLSON, J. K.; CONNELLY, J.; LINDON, J. C.; HOLMES, E. Metabonomics: A platform for studying drug toxicity and gene function. Nature Reviews Drug Discovery, v. 1, p. 153-161, 2002. NICHOLSON, J. K.; LINDON, J. C.; HOLMES, E. ‘Metabonomics’: Understanding the metabolic responses of living systems to pathophysiological stimuli via multivariate statistical analysis of biological NMR spectroscopic data. Xenobiotica, London, v. 29, p. 1181-1189, 1999. NICHOLSON, J. K.; WILSON, I. D. Opinion: Understanding ‘global’ systems biology: Metabonomics and the continuum of metabolism. Natural Review of Drug Discovery, v. 2, p. 668-676, 2003. 35 O’FARRELL, P.H. High resolution two-dimensional electrophoresis of proteins. Journal of Biological Chemistry, Bethesda, USA, v. 270, p. 4007-4021, 1975. OTT, K. H.; ARANIBAR, N.; SINGH, B.; STOCKTON, G. W. Metabonomics classifies pathways affected by bioactive compounds. Artificial neural network classification of NMR spectra of plant extracts. Phytochemistry, Amsterdam, v. 62, n. 6, p. 971-985, 2003. PARKS, E. J.; HELLERSTEIN, M. K. Carbohydrate-induced hypertriacylglycerolemia: Historical perspective and review of biological mechanisms. American Journal of Clinical Nutrition, Bethesda, USA, v. 71, p. 412-433, 2000. PATTON, G. W.; STEPHENS, R.; SIDOROV, I. A.; XIAO, X.; LEMPICKI, R. A.; DIMITROV, D. S.; SHOEMAKER, R. H.; TUDOR, G. Transcriptomic response to differentiation induction. BMC Bioinformatics, London, v. 81, n. 7, p. 1-10, 2006. PODGORNICK, R. Electrostatic contribution to the persistence length of a semiflexible dipolar chain. Physical review, E, Statistical, nonlinear, and soft matter physics, v. 70, p. 1-11, 2004. REO, N. V. NMR-based metabolomics. Drug Chemical Toxicology,Monticello, USA, v. 25, p. 375-382, 2002. RIGOUTSOS, I.; HUYNH, T.; MIRANDA, K.; TSIRIGOS, A.; MCHARDY, A.; PLATT, D. Short blocks from the noncoding parts of the human genome have instances within nearly all known genes and relate to biological processes. Proceedings of the National Academy of Sciences of the United States of America, Washington, v. 103, n. 17, p. 6605-6610, 2006. RISCHER, H.; OKSMAN-CALDENTEY, K. M. Unintended effects in genetically modified crops: revealed by metabolomics? Trends in Biotechnology, Oxford, v. 24, n. 3, p. 102-104, 2006. ROBERTS, J. K. NMR adventures in the metabolic labyrinth within plants. Trends in Plant Science, Oxford, v. 5, p. 30-34, 2000. ROBERTSON, D. G. Metabonomics in toxicology: a review. Toxicological Sciences, v. 85, p. 809-822, 2005. ROBERTSON, D. G.; REILY, M. D.; LINDON, J. C.; HOLMES, E.; NICHOLSON, J. K. Metabonomic technology as a tool for rapid throughput in vivo toxicity screening. Trends in Plant Science, Oxford, v. 10, p. 130-141, 2002. ROBERTSON, D. G.; REILY, M. D.; SIGLER, R. E.; WELLS, D. F.; PATERSON, D. A.; BRADEN, T. K. Metabonomics: Evaluation of nuclear magnetic resonance (NMR) and pattern recognition technology for rapid in vivo screening of liver and kidney toxicants. Toxicological Science, v. 57, p. 326-337, 2000. ROCHA, T. L.; COSTA, P. H. A. da; MAGALHÃES, J. C. C.; EVARISTO, R. G. S.; VASCONCELOS, E. A. R. de; COUTINHO, M. V.; PAES, N. S.; SILVA, M. C. M. da; 36 GROSSI-DE-SÁ, M. de F. Eletroforese bidimensional e análise de proteomas. Brasília: Embrapa Recursos Genéticos e Biotecnologia, 2005. 12p. (Embrapa Recursos Genéticos e Biotecnologia. Comunicado Técnico, 136). ROCHFORT, S. Metabolomics Reviewed: A New “Omics” Platform Technology for Systems Biology and Implications for Natural Products Research. Journal of Natural Products, Washington, v. 68, n. 12, p. 1813-1820, 2005. SMEDSGAARD, J.; NIELSEN, J. Metabolite profiling of fungi and yeast: from phenotype to metabolome by MS and informatics. Journal of Experimental Botany, Oxford, v. 56, n. 410, p. 273-286, 2005. STEUER, R.; KURTHS, J.; FIEHN, O.; WECKWERTH, W. Observing and interpreting correlations in metabolomic networks. Bioinformatics, Oxford, v. 19, n. 8, p. 1019-1026, 2003. STRAUS, S. K. Recent developments in solid-state magic-angle spinning, nuclear magnetic resonance of fully and significantly isotopically labelled peptides and proteins. Philosophical transactions of the Royal Society of London, Series B, Biological sciences, London, v. 359, n. 1446, p. 997-1008, 2004. VIANT, M. R.; ROSENBLUM, E. S.; TIEERDEMA, R. S. NMR-based metabolomics: A powerful approach for characterizing the effects of environmental stressors on organism health. Environmental Science and Technology, Washington, v. 37, p. 4982-4989, 2003. VIVANCO, F.; LOPEZ-BESCOS, L.; TUNON, J.; EGIDO, J. Proteomics and cardiovascular disease. Revista Espanhola de Cardiologia, v. 56, n. 3, p. 289-302, 2003. WANT, E. J.; O’MAILLE, G.; SMITH, C. A.; BRANDON, T. R.; URITBOONTHAI, C. Q.; TRAUGER, S. A.; SIUZDAK, G. Solvent-dependent metabolite distribution, clustering, and protein extraction from serum profiling with mass spectrometry. Analitical Chemistry, v. 78, p. 743-752, 2006. WATKINS S. M.; HAMMOCK B. D.; NEWMAN J. W.; GERMAN J. B. Individual metabolism should guide agriculture toward foods for improved health and nutrition. American Journal of Clinical Nutrition, Bethesda, USA, v. 74, p. 283-286, 2001. WECKWERTH, W. Metabolomics in systems biology. Annual Review of Plant Biology, Palo Alto, USA, v. 54, p. 669-689, 2003. WECKWERTH, W.; MORGENTHAL, K. Metabolomics: from patterns recognition to biological interpretation. Drug Discovery Today: Targets, v. 10, n. 22, p. 1551-1558, 2005. WILSON, I. D.; PLUMB, R.; GRANGER, J.; MAJOR, H.; WILLIAMS, R.; LENZ, E. M. HPLC-MS-based methods for the study of metabonomics. Journal of chromatography. B, Analytical technologies in the biomedical and life sciences, v. 817, p. 67-76, 2005. 37 ZANCAN, G. A microbiologia na era da genômica: o avanço de ciências como a física e a computação criou instrumentos poderosos de análise. Ciência e Cultura, São Paulo, v. 54, n. 1, p. 4-5, 2002. ZHANG, Q. ; ZHAO, Y.; WANG, B. ; TU, G. New Triterpenoid Saponins from Stelmatocrypton khasianum. Chemical & pharmaceutical bulletin, Tokyo, v. 51, n. 5, p. 574-578, 2003. 38
Baixar