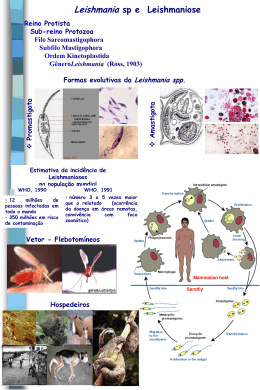
POLIMORFISMOS DE MIRNA NA SUSCEPTIBILIDADE A LEISHMANIOSE CUTÂNEA CAUSADA POR L. guyanensis NO ESTADO DO AMAZONAS Bolsista: Tirza Gabrielle Ramos de Mesquita Orientador: Dr. Rajendranath Ramasawmy ¹, Colaboradores : Luan Oliveira, Felipe Jules, José Junior, Wonei Vital, Sinésio Talhari, Anette Chrusciak-Talhari, Jorge Guerra² Fundação de Medicina Tropical Doutor Heitor Vieira Dourado¹, Universidade Do Estado do Amazonas² INTRODUÇÃO Leishmaniose é uma doença infecciosa crônica causada por parasitas protozoários do gênero Leishmania spp¹. A interação entre patógeno e hospedeiro possui vários níveis de complexidade, incluindo o background genético do hospedeiro, virulência do parasito, vetor e ambiente favorável para o desenvolvimento das lesões². No estado do Amazonas, L. guyanensis é a espécie mais prevalente que causa leishmaniose cutânea (LC).³ Há estudos que demonstram uma agregação familiar da LC e LM nas áreas endêmicas, 4 e foi sugerido que existe um gene com maior efeito controlando a susceptibilidade a leishmaniose tegumentar.5 MicroRNAs (miRNAs) são moléculas de RNA de fita simples não-codificantes de 18-26 nucleotídeos que induzem o silenciamento e, possivelmente, a expressão de seus genes alvos.6 RESULTADOS Para os SNPs hsa-miR-499 e hsa-miR-146 foram genotipadas 584 e 595 amostras, respectivamente. As frequências alélicas de ambos os polimorfismos estão dentro do equilíbrio de Hardy-Weinberg. O alelo C de hsamiR-146 está associado com a susceptibilidade a LC quando comparado ao grupo controle. Indivíduos homozigotos para o alelo C tem duas vezes mais chance de desenvolver a LC durante a infecção pela L. guyanensis (P= 0.02; OR=1.9 [95% CI 1.1 – 3.3]). Nenhuma associação foi observada com o SNP hsa-miR-499. OBJETIVOS Investigamos os variantes do gene miRNA na susceptibilidade a LC causada por L. guyanensis em pacientes com LC e indivíduos saudáveis (controles), sem sinal ou histórico de LC, procedentes das mesmas áreas endêmicas. MATERIAIS E MÉTODOS Os SNPS foram identificados por PCR-RFLP, para análise da discriminação alélica do gene miRNA. Os seguintes iniciadores foram desenhados para genotipar o SNP hsa-miR-146 (rs2910164): F: 5’-CAT GGG TTG TGT CAG TGT CAG AAC T-3’ e R: 5’-GCC TTC TGT CTC CAG TCT TCC3’, gerando um fragmento de 146 pb pelo PCR. A discriminação alélica foi realizada utilizando a enzima HpyCH4III, específica para o alelo G, gerando dois fragmentos (121+25 pb), enquanto na presença do alelo C o sítio de restrição desaparece (146 pb). Para o SNP hsa-miR-499 (rs3746444): F: 5’-CAC TTC CCT GCC AAA TCC CCG TCC CTC CCA-3’ e R: 5’-AGT GAT GTT TAA CTC CTC TCC GCG TGA-3’, gerando um fragmento de 140 pb pelo PCR. A discriminação alélica foi feita utilizando a enzima HpyCH4IV, específica para o alelo C, gerando dois fragmentos (110+30 pb), enquanto na presença do alelo T o sítio de restrição desaparece (140 pb). Frequências dos genótipos e alelos Genótipos Casos Controles miRNA-146 G>C n= 319 n= 276 CC GC GG Alelos C G 53 (17%) 150 (47%) 116 (36%) 25 (9%) 146 (53%) 105 (38%) 256 (40%) 382 (60%) n= 332 196 (36%) 356 (64%) n= 252 267 (80%) 61 (19%) 4 (1%) 206 (82%) 42 (17%) 4 (1%) NS NS NS 595 (90%) 69 (10%) 454 (90%) 50 (10%) NS NS miRNA-499 C>T TT CT CC Alelos T C Comparisons GG Vs.CC GC+GG Vs.CC GC Vs.CC G Vs.C p-value 0.02 0.006 0.006 0.1 OR [95%CI] pv 1.9 [1.1 - 3.3] 0.5 [0.3 - 0.82] 0.5 [0.29 - 0.82] 1.2 [0.9-1.5] 1 Tabela 1. Frequências genotípicas e alélicas do polimorfismo de nucleotídeo hsa-miR-499 e hsa-miR-146 em pacientes com leishmania cutânea (LC) e controles saudáveis. AGRADECIMENTOS Agradeço a FAPEAM, CNPq, CAPES e FMT-HVD. REFERENCIAS BIBLIOGRÁFICAS 1. Alvar J, Vélez ID, Bern C, Herrero M, Desjeux P, Cano J et al. Leishmaniasis worldwide and global estimates of its incidence. PLoS One 2012; 7: e35671. 2.Handman E, Elso C, Foote S. Genes and susceptibility to leishmaniasis. Adv Parasitol 2005; 59: 1-75. 3. Benicio Ede A, Gadelha EP, Talhari A, et al. Combining diagnostic procedures for the management of leishmaniasis in areas with high prevalence of Leishmania guyanensis. An Bras Dermatol 2011; 86:1141-4. 4. Castellucci L, Cheng LH, Araújo C, Guimarães LH, Lessa H, Machado P et al. Familial aggregation of mucosal leishmaniasis in northeast Brazil. Am J Trop Med Hyg 2005; 73: 69-73. 5. Alcaïs A, Abel L, David C, Torrez ME, Flandre P, Dedet JP. Evidence for a major gene controlling susceptibility to tegumentary leishmaniasis in a recently exposed Bolivian population. Am J Hum Genet 1997; 61: 968-79. 6. N. S. Geraci, J. C. Tan , M. A. Mcdowell. Characterization of microRNA expression profiles in Leishmania infected human phagocytes. Parasite Immunology, 2015, 37, 43–51 DOI: 10.1111
Baixar