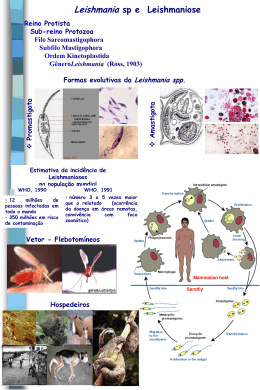
UNIVERSIDADE DO ESTADO DO AMAZONAS FUNDAÇÃO DE MEDICINA TROPICAL DR. HEITOR VIEIRA DOURADO PROGRAMA DE PÓS-GRADUAÇÃO EM MEDICINA TROPICAL MESTRADO EM DOENÇAS TROPICAIS E INFECCIOSAS ESTUDO DE ASSOCIAÇÃO DE POLIMORFISMOS DOS GENES IL-1b, FLI-1 e MCP-1 COM LEISHMANIOSE CUTÂNEA EM UMA POPULAÇÃO CASOCONTROLE DO ESTADO DO AMAZONAS, BRASIL KAROLINA DA COSTA SABINO MANAUS 2014 i KAROLINA DA COSTA SABINO ESTUDO DE ASSOCIAÇÃO DE POLIMORFISMOS DOS GENES IL-1b, FLI-1 e MCP-1 COM LEISHMANIOSE CUTÂNEA EM UMA POPULAÇÃO CASOCONTROLE DO ESTADO DO AMAZONAS, BRASIL Dissertação apresentada ao Programa de Pós-Graduação em Medicina Tropical da Universidade do Estado do Amazonas em Convênio com a Fundação de Medicina Tropical Dr. Heitor Vieira Dourado, para obtenção do grau de Mestre em Doenças Tropicais e Infecciosas. Orientador: Prof. Dr. Rajendranath Ramasawmy MANAUS 2014 ii Ficha Catalográfica S116e Sabino, Karolina da Costa. Estudo de associação de polimorfismos dos genes IL-1b, FLI-1 e FOLHA DE JULGAMENTO MCP-1 com leishmaniose cutânea em uma população caso-controle do estado do Amazonas, Brasil / Karolina da Costa Sabino – Manaus: Universidade do Estado do Amazonas, Fundação de Medicina Tropical Doutor Heitor Vieira Dourado, 2014. xvii. 93 f.: il. Dissertação apresentada ao Programa de Pós-Graduação em Medicina Tropical da Universidade do Estado do Amazonas – UEA/FMTHVD, 2014. Orientador: Profº. Dr. Rajendranath Ramasawmy 1. Leishmaniose 2.Polimorfismo genético 3. IL-1b 4. MCP-1 5. FLI-1 I. Título. CDU: 616.9 Ficha Catalográfica elaborada pela Bibliotecária Maria Eliana do N. Silva lotada na Escola Superior de Ciências da Saúde - UEA iii ESTUDO DE ASSOCIAÇÃO DE POLIMORFISMOS DOS GENES IL1b, FLI-1 e MCP-1 COM LEISHMANIOSE CUTÂNEA EM UMA POPULAÇÃO CASO-CONTROLE DO ESTADO DO AMAZONAS, BRASIL KAROLINA DA COSTA SABINO “Esta Dissertação foi julgada adequada para obtenção do Título de Mestre em Doenças Tropicais e Infecciosas, aprovada em sua forma final pelo Programa de Pós-Graduação em Medicina Tropical da Universidade do Estado do Amazonas em convênio com a Fundação de Medicina Tropical Dr. Heitor Vieira Dourado”. Banca Julgadora: ______________________________________ Presidente ______________________________________ Membro ______________________________________ Membro iv DEDICATÓRIA Dedico este trabalho aos meus pais, Maria Auxiliadora da Costa Sabino e Mizael da Silva Sabino, pelo apoio constante e incondicional, sem os quais seria impossível completar esta árdua caminhada. v AGRADECIMENTOS Em primeiro lugar a Deus, minha fonte de inspiração e sabedoria. Quem me sustentou e me deu forças em todos os momentos dessa jornada. Mesmo nos dias em que eu pensava em desistir e abandonar o sonho da pesquisa, Ele sempre colocava pessoas em meu caminho que pudessem me aconselhar e alegrar meu dia, e me mostrar que toda grande conquista requer uma árdua batalha. Obrigada meu Criador e Consolador, sem Ti não estaria aqui. Aos meus pais, Maria Auxiladora da Costa Sabino e Mizael da Silva Sabino meus sinceros e eternos agradecimentos, pois se fui atrás dos meus sonhos, foi porque vocês foram meus maiores incentivadores e patrocinadores. A vocês dedico todo meu amor, carinho e gratidão, por me educarem tão bem e por serem meus referenciais de caráter, honestidade, perseverança, mansidão e sabedoria. Por me ensinarem com exemplos concretos que a vida é cheia de lutas e dificuldades, mas que com fé e persistência sempre podemos vencer e tirar grandes lições de todas as adversidades. Aos meus queridos irmãos Karinna Sabino e Alexandre Sabino. Obrigada por existirem e por sempre torcerem por mim. Agradeço pelas palavras de incentivo e apoio que sempre liberaram a mim, e por toda ajuda que vocês me deram nessa etapa. Amo vocês de todo meu coração. Ao meu orientador Rajendranath Ramasawmy pela oportunidade de fazer o mestrado. À Dra Cintia Mara Costa de Oliveira, minha primeira e eterna orientadora. Quem me ensinou a dar os primeiros passos na pesquisa e que me inspirou pelo seu exemplo de caráter e seriedade. Obrigada pela amizade e oportunidade. À gerência de Leishmaniose na pessoa do Dr. Jorge Guerra e aos técnicos Amélia Bittencourt, Claudemir Félix e Rita pelo apoio na etapa de recrutamento dos pacientes. À Universidade do Estado do Amazonas (UEA) e à Fundação de Medicina Tropical Dr. Heitor Vieira Dourado (FMT/HVD) pela excelência no ensino e por proporcionarem aos seus alunos um ambiente favorável ao aprendizado. vi Aos colegas da turma de Pós-Graduação, que tornaram essa caminhada mais agradável. Obrigada pelas risadas, trocas de conhecimentos, confraternizações e convívio diário. Às amigas Luciane Souza e Priscila Bentes, que caminharam lado a lado comigo nesta jornada. Meninas, sem vocês esta caminhada teria sido muito mais árdua. De todo coração agradeço por tê-las em meu caminho, por todas as conversas, lamentações, incertezas, alegrias, gargalhadas e ajuda mútua. Levarei essa experiência para sempre comigo, e vocês certamente farão parte dessas lembranças. É bem verdade que cordão de três dobras não se rompe com facilidade, nós somos prova viva disso. Às amigas de todas as horas Belisa Maia, Brigite Stela e Lorena Sabrina. Presentes de Deus para minha vida e que nessa etapa tão crucial, foram muito mais que amigas, foram irmãs, conselheiras, psicólogas e incentivadoras. Amigas queridas, obrigada por acreditarem em mim, muitas vezes até mais do que eu. Amo vocês. Às amigas e aventureiras Bárbara Yole e Larissa Morais que estiveram comigo desde os tempos de graduação. Seguiram caminhos distintos dos meus, mas sempre estiveram presente para me ouvir, encorajar e torcer pelo meu sucesso. Que eu possa assistir de perto o sucesso de vocês também. A todos os amigos e familiares, tanto presentes quanto ausentes, mas que de alguma forma me deram uma palavra de incentivo e esperança. Que sempre mostraram estar na torcida por mim, desejando que tudo acabasse bem. À Geovana Broto, Heloísa Salomão e Helena D’Espindula por tão gentilmente terem nos recebido e ajudado com a parte estatística do projeto. Agradeço de coração, pois vocês nos receberam de modo tão acolhedor, e mesmo sabendo de nossas limitações foram pacientes e atenciosas. You girls are the best! Aos amigos do Laboratório de Pesquisa em Doenças Endêmicas (LPDE), pelos momentos de descontração e gargalhadas. Em especial à dona Maria, pela amizade, carinho, abraços apertados e pelos momentos de descontração, regado a muitas risadas e deliciosos cafezinhos. Às agências de fomento à pesquisa CNPq e CAPES pela bolsa concedida e pelos recursos disponibilizados para a execução do estudo. vii Meu agradecimento mais do que especial aos pacientes e voluntários sem leishmaniose, que por mais incômodo que fosse, aceitaram participar da pesquisa e depositaram total confiança em nossa equipe, nos recebendo em seus lares de forma tão acolhedora. Por fim, agradeço a todos que contribuíram de alguma forma, direta ou indiretamente, para que esse estudo fosse realizado. viii DECLARAÇÃO DAS AGÊNCIAS FINANCIADORAS À Superintendência da Zona Franca de Manaus – SUFRAMA e Fundação de Apoio Institucional Muraki-FMuraki pelo apoio na estrutura do Programa de Pós-Graduação em Medicina Tropical. À Coordenação de Aperfeiçoamento de Pessoal de Nível Superior (CAPES) pela concessão da bolsa de pesquisa. Ao Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq) pelo financiamento desta pesquisa (MCTI/CNPq/MS-SCTIE) sob numeração: 04181/20120. ix “I have no special talent. I am only passionately curious.” (Albert Einstein) “Nothing worth having was ever achieved without effort.” (Theodore Roosevelt) x RESUMO As leishmanioses representam um complexo de doenças com importante espectro clínico e diversidade epidemiológica, sendo causadas por um protozoário intracelular obrigatório pertencente ao gênero Leishmania. Nas áreas onde a leishmaniose é considerada endêmica, somente uma fração da população exposta ao parasita desenvolve a doença, sugerindo que fatores genéticos tenham um papel fundamental nesse contexto, associado a fatores ambientais, socioeconômicos, culturais, entre outros. Assim, com o propósito de identificar polimorfismos que possam ter influência no controle da susceptibilidade, o presente estudo teve como objetivo verificar o papel de polimorfismos genéticos em IL-1b, FLI-1 e MCP-1 associados com leishmaniose cutânea (LC) em uma população do estado do Amazonas. Para realização do estudo foram recrutados um total de 774 indivíduos, sendo 392 indivíduos com leishmaniose cutânea para compor o grupo caso, oriundos da demanda espontânea do ambulatório de Dermatologia da Fundação de Medicina Tropical Dr. Heitor Vieira Dourado (FMTHVD); e 382 indivíduos sem a doença para o grupo controle, provenientes da mesma área endêmica dos casos. O DNA genômico foi extraído a partir do creme leucocitário com o kit comercial de extração de DNA, de acordo com as instruções sugeridas pelo fabricante, ou pela técnica de “salting-out”. No que se refere aos SNPs IL1b_rs1143634 (IL-1b 3953 C/T), MCP1_rs1024611 (MCP-1 -2518 A/G), FLI1_rs619521 e FLI1_rs531894, não foram observadas associações genéticas com a susceptibilidade ou resistência à LC na população estudada. Para o SNP IL1b_rs16944 (IL-1b -511 C/T), foram detectadas diferenças estatisticamente significativas na distribuição alélica e genotípica do polimorfismo entre os grupos caso e controle, sugerindo associação do alelo C com susceptibilidade [P=0.027, OR=1.42 IC95% (1.04 – 1.94)], e do alelo T com resistência [P=0.036, OR=0.66 IC95% (0.45 – 0.96)] para LC. Para o SNP rs7930515 de FLI-1 foi observado valor de P “borderline”, podendo-se sugerir que o alelo C está associado com risco para LC [P=0.036, OR=2.38 IC95% (1.07 – 5.32)]. Os resultados obtidos no presente estudo reforçam o papel dos genes IL1-b e FLI-1 associados com resistência ou susceptibilidade a LC. Descritores: Leishmaniose, polimorfismo genético, IL-1b, MCP-1, FLI-1. xi ABSTRACT Leishmaniasis represents a complex of diseases with important clinical spectrum and epidemiological diversity, being caused by an obligate intracellular protozoan belonging to the genus Leishmania. In areas where leishmaniasis is considered endemic, just a part of the population exposed to the parasite get sick, suggesting that genetic factors have a key role in this context, associated with socioeconomic, environmental and cultural factors. Thus, in order to identify polymorphisms that might influence the control of susceptibility, this study aimed to determine the role of genetic polymorphisms in IL-1b, FLI-1 and MCP-1 associated with cutaneous leishmaniasis (CL) in a population of the state of Amazonas. To conduct the study were recruited 774 subjects, being 392 subjects with cutaneous leishmaniasis to compound the case group, all of them derived from the spontaneous demand of the dermatology ambulatory of Tropical Medicine Foundation Dr. Heitor Vieira Dourado (FMT-HVD); and 382 individuals without the disease to the control group, from the same endemic area of case group. Genomic DNA was extracted from the buffy coat with the commercial DNA extraction kit according to the instructions suggested by the manufacturer or by the technique of salting-out. In regard to IL-1b_rs1143634 (IL-1b 3953 C/T), MCP1_rs1024611 (MCP-1 -2518 A/G), FLI1_rs619521 and FLI1_rs531894 SNPs, were not observed genetic associations with susceptibility or resistance to LC in the studied population. For IL1b_rs16944 (IL-1b -511 C / T) SNP, statistically significant differences were found in allele and genotype distribution of the polymorphism between case and control groups, suggesting an association of the C allele with susceptibility [P = 0.027, OR = 1:42 95 % (1.04 - 1.94)], and the T allele with resistance [P = 0.036, OR = 0.66 95% (0.45 - 0.96)] to LC. For rs7930515 SNP of FLI1 was observed a borderline P-value, suggesting that the C allele is associated with risk for LC [P = 0.036, OR = 95% 2:38 (1:07 to 5:32)]. The results obtained in this study reinforce the role of IL1-b and FLI-1 genes associated with resistance or susceptibility to LC. Key-words: Leishmaniasis, genetic polymorphism, IL-1b, MCP-1, FLI-1. xii LISTA DE ABREVIATURAS, SIGLAS E SÍMBOLOS ATP – Adenosina trifosfato; C3 – Convertase 3; CCC – Cardiomiopatia chagásica crônica; CCL2 – quimiocina CC ligante 2 (gene); CMSP - Células mononucleares do sangue periférico; CMI anti-Leishmania – Imunidade mediada por células anti-Leishmania; CR1 – Receptor de complemento 1; CR3 – Receptor de complemento 3; DLL1 – delta-like 1 (gene); DMSO – Dimetilsulfóxido; DNA – Ácido desoxirribonucleico; dNTP – desoxirribonucleotídeo trifosfato; EDTA – Ácido etilenodiamino tetra-acético; ETS – E26 Transformation-Specific; FLI-1 – friend leukemia virus integration 1 (gene); GM-CSF – Fator estimulador de colônias de macrófagos e granulócitos; GTP – Guanosina trifosfato; Hae III – Haemophilus aegyptius III; IFNGR1 – interferon gamma receptor 1 (gene); IFN-γ – Interferon gama; IL – Interleucina; IL-1b – Interleucina 1 beta (gene); LCD – Leishmaniose Cutâneo-Difusa; LCL – Leishmaniose Cutâneo-Localizada; LIT – Liver Infusion Triptose; LMC – Leishmaniose Mucocutânea; LTA – Leishmaniose Tegumentar Americana; LV – Leishmaniose Visceral; MCP1 – gene para proteína quimioatrativa de monócito tipo 1; M-CSF – Fator estimulador de colônia de macrófago; min – minuto; xiii mL – mililitro; mm – milímetro; mM – milimolar; µL – microlitro; ng – nanograma; NNN – Mc Neal, Novy e Nicolle; NRAMP1 – proteína 1 do macrófago associado à resistência natural (gene); PCR - Reação em cadeia da polimerase; pM – picomolar; RFLP – Análise clássica de comprimento do fragmento de restrição; RNA – Ácido ribonucleico; rpm – rotações por minuto; Sb+5 - Antimônio pentavalente; seg – segundos; SF – soro fisiológico; SFM – Sistema fagocítico mononuclear; SLA - Antígeno solúvel de Leishmania; SNP – polimorfismo de nucleotídeo único; TAE – tampão Tris-Acetato-EDTA; TCLE – Termo de consentimento livre e esclarecido; TGFB1 – fator transformador de crescimento beta 1 (gene); TGF-β – Fator transformador de crescimento beta; Th1 – T helper 1; Th2 – T helper 2; TLR4 – Receptor toll-like 4 (gene); TNF-α – Fator de necrose tumoral alfa; U – Unidades; UV – Ultravioleta; xiv LISTA DE TABELAS Tabela 1. Características clínicas da leishmaniose humana....................................... 8 Tabela 2. Informações gerais dos marcadores polimórficos genotipados para MCP-1, IL-1b e FLI-1. ............................................................................................................. 23 Tabela 3. Condições gerais de PCR usadas para o marcador MCP1_rs1024611. .. 23 Tabela 4. Condições gerais de PCR usadas para os marcadores IL1b_rs16944 e IL1b_rs1143634. ....................................................................................................... 24 Tabela 5. Condições gerais de PCR usadas para os marcadores FLI1_rs7930515 e FLI1_rs619521. ......................................................................................................... 24 Tabela 6. Condições gerais de PCR usadas para o marcador FLI1_rs145675082. . 24 Tabela 7. Condições gerais de PCR usadas para o marcador FLI1_rs531894 ........ 25 Tabela 8. Condições gerais usadas na reação de digestão. .................................... 26 Tabela 9. Condições gerais usadas na reação de PCR-RFLP. ................................ 29 Tabela 10. Características gerais da população caso-controle em estudo. .............. 30 Tabela 11. Comparação das distribuições das frequências genotípicas e alélicas para os marcadores MCP1_rs1024611, IL1b_rs1143634, FLI1_rs619521 e FLI1_rs531894. .................................................................................................................................. 34 Tabela 12. Comparação da distribuição das frequências genotípica, alélica e de dominância para o marcador IL1b_rs16944 .............................................................. 34 Tabela 13. Comparação da distribuição das frequências genotípica, alélica e de dominância para o marcador FLI1_rs7930515 .......................................................... 36 xv LISTA DE FIGURAS Figura 1. Morfologia das duas principais formas de Leishmania ................................ 1 Figura 2. Ciclo parasita-hospedeiro das leishmanioses ............................................. 2 Figura 3. Distribuição da leishmaniose cutânea no mundo. ....................................... 4 Figura 4. Padrão eletroforético da PCR-RFLP para os SNPs IL1-B +3953 C/T e IL1-B -511 C/T respectivamente. ........................................................................................ 31 Figura 5. Padrão eletroforético da PCR-RFLP para o SNP MCP1 -2518 A/G. ......... 31 Figura 6. Padrão eletroforético da PCR-RFLP para os SNPs FLI1_rs7930515, FLI1_rs619521 e FLI1_rs531894 respectivamente. .................................................. 32 Figura 7. Análise de desequilíbrio de ligação entre os SNPs dos genes IL-1b e FLI-1 respectivamente.. ...................................................................................................... 32 xvi SUMÁRIO 1. INTRODUÇÃO ..................................................................................................... 1 1.1. Leishmaniose – Aspectos Gerais ......................................................................... 1 1.2. Leishmaniose Tegumentar Americana ................................................................. 6 1.3. Imunopatogênese da leishmaniose ...................................................................... 8 1.4. O Modelo experimental murino para leishmaniose ............................................ 11 1.5. Fatores genéticos na leishmaniose .................................................................... 13 1.5.1. IL-1b ................................................................................................................ 14 1.5.2. FLI-1 ................................................................................................................ 16 1.5.3. MCP-1 ............................................................................................................. 16 2. OBJETIVOS ....................................................................................................... 18 2.1. Objetivo Geral .................................................................................................... 18 2.2. Objetivos Específicos ......................................................................................... 18 3. MATERIAL E MÉTODOS ................................................................................... 19 3.1. Delineamento do estudo..................................................................................... 19 3.2. Área de estudo ................................................................................................... 19 3.3. População estudada ........................................................................................... 19 3.3.1. Definição de Caso ........................................................................................... 19 3.3.2. Recrutamento da população ........................................................................... 20 3.3.3. Grupo caso ...................................................................................................... 20 3.3.4. Grupo controle................................................................................................. 20 3.4. Coleta do material biológico ............................................................................... 21 3.5. Extração de DNA ................................................................................................ 21 3.6. Genotipagem ...................................................................................................... 22 3.6.1. PCR ................................................................................................................. 23 3.6.2.Digestão ........................................................................................................... 25 3.7. Análise estatística .............................................................................................. 27 3.8. Aspectos Éticos: ................................................................................................. 27 3.9. Suporte Financeiro: ............................................................................................ 27 xvii 4. RESULTADOS ................................................................................................... 30 4.1. Características Gerais da População em Estudo ............................................... 30 4.2. Genotipagem, HWE e DL ................................................................................... 31 4.2.1 Análise dos marcadores MCP1_rs1024611, IL1b_rs1143634, FLI1_rs619521 e FLI1_rs531894 .......................................................................................................... 33 4.2.2. Análise do marcador IL1b_rs16944 ................................................................. 33 4.2.3. Análise do marcador FLI1_rs7930515............................................................. 33 5. DISCUSSÃO ...................................................................................................... 37 5.1. MCP-1 ................................................................................................................ 38 5.2 FLI-1 .................................................................................................................... 39 5.3 IL-1b .................................................................................................................... 40 6. CONCLUSÃO ..................................................................................................... 43 7. REFERÊNCIAS BIBLIOGRÁFICAS .................................................................. 44 8. ANEXOS............................................................................................................. 54 Anexo A – Fluxograma do Estudo ............................................................................. 54 Anexo B – Termo de Consentimento Livre e Esclarecido ......................................... 55 Anexo C – Questionário ............................................................................................ 59 Anexo D – Protocolo de Extração ............................................................................. 60 Anexo E – Minuta de Artigo ....................................................................................... 61 1 1. INTRODUÇÃO 1.1 Leishmaniose – Aspectos Gerais As leishmanioses são consideradas um grande problema de saúde pública, representando um grupo de doenças com importante espectro clínico e diversidade epidemiológica (1,2). São uma das mais diversas e complexas doenças transmitidas por vetores, sendo causadas por um protozoário intracelular obrigatório pertencente ao gênero Leishmania (3). Leishmania é um protozoário pertencente à família Trypanosomatidae, ordem Kinetoplastidae, gênero Leishmania, subgêneros Leishmania e Viannia (4). Sete espécies de Leishmania têm sido associadas como agentes causadores da Leishmaniose Tegumentar Americana (LTA) no Brasil: Leishmania (Viannia) braziliensis, Leishmania (Viannia) guyanensis, Leishmania (Viannia) lainsoni, Leishmania (Viannia) naiffi, Leishmania (Viannia) shawi, Leishmania (Viannia) lindenberg, e Leishmania (Leishmania) amazonensis. Contudo, L. (V.) braziliensis é o principal agente etiológico da LTA no Brasil (5). Leishmania apresenta-se com duas formas principais: uma flagelada ou promastigota, encontrada no tubo digestivo do vetor, e outra aflagelada ou amastigota, observada nos tecidos dos hospedeiros vertebrados como parasita intracelular obrigatório das células do sistema fagocitário mononuclear, conforme ilustrado na figura 1 (1). Figura 1. Morfologia das duas principais formas de Leishmania: a) Forma flagelada ou promastigota do parasita. b) Forma aflagelada ou amastigota. Fonte: Ministério da Saúde, 2010. 2 O flebotomíneo responsável pela transmissão do parasito da LTA na América pertence à subfamília Phlebotominae e gênero Lutzomyia (4). No ciclo epidemiológico da LTA (esquematizado na figura 2), o flebotomíneo representa o elo entre os reservatórios e o homem, que se comporta apenas como hospedeiro acidental da Leishmania. Este parasito é dimórfico, assim, ao picar os reservatórios infectados, as fêmeas dos flebotomíneos adquirem as formas amastigotas do protozoário, que rapidamente se transformam em formas promastigotas no intestino dos vetores. Essas formas dividem-se ativamente e no novo repasto, são injetadas na pele do hospedeiro. As promastigotas são as formas infectantes do parasito que interagem com as células do sistema fagocitário mononuclear, perdem o flagelo e, sob a forma amastigota, passam a se multiplicar no interior das células, sendo a infecção combatida pelas defesas do hospedeiro, evoluindo para a cura espontânea; ou evoluindo para uma das diversas manifestações clínicas da leishmaniose (6,7). Figura 2. Ciclo parasita-hospedeiro das leishmanioses Fonte: http://dc236.4shared.com/doc/FeNBLQeC/preview.html As formas clínicas da leishmaniose são diversas, sendo descritas em: leishmaniose visceral (LV) - geralmente fatal quando não tratada; leishmaniose mucocutânea (LMC) - doença mutilante; leishmaniose cutâneo-difusa (LCD) - doença de longa duração devido à deficiência na resposta imune celular; e leishmaniose 3 cutânea (LC) que é incapacitante quando as lesões são múltiplas (2,8). Segundo estudo realizado por Toleto et al, 2013, a LC afetou a qualidade de vida de todos os pacientes com a doença, com 40% desses indivíduos sendo gravemente afetados LC. Desse modo, puderam concluir que a LC teve um impacto negativo de moderado à alto na qualidade de vida dos pacientes (9). A cada ano, estima-se que 0,7 – 1,2 milhões de crianças e adultos desenvolvem a forma sintomática da leishmaniose (cutânea 0,7-1,2 milhões; visceral 0,2-0,4 milhões), e ainda, que 350 milhões de pessoas estejam expostas ao risco. A incidência da doença é ainda mais relevante quando as infecções assintomáticas são incluídas (1,2,10). Nas regiões onde a LTA ocorre de forma endêmica, as condições socioeconômicas desfavoráveis da população facilitam uma maior exposição do indivíduo ao vetor, favorecendo o desenvolvimento da doença. Isso explica, por exemplo, a maior incidência da doença em homens brasileiros, trabalhadores rurais ou exploradores de mata (4). Assim, Teles et al., 2013 observaram que 80% dos pacientes estavam envolvidos com atividades que exigiam a permanência na floresta (caça, pesca e exploração de madeira), evidenciando a natureza da transmissão zoonótica da doença (11). As leishmanioses são consideradas endêmicas em noventa e oito países. Os dez países com a contagem mais alta de casos estimados são: Afeganistão, Algéria, Colombia, Costa Rica, Norte do Sudão, Brasil, Irã, Peru, Etiópia e Síria, que juntos correspondem a 70 – 75% da incidência global estimada de casos de LC, conforme esquematizado na figura 3 (2,3,8,10). A LTA é de ocorrência antiga no Brasil, e apresenta-se como uma doença com diversidade de agentes, de reservatórios e de vetores que apresenta diferentes padrões de transmissão e um conhecimento ainda limitado sobre alguns aspectos, o que a torna de difícil controle (1,5). A partir do início da década de 1980, foram notificados no Brasil casos de LTA em 19 estados e, em 2003, todos os estados registraram autoctonia. A população do Amazonas destaca-se como uma das mais acometidas por LTA no país (12). 4 Figura 3. Distribuição da Leishmaniose cutânea no mundo. Fonte: http://www.who.int/en/ A partir da década de 90, o Ministério da Saúde notificou uma média anual de 32 mil novos casos de LTA. Analisando-se os dados pertinentes em 2003, verificouse a seguinte situação: a Região Norte notificou aproximadamente 45% dos casos, predominando os estados do Pará, Amazonas e Rondônia; a Região Nordeste, 26% dos casos, principalmente no Maranhão, Bahia e Ceara; a Região Centro-Oeste, 15% dos casos, com maior frequência em Mato Grosso; a Região Sudeste, 11% dos casos, predominantemente em Minas Gerais; e a Região Sul, 3,0%, destacando-se o Paraná (6). Em 2011, de acordo com o Ministério da Saúde, foram registrados, no Brasil, 15.731 casos de LTA. A região Norte apresentou novamente o maior número de notificações, 7.362 casos, seguida das regiões Nordeste (5.234), Centro-Oeste (1.732), Sudeste (986) e Sul (324). Dentre os 7.362 casos registrados na região Norte, 2.971 casos foram notificados no Estado do Pará, 1.752 no Amazonas, 781 no Acre, 704 em Rondônia, 504 no Amapá, 368 em Roraima e 282 no Tocantins (13). A real prevalência das diferentes leishmanioses no continente americano é difícil de ser estabelecida em vista das subnotificações, diagnósticos incorretos, infecções inaparentes, variações de resposta do hospedeiro e multiplicidade de agentes etiológicos envolvidos (6). 5 No último ano, a Fundação de Medicina Tropical Dr. Heitor Vieira Dourado (FMTHVD), centro de referência para o tratamento de LTA no estado do Amazonas, registrou 679 casos da doença, sendo 667 dos casos relacionados com a forma cutânea e 12 casos com a forma mucosa da doença. Registrando nos últimos 5 anos aproximadamente 5 mil casos de LTA (14). Quanto ao diagnóstico laboratorial para as leishmanioses, existem muitos métodos de diagnóstico, incluindo parasitológico, imunológico, molecular e os que utilizam animais de laboratório, todos usados na detecção do parasita (15). O exame parasitológico é baseado na demonstração do parasito por meio de exames diretos e indiretos (1,15). Quanto ao arsenal imunológico, pode-se destacar o teste intradérmico (intradermorreação de Montenegro ou da leishmanina) e testes sorológicos (1,16). Tem-se ainda o diagnóstico molecular que é atualmente utilizado para a detecção de micro-organismos patogênicos e baseia-se no emprego de sondas de DNA e/ou RNA espécie especificas e na reação em cadeia de uma polimerase de DNA para aumento de sequências nucleotídicas especificas presentes em amostras clínicas (15–18). Existem muitos estudos na literatura usando a PCR como técnica alternativa com resultados promissores para melhorar o diagnóstico da leishmaniose (19–22). Em se tratando de arsenal terapêutico disponível para tratamento da leishmaniose, este é bastante precário, existindo, até o presente momento, dois grupos de medicamentos em uso: os antimoniais e os não-antimoniais. O tratamento básico da doença consiste na administração de estibogluconato sódico (Pentostan®), antimoniato de N-metil-glucamina (comercializado no Brasil como Glucantime®), ambos drogas de primeira geração e pentamidina ou anfotericina B, ambos de segunda geração (23,24). Em um estudo clínico randomizado, Neves et al., 2011 constataram que a pentamidina e o antimonial apresentam eficácias similares e não satisfatórias para o tratamento da LTA ocasionada por L.(V.) guyanensis. Os baixos resultados de eficácia do antimonial e pentamidina, bem como as dificuldades operacionais relacionadas à anfotericina B, evidenciam a necessidade urgente de novas opções terapêuticas para o tratamento da LTA (25). 6 A miltefosina foi a primeira droga de uso oral utilizada no tratamento da leishmaniose visceral (LV). Existem poucos efeitos adversos, tais como vômito e diarréia. Em relação à LTA, estudos utilizando a miltefosina no tratamento da forma cutânea da doença resultaram em boa eficácia contra L (V.) panamensis, mas para L (V.) braziliensis, não houve eficiência adequada (24). No cenário atual de combate à leishmaniose, nota-se a existência de importantes ferramentas que auxiliam na detecção e combate ao parasita. Podendose citar o diagnóstico, com a visualização de parasitas em tecidos e/ou sorologia através da técnica de exame direto; cultura e detecção do DNA do parasita; além do arsenal terapêutico, com destaque para o tratamento convencional com antimonial pentavalente; contudo, a resistência da leishmaniose em alguns países tem gerado novas abordagens de tratamento, com inserção de novas drogas, como anfotericina B, paromicina injetável e miltefosina oral (1,2,16). Apesar de consideráveis avanços no diagnóstico, tratamento e pesquisa científica básica, a leishmaniose ainda é considerada uma doença negligenciada e atrelada à pobreza. Obstáculos correntes encontrados na prevenção e manejo adequado incluem controle inadequado do vetor, inexistência de vacinas e dificuldade em desenvolver drogas mais acessíveis (2). 1.2 Leishmaniose Tegumentar Americana Por leishmaniose tegumentar entende-se um conjunto de enfermidades causadas por várias espécies de protozoários do gênero Leishmania, que acometem a pele e/ou mucosas do homem, e de diferentes espécies de animais silvestres e domésticos, com casos registrados mundialmente, destacando-se o Continente Americano, onde há registro de casos desde o extremo sul dos Estados Unidos até o norte da Argentina, com exceção do Chile e Uruguai. Na Amazônia Brasileira, a LTA é considerada uma infecção zoonótica primária de mamíferos silvestres e secundariamente de animais domésticos, em que o parasita é transmitido entre os animais e o homem pela picada das fêmeas de diversas espécies de flebótomos (Diptera, Psychodidae, Phlebotominae) do gênero Lutzomyia. A infecção caracteriza- 7 se pelo parasitismo das células do sistema fagocitário mononuclear (SFM) da derme e das mucosas do hospedeiro vertebrado (monócitos, histiócitos e macrófagos) (1,6,26,27). No Brasil, há várias formas clínico-epidemiológicas relacionadas aos diferentes subgêneros e espécies do parasita (28): • Leishmania (Viannia) guyanensis: limitada ao norte da Bacia Amazônica (Amapá, Roraima, Amazonas e Pará) e Guianas. É o agente predominante causador da doença no estado do Amazonas, produzindo lesões menores e mais numerosas, frequentemente localizadas acima da cintura. O acometimento mucoso é pouco frequente. • Leishmania (Viannia) braziliensis: ampla distribuição (sul do Pará ao Nordeste), centro-sul do país e Amazônia oriental. A doença caracteriza-se por úlcera cutânea (única ou múltipla) que pode evoluir para o acometimento mucoso (oro ou nasofaringe) por disseminação hematogênica; • Leishmania (Leishmania) amazonensis: distribuída pelas florestas da Amazônia, Nordeste (Bahia), Sudeste (Minas Gerais e São Paulo) e Centro-Oeste (Goiás). A doença humana é relativamente rara, podendo se manifestar como lesão ulcerada (geralmente única) ou quadro de leishmaniose cutâneo-difusa. Dependendo da espécie de Leishmania infectante e da resposta imune mediada por células da pessoa infectada, o indivíduo pode desenvolver um espectro de formas clínicas da doença, convencionalmente conhecidas como leishmaniose cutânea localizada (LCL) e leishmaniose mucocutânea (LMC), representando o polo responsivo, e leishmaniose cutâneo-difusa (LCD) que representa o polo não responsivo (26,29). As principais características clínicas da leishmaniose estão listadas na tabela 1. 8 Tabela 1. Características clínicas da leishmaniose humana. CARACTERÍSTICAS MUCOCUTÂNEA CUTÂNEA VISCERAL Lesão única (ocasionalmente Lesões Destruição ulcerativa do um pequeno número) ulcerada septo nasal com bordas elevadas e centro Órgãos internos necrótico Histopatologia e número de parasitas Nível de anticorpos antiLeishmania CMI anti-Leishmania (testes in vivo e in vitro) Acentuada proliferação de Reação granulomatosa Resposta inflamatória crônica com pouquíssimos com moderado número de parasitas parasitas Baixo Baixo Alto Fortemente positivo Positivo Negativo macrófagos com intenso parasitismo nos órgãos hematopoiéticos Fonte: Adaptado de Oliveira et al, 2004. 1.3 Imunopatogênese da leishmaniose A patogenia da LTA é fortemente influenciada pelo perfil imunogenético do homem e ainda, pela virulência da espécie de Leishmania infectante. Como resultado da interação entre as diferentes espécies de Leishmania e os mecanismos da resposta imune do homem ocorre um espectro de manifestações clínicas, histopatológicas e imunopatológicas (30). Infecções por Leishmania podem permanecer completamente assintomáticas. Esta observação chama a atenção para fatores do hospedeiro na susceptibilidade e expressão da doença - idade, estado nutricional e a eficácia das respostas imunes inata e adquirida dependentes de células T. Este último pode, por sua vez, ser afetado por polimorfismos imunogenéticos (2). Ao que parece, em humanos, o resultado da doença é influenciado pelo balanço entre as respostas Th1, Th2 e também Th17, além da influência de fatores genéticos do hospedeiro, tamanho do inóculo e a cepa do parasita. (31,32). A resposta imune tem um papel importante na patogênese da leishmaniose tegumentar. Na leishmaniose cutânea e na leishmaniose mucosa, uma intensa resposta Th1, com alta produção de IFN-γ e TNF-α é observada, juntamente com IL17, produzida pelas células Th17, responsável pela amplificação da resposta 9 inflamatória. Esse tipo de resposta inflamatória, apesar de estar associada ao controle da multiplicação e disseminação do parasita, se não modulada, pode causar dano tecidual. Por outro lado, a diminuição ou ausência das respostas Th1/Th17 e presença de citocinas regulatórias como IL-10, observada em indivíduos com leishmaniose disseminada e leishmaniose cutânea difusa, favorece a multiplicação e disseminação do parasita (32,33). A infecção humana, pelas espécies de Leishmania que causam a LTA, tem seu início logo após a inoculação das formas promastigotas do parasita na pele, o que acontece durante a hematofagia pelas espécies de flebotomíneos vetores. A partir desse momento, inicia-se um processo de escape do parasita frente às defesas do organismo, o qual, quando vencido pelo parasita, resultará na sua fagocitose pelas células do sistema fagocítico mononuclear (SFM), principalmente o macrófago (30). As células T “helper” CD4+ tem uma função central no sistema imune, promovendo respostas adaptativas adequadas a patógenos específicos. De acordo com as citocinas que produzem após estimulação antigênica, estas células podem ser separadas em subpopulações, das quais é de interesse mencionar: T “helper” 1 (Th1), T “helper” 2 (Th2) e T ”helper” 17(Th17). As células Th1 produzem IFN-γ e IL-12 e estão associadas à proteção contra os patógenos intracelulares, como as leishmanias; células Th17 produzem IL-17A e IL-17F, em adição a outras citocinas tais como IL-21 e IL-22. IL-17 realiza uma importante função no recrutamento de neutrófilos, e uma vez que estas células tem um papel importante na infecção por Leishmania, pode-se dizer que células Th17 tem papel significativo nessa doença. Células Th2 produzem interleucina IL-4, IL-5 e IL-13 que estão envolvidas nos processos alérgicos e na proteção contra agentes extracelulares. No caso das infecções por micro-organismos intracelulares, a ativação das células Th2 leva ao agravamento (1,32,34). Assim, a resistência do hospedeiro está associada à ativação seletiva e diferenciação de células efetoras T “helper” CD4+ (Th1), as quais secretam um padrão de citocinas específicas, tendo como principais indutores dessa resposta IFN-γ e IL12, conhecidos como citocinas guia. IL-4 é responsável pela indução da resposta de 10 células T CD4+ (Th2), além das citocinas IL-23, IL-1, IL-6 e TGF-β para células Th17 (7,34). Considerando-se que a Leishmania é um parasita intracelular obrigatório, a ativação do sistema complemento pode beneficiá-lo, pois ao serem gerados fragmentos da cascata (C3 e C3bi) que se ligam aos receptores dos macrófagos (CR1 e CR3) e também se depositam na parede do parasito, tem-se facilitada sua fagocitose. Além disso, sua entrada, utilizando esses receptores, promove baixo metabolismo oxidativo no macrófago, o que favorece a continuidade do seu ciclo biológico (6,35). A fagocitose dos organismos patogênicos induz os macrófagos a produzir citocinas que controlam vários aspectos da defesa do hospedeiro, incluindo fatores quimiotáticos, tais como as quimiocinas, fatores de ativação tais como TNF-α, IL-12, e IL-1, estimulantes de fase aguda, tais como IL-6, fatores moduladores, tais como a IL-10, e citocinas estimuladoras da hematopoiese, tais como M-CSF e GM-CSF (35). A modulação da resposta imune celular é definida com base no tipo de interação entre a espécie de Leishmania e o perfil imunogenético do hospedeiro, resultando no desenvolvimento de diferentes formas clínicas de leishmaniose. Na leishmaniose cutânea e leishmaniose mucosa, existe uma forte resposta Th1, que controla a multiplicação do parasita, porém há lesão tecidual. Enquanto que, na leishmaniose disseminada e na leishmaniose cutânea difusa, a diminuição ou ausência da resposta Th1 favorece a multiplicação e disseminação da Leishmania (33). Células mononucleares do sangue periférico (CMSP) de pacientes com leishmaniose cutânea secretam altos níveis de IFN-γ e TNF-α e baixos níveis de IL-5 e IL-10 quando estimuladas com antígeno solúvel de Leishmania (SLA), sendo a célula T CD4+ o tipo celular que contribui com a maior parte da produção de IFN-γ tanto no sangue periférico quanto nos tecidos, embora haja participação de células T CD8+ na produção dessa citocina (33). Nas formas cutaneomucosa e mucosa, associadas à forte resposta de hipersensibilidade e proliferação de linfócitos, o perfil de linfócitos T CD8 e CD4 no infiltrado das lesões tem mostrado predomínio de CD4. Desse modo, parece 11 significativo que na forma LCM/LM tenha sido identificado um perfil de células CD4 maior que nas outras formas da LTA, sugerindo a possibilidade de níveis significativos de IFN-γ e TNF-α nessas formas, já que estas citocinas estão fortemente associadas à resposta de hipersensibilidade (30). Em um estudo realizado por Gonzalez-Lombana et al., 2013 foram observados que níveis elevados de IFN-γ promoveram uma infiltração de monócitos aumentada, enquanto IL-17 contribuiu para o aumento de neutrófilos. Contudo, foi constatado que IFN-γ não contribuiu para o agravamento da patologia, ao invés, regulou a resposta de IL-17. Assim, o bloqueio de IFN- γ levou a um aumento significativo de IL-17, neutrófilos e agravamento da doença. Dessa maneira, nota-se que a resposta imune inflamatória é critica no controle dos parasitas da leishmaniose, mas também, se não balanceada, promove a exacerbação da doença (36). Pacientes com leishmaniose cutânea difusa apresentam negatividade do teste de Montenegro, baixa ou ausência de produção de IFN-γ para antígeno de Leishmania, indicando um forte bloqueio da resposta imune celular, impossibilitando aos pacientes o controle da infecção. Células mononucleares de pacientes com leishmaniose cutânea difusa expressam grandes quantidades de IL-10 e IL- 4 e baixas quantidades de IFN-γ durante a fase ativa da doença (30,33). 1.4 O Modelo experimental murino para leishmaniose Modelos experimentais de infecção por Leishmania são modelos bem estudados e apropriados para o entendimento da doença (37). Dentre os modelos existentes, o camundongo oferece vantagens particulares para estudos de biologia e doença humana (38): i) O camundongo, em seu desenvolvimento, plano corpóreo, fisiologia, comportamento e doenças, pode apresentar aspectos que são comuns aos seres humanos; ii) A quase totalidade (99%) dos genes do camundongo tem homólogos em seres humanos; iii) O genoma do camundongo suporta mutagênese direcionada e específica, por recombinação homóloga em células estaminais embrionárias, permitindo que os genes sejam alterados de forma eficiente. 12 A maior parte do que se conhece sobre a resposta de um indivíduo à infecção por Leishmania e seus mecanismos imunológicos provêm de estudos em camundongos. Algumas linhagens puras de camundongos são susceptíveis à infecção por Leishmania, enquanto outras são extremamente resistentes (3,29). Infecção subcutânea por L. major é um dos modelos mais bem estudados. Acerca desse modelo, características distintas do espectro clínico da leishmaniose humana são reproduzidas em linhagens puras de camundongos (29). Camundongos da linhagem BALB/c são altamente susceptíveis, pois quando infectados, estes desenvolvem grandes úlceras de pele, que expandem e metastizam, levando à morte. Em contraste, camundongos das linhagens C3H/He, CBA, C57BL/6 e 129Sv/Ev são resistentes à infecção por L. major, desenvolvendo pequenas lesões que curam em 10-12 semanas, e também são resistentes a reinfecção (29). Um estudo realizado por Hezarjaribi et al., 2013 demostrou através do modelo experimental murino que IL-22 leva a um aumento na produção de IFN-γ e diminuição na produção de IL-4 e que portanto tem um papel importante na estimulação da resposta do tipo Th1, resultando na cura das lesões (39). A relevância do balanço Th1/Th2 na regulação do desfecho da doença tem sido minuciosamente investigada (29). A análise da resposta imune em camundongos BALB/c tem mostrado que estes respondem de maneira diferente, com uma variação nas respostas Th1 e Th2, dependendo da espécie pelo qual este é infectado, L. major ou L. braziliensis (40). A resistência está ligada a resposta do tipo Th1, com produção de IL-12, IFN-γ e inibição da produção de citocinas Th2, enquanto que a susceptibilidade está relacionada com a predominância da resposta Th2, determinada pela presença de IL4. Citocinas Th2 levam ao desenvolvimento de lesões graves em camundongos infectados com L. major, provavelmente inativando células infectadas (29). Dependendo da espécie pelo qual o camundongo é infectado, a análise da resposta imune mostra que este responde de maneira diferente, com uma variação nas respostas Th1, Th2 e Th17 (40). Em camundongos infectados por L. (L.) amazonensis, a definição da progressão e desfecho da infecção é feita com base no 13 conhecimento da genética dos camundongos, além de fatores adicionais como tamanho do inóculo, sítio de inoculação, cepa de Leishmania usada e sua fonte de isolamento, que também podem influenciar nessas características (37). Em estudo realizado por Courret et al., 2003, um modelo para simular, em longo prazo, o parasitismo natural foi proposto, onde baixas doses (10-1000 parasitas) de promastigotas metacíclicas de L. (L.) amazonensis foram inoculadas na orelha de camundongos BALB/c. Os resultados mostraram que as manifestações cutâneas da lesão dependem do tamanho do inóculo, onde muito raramente, animais infectados com apenas 10 parasitas desenvolveram lesão; a maioria dos animais infectados com 100 parasitas e todos com 1000 parasitas desenvolveram lesão progressiva (41). 1.5 Fatores genéticos na leishmaniose Em genética, doenças complexas são aquelas que não apresentam padrão de herança mendeliana clássica, em que há perfeita correlação entre genótipo e fenótipo. A ocorrência dessas doenças é controlada por componente genético, resultado da ação integrada de um número variado de genes, associado a fatores ambientais, socioeconômicos, culturais, além de fatores inerentes ao hospedeiro, como estado nutricional, idade, gravidez, infecção por HIV, etc. (42,43) O fato de alguns indivíduos não apresentarem nenhum sinal ou sintoma de infecção, enquanto outros desenvolvem formas crônicas ainda não foi claramente elucidado. Acredita-se que a genética do hospedeiro, fatores ambientais ou do parasito podem estar envolvidos na susceptibilidade à doença (44). A resposta imune e o conhecimento da genética do hospedeiro podem ser importantes no entendimento da patogênese da doença. Uma maneira de explorar o papel de moléculas pró e anti-inflamatórias em humanos é determinar se polimorfismos genéticos afetam a expressão ou função de proteínas, e se estas estão associadas com o desfecho da doença (45). Por exemplo, um estudo realizado em Danghã, uma área endêmica para leishmaniose cutânea no norte do Irã demonstrou que mutações no gene TLR4 14 humano estão associadas com um risco aumentado de desenvolver leishmaniose cutânea, podendo levar a um aumento da susceptibilidade e gravidade na infecção por L. major (44). Variantes alélicas de NRAMP1 estão fortemente associadas com leishmaniose visceral, indicando que este é um importante gene de suscetibilidade em humanos (46). Estudos sugerem que as variações genéticas deste gene afetam a suscetibilidade na leishmaniose visceral (45–49). A análise de polimorfismos de TNFα em uma população da Venezuela mostrou que a susceptibilidade para a forma cutâneo-mucosa da doença pode ser diretamente associada com polimorfismos do gene TNFα, evidenciando um alto risco relativo de 7,5 (P<0,001). (51) Alguns estudos demonstraram associação do polimorfismo dos genes IL-6 e CCL2 com o aumento da suscetibilidade para a forma mucosa da leishmaniose (45,52). Variantes do gene DLL1, localizado no cromossomo 6q27, apresentaram-se associadas com a susceptibilidade para LV (53), bem como os polimorfismos do gene TGFB1, uma citocina com uma função biologicamente relevante na história natural da doença, que pode contribuir para a susceptibilidade global na infecção por leishmania per se e para a gravidade da doença clínica na leishmaniose visceral (54). Ainda, polimorfismo no gene IL-4 foi associado na susceptibilidade para leishmaniose visceral, enquanto que IFNGR1 está especificamente relacionado com a susceptibilidade à leishmaniose dermal pós-kalazar (46,55). 1.5.1 IL-1b Entre as citocinas pro-inflamatórias do tipo Th1, interleucina 1 (IL-1) tem papel chave na resposta inflamatória com efeitos pleiotrópicos em muitas células e vias de sinalização, tendo um papel importante no desenvolvimento da patologia. IL-1 consiste de um grupo de três moléculas nomeadas IL-1 alfa, IL-1 beta e IL-1 receptor antagonista (56). 15 IL-1b é classificado como um dos mediadores solúveis mais importantes da inflamação, sendo produzido principalmente por monócitos, macrófagos e células dendríticas e medeia uma ampla gama de reações envolvidas na resposta de fase aguda (57). Polimorfismos genéticos em IL-1b estão associados com a susceptibilidade a doenças autoimunes graves, inflamatórias e infecciosas tais como HIV, TB, gastrite crônica associada com Helicobacter pylori, esclerose múltipla, lupus eritematoso sistêmico, malária e leishmaniose (58–63). Recentemente, um estudo realizado no México sugeriu que a IL-1b possivelmente tem um papel chave na determinação da gravidade da doença em pacientes com leishmaniose cutânea difusa (63). IL-1b é uma citocina com atividade pirogênica que é normalmente ativada em resposta a uma infecção ou injúria (64). Polimorfismo em IL-1b representa uma variável que influencia o risco de desenvolver a doença, em pacientes infectados com o parasita (63). Os inflamassomas tem importante papel na expressão de IL-1b, pois estes são responsáveis por promover a ativação dessa interleucina através de diversos estímulos durante as infecções por micro-organismos (65). Resultados de Lima-Junior et al., 2013 sugerem que independentemente do “background” genético do camundongo ou das formas parasitárias usadas na infecção (parasitas mortos, atenuados ou vivos), o inflamassoma é de grande importância no controle da infecção parasitária em LC (66). NALP3, que pertence a uma larga família de receptores NOD-like intracelulares, associa-se com outras proteínas por oligomerização para formar NALP3-inflamassoma, que é responsável por ativar caspase-1, enzima responsável por clivar precursor IL1-b inativo em IL1-b ativo (65–67). Lima-Junior et al., 2013 observaram níveis significativos na produção de IL-1b por macrófagos derivados de medula óssea obtidos de camundongos C57BL/6 em resposta a infecção por L. amazonensis, mas o mesmo não foi observado em camundongos que possuíam a deleção do gene produtor de caspase-1. Assim, 16 puderam afirmar que a secreção de IL-1b é dependente da ativação de caspase-1 (66). 1.5.2 FLI-1 Outro importante gene envolvido no controle da susceptibilidade da leishmaniose e que apresenta participação no processo de cura das lesões é o FLI1. Pertencente à família ETs de fatores de transcrição, é preferencialmente expresso em linhagens de células hematopoiéticas e células endoteliais vasculares (68). Resultados apresentados por Asano et al., 2009 estabelecem o gene FLI1 como um regulador chave na homeostase da expressão de colágeno na pele, e, portanto, pode atuar na via de cicatrização das lesões. Além disso, o estudo demonstra que o domínio C-terminal do FLI-1 tem um papel essencial no controle da expressão de colágeno in vivo (69). Ainda, Castellucci et al., 2011 demonstraram associação entre polimorfismos em FLI-1 e susceptibilidade para leishmaniose cutânea causada por L. braziliensis (70). 1.5.3 MCP-1 Uma maneira de explorar o papel das quimiocinas em humanos é determinar se polimorfismos que afetam expressão/função estão associados com o desfecho da doença. Assim, Ramasawmy et al., 2010 demonstraram, através de estudo casocontrole, uma associação entre SNP (polimorfismo de nucleotídeo único) em MCP-1 e susceptibilidade para leishmaniose mucosa por L. braziliensis. Os dados apresentados pelos autores fornecem evidências de base populacional e familiar para uma associação entre o alelo G do SNP na região promotora CCL2 -2518 pb e susceptibilidade para LM. Esses dados foram correlacionados com elevados níveis de MCP-1 no plasma de indivíduos homozigotos para o alelo G, e com alta liberação de MCP-1 por macrófagos estimulados e não estimulados (45). Ainda, dados gerados por De Moura et al., 2005 sugerem que a capacidade leishmanicida de MCP-1 contribui para a cura das lesões causadas por L. braziliensis 17 (71). Também, Rovin et al., 1999 afirmam que variações genéticas ocorrem em regiões reguladoras dos genes de quimiocina, e que tais polimorfismos afetam a transcrição desses genes em resposta a estímulos inflamatórios. Esses polimorfismos em MCP-1 podem, em parte, determinar a gravidade da inflamação nos órgãos em doenças onde a infiltração leucocitária nos tecidos é dependente de MCP-1 (72). Estudo realizado em uma população recrutada no Instituto do Coração no Hospital das Clínicas em São Paulo demonstrou que a variante MCP-1 associada com um baixo nível de transcrição de MCP-1 se comporta como um modificador genético de desfecho clínico para infecção por T. cruzi, e os indivíduos com o genótipo MCP-1 -2518 AA tem um risco quatro vezes maior de desenvolver cardiomiopatia chagásica crônica (CCC) do que aqueles sem esse genótipo, sugerindo que polimorfismos em MCP-1 podem desempenhar papel importante na susceptibilidade a CCC, dependendo do genótipo apresentado por cada indivíduo (73). Diante do exposto, nota-se que a leishmaniose é uma doença infecciosa cuja susceptibilidade é em parte controlada geneticamente. A participação de variantes dos genes IL-1b, FLI-1 e MCP-1 no controle da suscetibilidade de populações humanas a doenças infecciosas causadas por patógenos intracelulares de interesse, tais como a leishmaniose, ainda precisa ser investigada. Dessa forma, entende-se que esforços no sentido de se elucidar os mecanismos moleculares de controle e interação patógeno/hospedeiro e os mecanismos de desenvolvimento da doença tendem a contribuir de forma crítica para um maior entendimento de sua patogênese, levando ao desenvolvimento de melhores estratégias de diagnóstico, tratamento e prevenção. 18 2. OBJETIVOS 2.1. Objetivo Geral Verificar o papel dos polimorfismos genéticos em IL-1b, FLI-1 e MCP-1 associados com leishmaniose cutânea em uma população caso-controle do estado do Amazonas, Brasil. 2.2. Objetivos Específicos - Determinar a frequência dos alelos e genótipos dos SNPs dos genes IL-1b, FLI1 e MCP-1 entre os pacientes com leishmaniose cutânea e indivíduos sem a doença; - Comparar as frequências encontradas dos SNPs e sua possível associação no controle da susceptibilidade ou resistência à LTA. 19 3. MATERIAL E MÉTODOS 3.1. Delineamento do estudo Trata-se de um estudo realizado em uma população caso-controle do estado do Amazonas, recrutada no período compreendido entre novembro de 2008 e dezembro de 2013. 3.2. Área de estudo Consiste de regiões endêmicas para leishmaniose localizadas nas proximidades do município de Manaus, especificamente das rodovias AM-010, que liga Manaus ao município de ltacoatiara; e BR-174, que liga Manaus a Boa Vista, capital do estado de Roraima. Estas regiões endêmicas são áreas de floresta tropical que ao longo dos anos sofreram desmatamento, dando lugar a assentamentos populacionais, cuja principal fonte de renda é a atividade agropecuária. Sendo assim, as principais atividades ocupacionais destes indivíduos e seu tipo de moradia, próximo às áreas de mata, tornaram essa população altamente exposta à infecção por Leishmania sp. 3.3. População estudada Foram recrutados pacientes de ambos os sexos com diagnóstico confirmado para leishmaniose cutânea provenientes da demanda espontânea do ambulatório de Dermatologia da FMT-HVD para compor o grupo caso; e indivíduos sem sinal ou histórico de leishmaniose, para compor o grupo controle, todos provenientes de áreas endêmicas para leishmaniose no estado do Amazonas. 3.3.1 Definição de Caso 20 A leishmaniose cutânea é definida como a presença de uma ou mais lesões ulceradas na pele, sem evidência clínica de comprometimento mucoso, além da detecção do parasito através do exame direto do escarificado da lesão. 3.3.2 Recrutamento da população No momento da inclusão dos indivíduos voluntários, foram explicados os detalhes e potenciais consequências do estudo e os procedimentos aos quais estes seriam submetidos em caso de concordância em participar. Todos os candidatos foram solicitados a ler o Termo de Consentimento Livre e Esclarecido (TCLE) (Anexo B), devidamente aprovado pelo(s) Comitê(s) de Ética (CEP) pertinente(s). Aos que concordaram em participar, foi assinado o TCLE correspondente ao seu caso específico, e aplicado questionário (Anexo C). Ao voluntário menor de idade, um formulário próprio foi apresentado e assinado por um dos pais ou guardião legal. Para o voluntário não alfabetizado, o TCLE foi lido e explicado e, em caso de aceite, sua impressão digital foi coletada. 3.3.3 Grupo de indivíduos com LC (caso) A população de estudo desse grupo foi recrutada exclusivamente no ambulatório de dermatologia da FMT-HVD por demanda espontânea. Foram admitidos apenas pacientes com leishmaniose cutânea que tiveram seu diagnóstico confirmado através do exame direto do escarificado da lesão realizado por técnicos microscopistas capacitados para este fim. Após o diagnóstico positivo de LTA, os pacientes que concordaram em participar do estudo, mediante assinatura do TCLE e aplicação de questionário (Anexos B e C, respectivamente) foram submetidos a uma coleta de 5 mL de sangue total. 3.3.4 Grupo de indivíduos sem a doença (controle) 21 Para compor este grupo, foram recrutados indivíduos sem história prévia ou manifestação da LTA, levando-se em consideração ausência de citratrizes, não exposição prévia ao tratamento e ausência de parentesco. Todos os indivíduos deste grupo foram provenientes de áreas endêmicas adjacentes às rodovias AM-010 e BR174, de onde procedeu a maioria dos indivíduos do grupo caso. A seleção dessas localidades foi realizada tendo como base a escolha de regiões com casos confirmados de leishmaniose, podendo-se destacar as principais: BR-174 - Ramal do Pau-Rosa e Ramal da Cooperativa e suas vicinais (Km 21); AM010 - Ramal Água Branca I (Km 32) e Ramal Água Branca II (Km 35), Ramal do Leão (Km 37). O recrutamento foi realizado por meio de visitas da equipe de investigadores a essas localidades no mesmo período em que foram recrutados os indivíduos doentes, percorrendo-se toda a extensão do ramal, com a finalidade de alcançar todas as moradias. 3.4. Coleta do material biológico Todos os indivíduos que concordaram em participar do estudo, foram submetidos a uma punção venosa para coleta de 5 mL de sangue periférico. As amostras de sangue foram coletadas em tubos tipo Vacutainer® contendo EDTA como anticoagulante, em condições ideais de assepsia. O sangue coletado foi centrifugado a 3.600 rpm durante 10 minutos para obtenção do creme leucocitário (“buffy-coat”) que foi posteriormente aspirado e transferido para um microtubo de 1,5 mL para extração do DNA genômico. O processamento das amostras deu-se no Laboratório de Pesquisa em Doenças Endêmicas (LPDE) da FMT-HVD. 3.5. Extração de DNA 22 O DNA genômico foi extraído a partir do creme leucocitário obtido conforme descrito anteriormente. As extrações realizadas até o ano de 2011 foram feitas com o kit comercial de extração de DNA (WIZARD®, Promega Corporation, EUA), de acordo com as instruções sugeridas pelo fabricante. As amostras obtidas a partir do ano de 2012 foram extraídas pela técnica de “salting-out”, conforme protocolo de Sambrook et al., 1989 com algumas modificações (Anexo D) (74). Após a extração de DNA, sua concentração e pureza foram determinadas em espectrofotômetro, através da razão das absorbâncias em 260nm e 280nm para leitura de DNA e proteína, respectivamente. Dessa forma, esta razão pode fornecer um parâmetro indicativo da qualidade do DNA, sendo a amostra considerada ideal quando a razão das absorbâncias apresenta-se na faixa de 1,8 a 2,2. Excesso de proteínas valores no inferiores material extraído a e 1,8, valores é indicado superiores por a 2,2 indicam excesso de solventes orgânicos. Sendo assim, as amotras extraídas que foram consideradas adequadas (através dos parâmetros de concentração e pureza) foram armazenadas em freezer à -80°C, localizados exclusivamente em laboratórios da FMT-HVD. 3.6. Genotipagem Os marcadores do tipo Polimorfismo de Nucleotídeo Único (do inglês, SNP; Single Nucleotide Polymorphism) dos genes MCP-1 e IL-1b foram selecionados utilizando-se material disponível na literatura e a partir de bancos de dados de domínio público para obtenção das sequências. Para os SNPs de FLI-1, os iniciadores (rs7930515, rs619521, rs145675082, rs531894) foram desenhados a partir de sequências disponíveis em base de dados do NCBI. A genotipagem foi realizada pela técnica de PCR-RFLP (Reação em Cadeia da Polimerase - Análise clássica de comprimento do fragmento de restrição) para descriminação alélica. 23 Tabela 2. Informações gerais dos marcadores polimórficos genotipados para MCP-1, IL-1b e FLI-1. CROMOSSOMO GENE / dbSNP 17 2 2 11 11 11 11 MCP-1 / rs1024611 IL-1b / rs1143634 IL-1b / rs16944 FLI-1 / rs531894 FLI-1 / rs145675082 FLI-1 / rs619521 FLI-1 / rs7930515 POSIÇÃO FÍSICA 34252769 112832813 112837290 128758861 128750108 128708262 128694899 VARIAÇÃO [G/A] [C/T] [T/C] [A/G] [C/T] [A/G] [A/C] 3.6.1. PCR Foi realizada a amplificação das sequências gênicas que incorporam as regiões de mutação utilizando-se a técnica de PCR Convencional, de modo que cada PCR foi realizada num volume final de 25 µL contendo 50 ng de DNA genômico, iniciadores senso e antisenso, dNTP, taq polimerase e MgCl2 de acordo com as tabelas 3 a 7, que descrevem especificamente as condições de concentração e temperatura para cada marcador utilizado. Tabela 3. Condições gerais de PCR usadas para o marcador MCP1_rs1024611 (MCP-1 -2518). Reagentes Concentração Ciclo Tampão de PCR 1x 95ºC – 5’ MgCl2 1,6 mM 15x dNTP 40 µM 95ºC – 20’’ 65ºC – 20’’ 72ºC – 45’’ Iniciadores 0,2 µM 25x Taq Polimerase 2,0 U 95ºC – 20’’ 55ºC – 20’’ 72ºC – 45’’ Volume final 25 µL 72ºC – 7’ 4ºC – ∞ 24 Tabela 4. Condições gerais de PCR usadas para os marcadores IL1b_rs16944 (IL1b -511) e IL1b_rs1143634 (IL-1b 3953). Reagentes Concentração Tampão de PCR 1x MgCl2 2,0 mM dNTP 40 µM Iniciadores 0,2 µM Taq Polimerase 1,0 U Volume final 25 µL Ciclo 94ºC – 5’ 94ºC – 30’’ 53ºC – 30’’ 72ºC – 30’’ 40x 72ºC – 7’ 4ºC – ∞ Tabela 5. Condições gerais de PCR usadas para os marcadores FLI1_rs7930515 e FLI1_rs619521. Reagentes Concentração Tampão de PCR 1x MgCl2 4,0 mM dNTP 40 µM Iniciadores 0,2 µM Taq Polimerase 1,0 U Volume final 25 µL Ciclo 94ºC – 5’ 94ºC – 30’’ 62ºC – 30’’ 72ºC – 30’’ 40x 72ºC – 7’ 4ºC – ∞ Tabela 6. Condições gerais de PCR usadas para o marcador FLI1_rs145675082. Reagentes Concentração Ciclo Tampão de PCR MgCl2 1x 3,0 mM 94ºC – 5’ dNTP 40 µM Iniciadores 0,2 µM Taq Polimerase 1,0 U Volume final 25 µL 94ºC – 30’’ 62ºC – 30’’ 72ºC – 30’’ 72ºC – 7’ 4ºC – ∞ 40x 25 Tabela 7. Condições gerais de PCR usadas para o marcador FLI1_rs531894. Reagentes Concentração Tampão de PCR 1x MgCl2 3,2 mM dNTP 40 µM Iniciadores 0,2 µM Taq Polimerase 1,0 U Volume final 25 µL Ciclo 94ºC – 5’ 94ºC – 30’’ 62ºC – 30’’ 72ºC – 30’’ 40x 72ºC – 7’ 4ºC – ∞ A PCR foi realizada em termociclador Mastercycler® ep gradient S, Eppendorf, Germany. A identificação do produto amplificado foi realizada pela técnica de eletroforese em gel de agarose 2% corado com brometo de etídeo, usando-se 5 μL de produto de PCR, sendo visualizados e fotografados sob luz UV em um sistema de fotodocumentação. 3.6.2 Digestão Após a amplificação gênica através da técnica de PCR, foi realizada a digestão com as enzimas de restrição específicas para a identificação dos SNPs. Os produtos amplificados foram digeridos nas condições específicas de temperatura e tempo de incubação, dependendo das condições ideais de cada enzima de restrição, e tampão específico, de acordo com a tabela 8. Após a clivagem do DNA através da digestão enzimática, os fragmentos obtidos foram analisados por eletroforese em gel de agarose em diferentes concentrações, dependendo do tamanho do fragmento produzido pela enzima de restrição, com tampão TBE 1X, corado com brometo de etídeo e fotografados sob luz UV em um sistema de fotodocumentação. O marcador de tamanho molecular utilizado para a estimativa dos fragmentos gerados foi o ladder de 100 bp. 26 As enzimas e os respectivos tamanhos de fragmentos digeridos estão descritos na tabela 9. Tabela 8. Condições gerais usadas na reação de digestão. REAGENTES CONCENTRAÇÃO TEMPERATURA (ºC) E TEMPO DE INCUBACAO (h) ELETROFORESE CONCENTRAÇÃO DO GEL DE AGAROSE 1x 2U 10 µL 25 µL 37ºC 4h 2,0% 1x 1x 2U 10 µL 25 µL 65 ºC 4h 2,5% 1x 2U 10 µL 25 µL 37 ºC 4h 2,0% 1x 2U 10 µL 25 µL 37 ºC 4h 3,0% 1x 1x 2U 10 µL 25 µL 37 ºC 4h 3,0% 1x 2U 10 µL 25 µL 37 ºC 4h 3,0% 1x 2U 10 µL 25 µL 37 ºC 4h 3,0% IL1b_rs16944 Tampão de digestão Enzima Ava I H2O (qsp) Volume final IL1b_rs1143634 Tampão de digestão BSA Enzima Taqα I H2O (qsp) Volume final MCP1_rs1024611 Tampão de digestão Enzima Pvu II H2O (qsp) Volume final FLI1_rs7930515 Tampão de digestão Enzima Bfa I H2O (qsp) Volume final FLI1_rs145675082 Tampão de digestão BSA Enzima Nla III H2O (qsp) Volume final FLI1_rs619521 Tampão de digestão Enzima Pvu II H2O (qsp) Volume final FLI1_rs531894 Tampão de digestão Enzima Alu I H2O (qsp) Volume final 27 3.7. Análise estatística Análise estatística descritiva foi aplicada para os grupos caso e controle. Para as variáveis qualitativas, calcularam-se as frequências juntamente com as porcentagens em cada grupo; quanto as variáveis quantitativas, foi calculado média, desvio padrão, valores mínimo e máximo. Quanto às frequências alélicas e genotípicas dos SNPs, inicialmente foi realizado teste para verificar se estavam em equilíbrio de Hardy-Weinberg (HWE), bem como a estimativa do grau de Desequilíbrio de Ligação (DL) entre alelos de pares de marcadores usando o parâmetro r2. A análise foi realizada após implementação do software Haploview v.4.2. Dados genotípicos e fenotípicos obtidos para a população caso-controle foram utilizados para análise de associação com leishmaniose cutânea, para obtenção da significância das diferenças nas frequências observadas para o polimorfismo nos dois grupos (caso e controle) através do teste de chi-quadrado (X2) e “Odds ratio” (OR) utilizando-se o programa estatístico SPSS v.22. 3.8. Aspectos Éticos: O presente estudo faz parte de um projeto maior intitulado “Polimorfismos genéticos dos genes envolvidos na resposta imune e na cicatrização das lesões em pacientes com leishmaniose cutânea”, aprovado pelo CEP sob numeração CAAE:09995212.0.0000.0005. 3.9. Suporte Financeiro: O estudo recebeu suporte financeiro através do financiamento do projeto intitulado “Estudo da influência de polimorfismos genéticos na suscetibilidade à leishmaniose tegumentar causada por Leishmania guyanensis em população de áreas endêmicas de Manaus, estado do Amazonas”, pelo CNPq (MCTI/CNPq/MS-SCTIE) 28 sob numeração: 404181/2012-0 e coordenado pelo Prof. Dr. Rajendranath Ramasawmy. 29 Tabela 9. Condições gerais usadas na reação de PCR-RFLP. GENE MARCADOR POLIMORFISMO INICIADORES Ta PRODUTO DE PCR ENZIMA / SÍTIO DE RESTRIÇÃO ALELOS: PRODUTOS DE DIGESTÃO rs16944 -511C/T F: 5' TGG CAT TGA TCT GGT TCA TC 3' R: 5' GTT TAG GAA TCT TCC CAC TT 3' 53 °C 305 pb AvaI 5'...C↓YCGRG...3' 3'...GRGCY↑C...5' C: 190/115 T: 305 + 3953 C/T F: 5' GTT GTC ATC AGA CTT TGA CC 3' R: 5' TTC AGT TCA TAT GGA CCA GA 3' 53 °C 250pb TaqαI 5'...T↓CGA...3' 3'...AGC↑T...5' C: 136/114 T: 250 rs1024611 -2518 A/G F: 5' CCG AGA TGT TCC CAG CAC AG 3' R: 5' CTG CTT TGC TTG TGC CTC TT 3' 65/55 °C 930 pb Pvu II 5'...CAG↓CTG...3' 3'...GTC↑GAC...5' G: 708/222 A: 930 rs7930515 A/C F: 5' GGG TAA GTG GCC TCG TTG CAC GAG 3' R: 5' GGC GCT AGG GAT CTG TGC GGA CGG 3' 62ºC 243 pb BfaI 5'...C↓TAG...3' 3'...GAT↑C...5' A: 139/97/7 C: 236/7 rs619521 A/G F: 5' AGC TAG GCA CAG GTG CAC CTA GAC 3' R: 5' CCT GAA GGT GGG TGC CTT GGT TTC 3' 62ºC 324pb Pvu II 5'...CAG↓CTG...3' 3'...GTC↑GAC...5' G: 180/144 A: 324 rs145675082 C/T F: 5' GCC AAA ACT AGC GTG CTC CCT GTG 3' R: 5' GCT TGG CTG GAG GTG TCT GGT GAA AG 3' 62ºC 171pb NlaIII 5'...CATG↓...3' 3'...↑GTAC...5' C: 156/15 T: 171 rs531894 A/G F: 5' TAA GGA AGT GAA TAT CCC AGT CCT GTT 3' R: 5' ATG AGG AAG GTG TCT ACC TTC CTG 3' 62ºC 286pb Alu I 5'...AG↓CT...3' 3'...TC↑GA...5' G: 147/97/42 A: 244/42 IL-1b rs1143634 MCP-1 FLI-1 30 4. RESULTADOS 4.1. Características Gerais da População em Estudo Foram recrutados 392 indivíduos com a manifestação clínica da doença para compor o grupo caso. Desse total, 311 (79%) eram do masculino e 80 (20%) do sexo feminino. A idade teve uma variação de 5-70 anos de idade com média de 32,0 anos. Adicionalmente, analisou-se a média de idade de acordo com o sexo, tendo-se observado a mesma média (32,0 anos) tanto para o gênero masculino quanto feminino. Para compor o grupo controle, foram recrutados 382 indivíduos sem sinais e sintomas de LC, sendo 225 (59%) do sexo masculino e 157 (41%) pertencente ao sexo feminino. A idade dos indivíduos do grupo controle variou de 5-89 anos de idade, com uma média de 36,0 anos. Novamente, analisou-se a média de idade associada ao gênero, obtendo-se média de 38,0 anos para o gênero masculino e 34,0 anos para o gênero feminino. A tabela 10 sumariza a análise descritiva da população caso-controle quanto ao sexo e idade. Tabela 10. Características gerais da população caso-controle em estudo. Sexo (%) Masculino Feminino Dado faltante Média de idade – Anos (± DP) Quanto ao sexo Masculino Feminino DP = Desvio Padrão CASO CONTROLE n= 392 311 (79) 80 (20) 1 (3) n= 382 225 (59) 157(41) ... 32 ± 14.1 36 ± 17.7 32 ± 13.7 32 ± 15.5 38 ± 17.6 34 ± 17.5 31 4.2. Genotipagem, HWE e DL Os marcadores analisados apresentaram-se em HWE, com exceção do marcador FLI1_rs145675082, que por não estar em equilíbrio foi excluído da análise. Quanto à genotipagem, esta foi realizada por PCR-RFLP conforme ilustrado nas figuras 4, 5 e 6; e apresentou êxito ≥90% para todos os marcadores testados. A B Figura 4. Padrão eletroforético da PCR-RFLP para os SNPs IL1-B +3953 C/T e IL1-B -511 C/T respectivamente. A: Digestão do produto de PCR com TaqαI produz fragmentos de 136 e 114 pb (Alelo T) ou 250 pb (Alelo C). Colunas 1 e 2 heterozigoto CT; Colunas 3 e 5 homozigoto CC; e Coluna 4 homozigoto CC. B: Digestão do produto de PCR com Ava I produz fragmentos de 190 e 115 pb (Alelo C) ou 305 pb (Alelo T). Colunas 2, 3 e 5 homozigoto TT; Coluna 7 homozigoto CC, Colunas 1, 4 e 6 heterozigoto CT. M: marcador de 100 pb; Figura 5. Padrão eletroforético da PCR-RFLP para o SNP MCP1 -2518 A/G. Digestão do produto de PCR com Pvu II produz fragmentos de 708 e 222 pb (tipo selvagem) ou 930 pb (tipo mutante). Colunas 3 e 6 homozigoto mutante; Coluna 2 homozigoto do tipo selvagem, Colunas 1, 4 e 5 heterozigoto. M: marcador de 100 pb; 32 Figura 6. Padrão eletroforético da PCR-RFLP para os SNPs FLI1_rs7930515, FLI1_rs619521 e FLI1_rs531894 respectivamente. rs7930515: Digestão do produto de PCR com Bfa I produz fragmentos de 139, 97 e 7 pb (Alelo A) ou 236 e 7 pb (Alelo C). Coluna 1A, produto de PCR de 243 pb; Coluna 1B homozigoto CC; Coluna 1C heterozigoto AC; Coluna 1D homozigoto AA. rs619521: Digestão do produto de PCR com Pvu II produz fragmentos de 180 e 144 pb (Alelo G) ou 324 pb (Alelo A). Coluna 2A, produto de PCR de 324 pb; Coluna 2B homozigoto AA; Coluna 2C heterozigoto AG; Coluna 2D homozigoto GG. rs531894: Digestão do produto de PCR com Alu I produz fragmentos de 147, 97 e 42 pb (Alelo G) ou 244 e 42 pb (Alelo A). Coluna 3A, produto de PCR de 286 pb; Coluna 3B homozigoto AA; Coluna 3C heterozigoto AG; Coluna 3D homozigoto GG. M: marcador de 100 pb. Análise para verificar o DL entre os SNPs dos genes IL-1b e FLI-1 foi realizada utilizando-se para cálculo o parâmetro r2, conforme ilustra a figura 7. A análise do DL confirmou independência para todos os SNPs testados. Para o gene MCP-1 não foi possível gerar o ”plot” de DL, pois para este gene foi realizada a genotipagem de apenas um marcador. A B r2 r2 Figura 7. Análise de desequilíbrio de ligação entre os SNPs dos genes IL-1b e FLI-1 respectivamente. O número no interior do quadrado indica a proporção de DL em porcentagem, calculado utilizando-se o parâmetro r2. O código de cores reflete a intensidade de DL entre dois loci. 33 4.2.1 Análise dos marcadores MCP1_rs1024611, IL1b_rs1143634, FLI1_rs619521 e FLI1_rs531894. As frequências alélicas e genotípicas para os marcadores MCP1_rs1024611, IL1b_rs1143634, FLI1_rs619521 e FLI1_rs531894 estão descritas na tabela 11. Conforme análise, não foi observada evidência de associação desses marcadores com leishmaniose cutânea na amostra populacional estudada, pois não foram observadas diferenças estatisticamente significativas nas frequências alélicas e genotípicas quando foram comparados os grupos caso e controle para os três modelos de comparação que foram aplicadas (genotípico, alélico e de dominância), todos com valores de P superiores a 0.05. 4.2.2. Análise do marcador IL1b_rs16944 Para o SNP IL1b_rs16944 do gene IL-1b, análise da distribuição das frequências alélica, genotípica e de dominância foram aplicadas entre os grupos caso e controle, observando-se evidência positiva de associação desse SNP (modelo aditivo P=0.026 e dominante P=0.027) com leishmaniose cutânea na amostra estudada, conforme descrito na tabela 12. 4.2.3. Análise do marcador FLI1_rs7930515 Para o marcador FLI1_rs7930515 do gene FLI-1, também foram aplicadas análises da distribuição das frequências alélica, genotípica e de dominância entre os grupos caso e controle, podendo-se observar valores de P “boderline” (modelo aditivo P=0.075 e dominante P=0.036), com OR de 2.38 [IC 95% 1.07 – 5.32]. 34 Tabela 11. Comparação das distribuições das frequências genotípicas e alélicas para os marcadores MCP1_rs1024611, IL1b_rs1143634, FLI1_rs619521 e FLI1_rs531894. GRUPO CASO GRUPO CONTROLE n= 346 (%) n= 324 (%) AA 83 (24) 70 (22) AG 179 (52) 157 (48) GG 84 (24) 97 (30) IL1b_rs1143634 345 (50) 347 (50) n= 388 (%) 297 (46) 351 (54) n= 371 (%) Genótipo CC 296 (76) 282 (76) CT 85 (22) 78 (21) TT 7 (2) 11 (3) 677 (87) 99 (13) n= 354 (%) 642 (87) 100 (13) n= 316 (%) 131 (37) 169 (48) 54 (15) 114 (36) 160 (51) 42 (13) 431 (61) 277 (39) n= 337 (%) 388 (61) 244 (39) n= 318 (%) 87 (26) 156 (46) 94 (28) 330 (49) 344 (51) MCP1_rs1024611 P X2 0.25 2.76 0.14 2.18 0.56 1.15 0.17 0.68 0.77 0.68 0.19 0.66 76 (24) 154 (48) 88 (28) 0.40 0.82 306 (48) 330 (52) 0.09 0.76 Genótipo Alelo A G Alelo C T FLI1_rs531894 Genótipo AA AG GG Alelo A G FLI1_rs619521 Genótipo AA AG GG Alelo A G 35 Tabela 12. Comparação da distribuição das frequências genotípica, alélica e de dominância para o marcador IL1b_rs16944. GRUPO CASO GRUPO CONTROLE IL1b_rs16944 Genótipo n= 374 (%) n= 352 (%) CC 81 (22) 54 (15) CT 183 (49) 167 (48) TT 110 (29) 131 (37) CC + CT 264 (71) 221 (63) TT 110 (29) 131 (37) CT + TT 293 (78) 298 (85) CC 81 (22) 54 (15) Alelo C T 345 (46) 403 (54) 275 (39) 429 (61) P OR [95% CI] 0.026 ... 0.027 1.42 [1.04 – 1.94] 0.036 0.66 [0.45 – 0.96] 0.007 1.34 [1.084-1.646] Dominância 36 Tabela 13. Comparação da distribuição das frequências genotípica, alélica e de dominância para o marcador FLI1_rs7930515. GRUPO CASO GRUPO CONTROLE FLI1_rs7930515 Genótipo n= 347 (%) n= 335 (%) AA 9 (3) 20 (6) AC CC 144 (41) 194 (56) 142 (42) 173 (52) AA + AC CC 153 (44) 194 (56) AC + CC AA Alelo A C P OR [95% CI] 0.075 ... 162 (48) 173 (52) 0.28 0.84 [0.62 – 1.14] 338 (97) 9 (3) 315 (94) 20 (6) 0.036 2.38 [1.07 – 5.32] 345 (46) 403 (54) 275 (39) 429 (61) 0.10 1.23 [0.96 – 1.56 ] Dominância 37 5. DISCUSSÃO As leishmanioses ainda são consideradas um grande problema de saúde pública, representando um complexo de doenças com importante espectro clínico e diversidade epidemiológica. Nas áreas onde a leishmaniose é considerada endêmica, somente uma fração da população exposta ao parasita desenvolve a doença (8,1,75). O fato de alguns indivíduos não apresentarem nenhum sinal ou sintoma de infecção, enquanto outros desenvolvem formas crônicas ainda não foi claramente elucidado (44). Acredita-se que a patogenia da LTA é fortemente influenciada pelo perfil imunogenético do homem, que se encontra igualmente associado à resposta imune mediada por células T e pela virulência da espécie de Leishmania infectante, além da ação integrada de fatores ambientais, socioeconômicos e culturais (8). Para avaliar o impacto de fatores genéticos em leishmaniose cutânea, neste estudo optou-se por analisar SNPs em IL-1b, MCP-1 e FLI-1 associados à LC, provenientes da literatura, para replicação. A população estudada neste projeto foi composta por pacientes com LC, recrutados no ambulatório de Dermatologia da FMT-HVD por demanda espontânea, provenientes de áreas endêmicas para leishmaniose no Amazonas, com destaque para o ramal da Cooperativa e ramal do Pau-Rosa, com suas respectivas vicinais; e indivíduos sem a doença, provenientes das mesmas áreas endêmicas dos casos. De acordo com os achados no presente estudo, dos indivíduos acometidos com LC, 79% eram do sexo masculino e 20% do sexo feminino. A média de idade para os casos foi de 32 anos de idade. Nossos dados estão de acordo com os achados de Benicio et al., 2011, onde foram observados 78% de indivíduos do sexo masculino e 22% do sexo feminino, com média de idade de 29 anos (76); e com os de Romero et al., 2002, em que 82% dos indivíduos com leishmaniose eram do sexo masculino, com média de idade 27 anos (77). Segundo o Manual de Vigilância da Leishmaniose Tegumentar Americana, a doença atinge indivíduos do sexo masculino principalmente, em fase produtiva, o que caracteriza o acometimento de indivíduos cuja principal atividade ocupacional está associada à penetração em áreas de mata, desflorestamento, agropecuária ou 38 exercícios militares, tornando essa população altamente exposta à infecção por Leishmania sp (1). Para o grupo controle, 59% dos indivíduos correspondiam ao sexo masculino e 41% ao sexo feminino, com idade média de 36 anos. Ter um grupo controle com idade média superior ao caso é encarado como fator positivo, pois dessa maneira tem-se uma maior segurança em trabalhar com um grupo que ultrapassou a idade média do grupo caso, sob as mesmas condições de risco, no entanto não desenvolveu a doença. 5.1. MCP-1 Polimorfismos genéticos em MCP-1 estão associados com diversas doenças infecciosas, tais como hepatite B e C, tuberculose, leishmaniose mucosa, doença de chagas, entre outras (45,73,78–81). Estudo realizado por Flores-Villanueva et al., 2005 em populações do México e Korea, demonstrou associação do alelo G do polimorfismo MCP1_ rs1024611 (MCP1 -2518 A/G) com suscetibilidade para desenvolver a forma ativa da tuberculose. Indivíduos que possuíam os genótipos AG e GG apresentaram maior risco de desenvolver tuberculose do que os portadores do genótipo AA. Assim, os achados sugeriram que indivíduos que carregam o genótipo GG produzem maiores concentrações de MCP-1, que inibe a produção de IL-12p40 em resposta ao M. tuberculosis e aumenta a probabilidade de progressão da infecção pelo bacilo para uma tuberculose pulmonar ativa (79). Ramasawmi et al., 2010 realizaram estudo para verificar associação do polimorfismo em MCP1 -2518 A/G com LC e LM. Os dados apresentados pelos autores fornecem evidências de base populacional e familiar para uma associação entre o alelo G desse SNP e susceptibilidade para LM causada por L. braziliensis, e não para LC. Esses dados foram correlacionados com elevados níveis de MCP-1 no plasma de indivíduos homozigotos para o alelo G, e com alta liberação de MCP-1 por macrófagos estimulados e não estimulados (45). 39 Esses dados corroboram com os achados no presente estudo, em que não foi possível encontrar associação entre o polimorfismo MCP1 -2518 A/G e LC por não haver diferença estatisticamente significativa entre os grupos de caso e controle (modelo aditivo P=0.25, X2=2.76). Segundo análise realizada por Ramasawmy et al., 2010, a comparação do grupo de indivíduos com LC e de indivíduos sem o fenótipo não foi estatisticamente significativa tanto para os modelos aditivo e dominante, sugerindo que esse polimorfismo não é agente influenciador para o fenótipo LC (45). Apesar de a grande maioria dos casos notificados no estado do Amazonas serem causados por L. guyanensis, espécie que difere da observada por Ramasawmy et al, 2010 (L. braziliensis), a semelhança no resultado encontrado pode ser explicada pelo fato de ambas pertencerem ao subgênero Viannia, e também pelo perfil genético bastante similar das duas populações estudadas. No entanto, é importante ressaltar que outros estudos nesta população devem ser conduzidos para outros fenótipos da leishmaniose, em especial LM a fim de melhor elucidar o possível envolvimento desse gene com leishmaniose per se. 5.2 FLI-1 Polimorfismos em FLI-1 foram descritos pela primeira vez em associação com L. major baseado em modelo murino (82). Sakthianandeswaren et al., 2010, validaram a importância do FLI-1 na resolução e cicatrização das lesões e identificaram três polimorfismos na região promotora deste gene, indicando-o como novo candidato influenciando na resistência à L. major. Nesse estudo, observou-se que em camundongos resistentes, o pico na expressão de FLI-1 em linfonodos na sexta semana de infecção correspondia ao observado no pico fenotípico no mesmo período, em que camundongos resistentes apresentaram estabilização e cicatrização da lesão. Em contraste, as lesões presentes em camundongos susceptíveis, seis semanais após infecção continuavam a crescer em tamanho e gravidade, possivelmente devido à ausência de uma expressão aumentada de FLI-1. 40 Posteriormente, Castellucci et al., 2011, baseado no estudo citado anteriormente, examinaram FLI-1 como um gene controlando LC causada por L. braziliensis num estudo de associação baseado em família (70). No estudo de Castelluci et al., 2011, os SNPs de FLI-1 foram genotipados inicialmente em 168 famílias nucleares compostas por 250 casos de LC e 87 casos de LM, todos provenientes de Corte de Pedra, área de endemicidade para L. braziliensis, Bahia. Foi demonstrado associação do alelo C (P=0.003; OR=1.64) no SNP FLI1_rs7930515 e risco para LC, que após análise primária, foi replicada numa segunda amostra de 402 casos de LC em 157 famílias nucleares (P=0.014;OR=1.60) recrutadas na mesma região. No presente estudo, dentre os quatro SNPs genotipados para FLI-1, apenas verificou-se tendência para associação com LC no SNP FLI1_rs7930515 (modelo aditivo P=0.075 e dominante P=0.036). Esta tendência à associação pode ser observada entre o alelo C e susceptibilidade para leishmaniose cutânea pelo modelo de dominância [P=0.036 OR=2.38 (IC 95% 1.07 – 5.32)]. Os dados observados no presente estudo corroboram com os achados de Castellucci et al., 2011, em que o alelo C estava associado com risco para LC, no entanto nossos achados podem apenas sugerir uma associação, uma vez que o valor de P apresentou-se “boderline”. Esses dados, somados a outros estudos evidenciam a importância do gene FLI1 como candidato influenciando resistência/susceptibilidade na leishmaniose, além de ter sido correlacionado com uma resposta de cura de lesão em modelo murino (70,82,83). Estudos posteriores devem ser conduzidos para o mesmo marcador aumentando-se o tamanho de amostra, a fim de desvendar se a associação encontrada no presente estudo se tornaria mais forte, a ponto de ser considerada significativa (P<0.05); ou se este indício de associação desapareceria, podendo-se descartá-lo como marcador associado a LC na polulação estudada. 5.3 IL-1b 41 Interleucina 1 (IL-1) pertence ao grupo de citocinas pro-inflamatórias do tipo Th1, e tem papel chave como mediador da resposta inflamatória com efeitos pleiotrópicos numa variedade de células e vias de sinalização, e tem função importante no desfecho da condição patológica (56). IL-1b é uma citocina com atividade pirogênica que é normalmente ativada em resposta a uma infecção ou injúria (64). Os inflamassomas tem papel um importante nesse contexto, devido sua função de promover a ativação de IL-1b através de vários estímulos durante a infecção por micro-organismos (65). Polimorfismos genéticos em IL-1b estão associados com susceptibilidade a diversas doenças autoimunes, inflamatórias e infecciosas, dentre elas tuberculose, gastrite crônica associada a Helicobacter pylori, HIV, esclerose múltipla, lupus eritematoso sistêmico, malária e leishmaniose (58–63). Existem dois polimorfismos bastante comuns no gene IL-1b influenciando a expressão da citocina: um localizado no exon 5 (IL-1b 3953 C/T) e outro localizado na região promotora (IL1-b -511 C/T) (84). Fernández-Figueroa et al., 2012 demonstraram que o polimorfismo em IL-1b (511 C/T) está associado com um alto risco de contrair a doença em pacientes infectados com L. mexicana. Também foram capazes de demonstrar que pacientes com a forma mais grave da doença e que albergam um maior número de parasitas de L. mexicana, apresentam uma produção aumentada de IL1-b por monócitos in vitro, uma acentuada expressão sérica de IL-1b e uma distribuição difusa de IL-1b nas lesões (63). Estudo realizado para verificar associação do polimorfismo IL-1b -511 C/T com LV, demonstrou que o alelo C pareceu conferir susceptibilidade a LV, enquanto a presença do alelo T atuou como possível fator influenciador para resistência à doença (84). Esses achados corroboram com os do presente estudo, em que se observou associação dos alelos C e T do polimorfismo IL-1b -511 C/T com susceptibilidade e resistência, respectivamente, à LC. Pelo modelo de dominância, pode-se sugerir a presença do alelo C conferindo risco [P = 0.027, OR=1.43 (IC 95% 1.04 – 1.94)] e o 42 contrário, do alelo T conferindo resistência [P = 0.036, OR = 0.66 (IC 95% 0.45 – 0.96)]. Moravej et al., 2012, que também obtiveram o mesmo perfil de associação desse polimorfismo com LV, sugerem que indivíduos portadores do alelo IL-1b -511 C produzem mais IL-1b do que os portadores do alelo T, resultando no desenvolvimento da doença e resolução da lesão (84). Ainda, segundo dados de Fernández-Figueroa et al., 2012, a distribuição de IL1b nos tecidos está associada à carga parasitária no hospedeiro, assim indivíduos com a forma mais grave da doença, e que, portanto possuíam carga parasitária maciça, apresentavam maiores níveis celulares de IL-1b. O oposto também foi observado (63). Associação de IL1b 3953 C/T foi observada conferindo risco para desenvolvimento da forma grave de malária em uma população de Gambia (61). Da mesma forma, associação desse polimorfismo foi observada com susceptibilidade para infecção por HIV (85). Mas não para leishmaniose visceral em uma população iraniana (84). Dados de Moravej et al., 2012 corroboram com os achados deste estudo, em que também não foi possível encontrar associação do polimorfismo em IL-1b 3953 C/T com LC (Modelo aditivo, P=1.15, X2=0.56). Pesquisas conduzidas ao longo das últimas décadas vem resultando em sólido corpo de evidências em favor da existência de fatores genéticos atuando no controle da susceptibilidade do hospedeiro à leishmaniose através de polimorfismos em genes indutores de citocinas ou quimiocinas com influência direta na resposta immune, resultando num amplo espectro de manifestações da doença. Dessa forma, a identificação dos genes e do mecanismo biológico por trás do efeito genético observado pode significar um enorme avanço no entendimento da patogênese de doenças complexas em geral. Assim, os dados gerados no presente estudo, somados aos já existentes na literatura, vem contribuir de modo crítico para um maior entendimento da patogênese da LC, podendo-se, futuramente, levar ao desenvolvimento de melhores estratégias de diagnóstico, tratamento e prevenção. 43 6. CONCLUSÃO Através dos resultados gerados neste estudo pode-se concluir: Não foram observadas associações genéticas com a susceptibilidade/resistência à LC para os SNPs IL1b_rs1143634 (IL-1b 3953 C/T), MCP1_rs1024611 (MCP-1 -2518 A/G), FLI1_rs619521 e FLI1_rs531894 na população estudada. Foram detectadas diferenças estatisticamente significativas na distribuição alélica e genotípica do polimorfismo em IL1b_rs16944 (IL-1b -511 C/T) entre os grupos caso e controle, sugerindo associação do alelo C com susceptibilidade, e do alelo T com resistência para LC. Para o SNP FLI1_rs7930515 foi observado uma tendência para associação, podendose sugerir que o alelo C estava associado com risco para LC. Os resultados obtidos no presente estudo reforçam o papel dos genes IL1-b e FLI-1 associados com resistência a LC. 44 7. REFERÊNCIAS BIBLIOGRÁFICAS 1. Ministério da Saúde. Secretaria de Vigilância em Saúde. Departamento de Vigilância Epidemiológica. Manual de Vigilância da Leishmaniose Tegumentar Americana [Internet]. 2a ed. Brasília: Ministério da Saúde; 2010. Available from: http://bvsms.saude.gov.br/bvs/publicacoes/manual_vigilancia_leishmaniose_te gumentar_americana.pdf 2. Murray HW, Berman JD, Davies CR, Saravia NG. Advances in leishmaniasis. Lancet [Internet]. 2005;366(9496):1561–77. Available from: http://www.ncbi.nlm.nih.gov/pubmed/16257344 3. Sharma U, Singh S. Immunobiology of leishmaniasis. Indian J Exp Biol [Internet]. 2009 Jun;47(6):412–23. Available from: http://www.ncbi.nlm.nih.gov/pubmed/19634705 4. Neto VA, Gryschek RCB, Amato VS, Tuon FF. Parasitologia: Uma abordagem clínica. Rio de Janeiro: Elsevier; 2008. 5. De Brito MEF, Andrade MS, Dantas-Torres F, Rodrigues EHG, Cavalcanti MDP, de Almeida AMP, et al. Cutaneous leishmaniasis in northeastern Brazil: a critical appraisal of studies conducted in State of Pernambuco. Rev Soc Bras Med Trop [Internet]. 2012 Aug [cited 2013 May 6];45(4):425–9. Available from: http://www.ncbi.nlm.nih.gov/pubmed/22836662 6. Ministério da Saúde. Secretaria de Vigilância em Saúde. Departamento de Vigilância Epidemiológica. Atlas de Leishmaniose Tegumentar Americana: Dignóstico clínico e diferencial [Internet]. 1a ed. Brasília: Ministério da Saúde; 2006. Available from: http://portal.saude.gov.br/portal/arquivos/pdf/atlas_lta.pdf 7. Neves DP. Parasitologia Humana. 11a ed. São Paulo: Atheneu; 2004. 8. Desjeux P. Leishmaniasis: current situation and new perspectives. Comp Immunol Microbiol Infect Dis [Internet]. 2004 Sep [cited 2013 Feb 27];27(5):305– 18. Available from: http://www.ncbi.nlm.nih.gov/pubmed/15225981 9. Toledo ACDC, da Silva RE, Carmo RF, Amaral TA, Luz ZMP, Rabello A. Assessment of the quality of life of patients with cutaneous leishmaniasis in Belo Horizonte, Brazil, 2009-2010. A pilot study. Trans R Soc Trop Med Hyg [Internet]. 2013 May [cited 2014 Oct 28];107(5):335–6. Available from: http://www.ncbi.nlm.nih.gov/pubmed/23474473 10. Alvar J, Vélez ID, Bern C, Herrero M, Desjeux P, Cano J, et al. Leishmaniasis worldwide and global estimates of its incidence. PLoS One [Internet]. 2012 Jan [cited 2014 Jul 16];7(5):e35671. Available from: http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=3365071&tool=pmce ntrez&rendertype=abstract 45 11. Teles CBG, Basano SA, Zagonel-Oliveira M, Campos JJ, Oliveira AFJ De, Freitas RA De, et al. Epidemiological aspects of American cutaneous leishmaniasis and phlebotomine sandfly population, in the municipality of Monte Negro, State of Rondônia, Brazil. Rev Soc Bras Med Trop [Internet]. 2013 Jan [cited 2013 Apr 9];46(1):60–6. Available from: http://www.ncbi.nlm.nih.gov/pubmed/23563827 12. Naiff-Júnior RD, Pinheiro FG, Farias M De, Castro LM. Estudo de uma série de casos de leishmaniose tegumentar americana no município de Rio Preto da Eva, Amazonas, Brasil. Rev Patol Trop [Internet]. 2009;38(2):103–14. Available from: http://www.revistas.ufg.br/index.php/iptsp/article/view/6610/4868 13. Ministério da Saúde. Secretaria de Vigilância em Saúde. Departamento de Vigilância Epidemiológica. Leishmaniose Tegumentar Americana. Casos de Leishmaniose Tegumentar Americana. Brasil, Grandes Regiões e Unidades Federadas. 1990 a 2011 [Internet]. 2011 [cited 2013 Apr 3]. Available from: http://portal.saude.gov.br/portal/arquivos/pdf/2012_11_casos_de_lta_entre_19 90_e_2011.pdf 14. Fundação de Medicina Tropical Dr Heitor Vieira Dourado. Diretoria de Ensino e Pesquisa. Departamento de Epidemiologia e Saúde Pública. SINAM. Leishmaniose (LTA) notificada na FMT-HVD – 2011-2013. 15. Singh S. New developments in diagnosis of leishmaniasis. Indian J Med Res [Internet]. 2006 Mar [cited 2013 May 8];123(3):311–30. Available from: http://www.ncbi.nlm.nih.gov/pubmed/16778313 16. Ministério da Saúde. Secretaria de Vigilância em Saúde. Departamento de Vigilância Epidemiológica. Manual de Vigilância da Leishmaniose Tegumentar Americana [Internet]. 2a ed. Brasília; 2007. Available from: http://portal.saude.gov.br/portal/arquivos/pdf/manual_lta_2ed.pdf 17. Hackel C. Identificação específica do M. leprae pela técnica da Reação em Cadeia da Polimerase(PCR). Hansen Int. 1990;15(1/2):67–75. 18. CUNHA FMB. Reação em Cadeia da Polimerase (PCR) - Valor diagnóstico na hanseníase neural pura. [Tese]. Curitiba: Universidade Federal do Paraná; 2003. p. 129. 19. Gatti S, Gramegna M, Klersy C, Madama S, Bruno a, Maserati R, et al. Diagnosis of visceral leishmaniasis: the sensitivities and specificities of traditional methods and a nested PCR assay. Ann Trop Med Parasitol [Internet]. 2004 Oct [cited 2013 May 8];98(7):667–76. Available from: http://www.ncbi.nlm.nih.gov/pubmed/15509421 20. Lemrani M, Hamdi S, Laamrani A, Hassar M. PCR detection of Leishmania in skin biopsies. J Infect Dev Ctries [Internet]. 2009 Jan [cited 2013 Apr 12];3(2):115–22. Available from: http://www.ncbi.nlm.nih.gov/pubmed/19755741 46 21. De Paiva Cavalcanti M, Felinto de Brito ME, de Souza WV, de Miranda Gomes Y, Abath FGC. The development of a real-time PCR assay for the quantification of Leishmania infantum DNA in canine blood. Vet J [Internet]. Elsevier Ltd; 2009 Nov [cited 2013 Mar 19];182(2):356–8. Available from: http://www.ncbi.nlm.nih.gov/pubmed/18603455 22. Mohammadiha A, Mohebali M, Haghighi A, Mahdian R, Abadi a R, Zarei Z, et al. Comparison of real-time PCR and conventional PCR with two DNA targets for detection of Leishmania (Leishmania) infantum infection in human and dog blood samples. Exp Parasitol [Internet]. Elsevier Inc.; 2013 Jan [cited 2013 May 8];133(1):89–94. Available from: http://www.ncbi.nlm.nih.gov/pubmed/23159412 23. Gil EDS, Cunha LC, Paula JR De, Bezerra JCB, Aguiar FA. Leishmaníase : Arsenal Terapêutico e Alvos Moleculares. Vita Sanitas [Internet]. 2007;1(1). Available from: http://www.fug.edu.br/revista/artigos/Organizados/Leishmaniase.pdf 24. Amato VS. Informe Mensal sobre Agravos à Saúde Pública: Tratamento da Leishmaniose Tegumentar Americana. Boletim Epidemiológico Paulista [Internet]. São Paulo; 2006 Feb; Available from: http://www.cve.saude.sp.gov.br/agencia/bepa26_lta.htm 25. Neves LO, Talhari AC, Gadelha EPN, Silva Júnior RM da, Guerra JA de O, Ferreira LC de L, et al. Estudo clínico randomizado comparando antimoniato de meglumina, pentamidina e anfotericina B para o tratamento da leishmaniose cutânea ocasionada por Leishmania guyanensis. An Bras Dermatol [Internet]. 2011 Dec [cited 2013 Apr 19];86(6):1092–101. Available from: http://www.scielo.br/scielo.php?script=sci_arttext&pid=S036505962011000600005&lng=pt&nrm=iso&tlng=en 26. Silveira FT, Lainson R, Corbett CEP. Clinical and Immunopathological Spectrum of American Cutaneous Leishmaniasis with Special Reference to the Disease in Amazonian Brazil - A Review. Mem Inst Oswaldo Cruz. 2004;99(May):239–51. 27. Carvalho MDLR, de Andrade ASR, Fontes CJF, Hueb M, de Oliveira Silva S, Melo MN. Leishmania (Viannia) braziliensis is the prevalent species infecting patients with tegumentary leishmaniasis from Mato Grosso State, Brazil. Acta Trop [Internet]. 2006 Jul [cited 2013 Mar 30];98(3):277–85. Available from: http://www.ncbi.nlm.nih.gov/pubmed/16846569 28. Romero G a, Vinitius De Farias Guerra M, Gomes Paes M, de Oliveira Macêdo V. Comparison of cutaneous leishmaniasis due to Leishmania (Viannia) braziliensis and L. (V.) guyanensis in Brazil: clinical findings and diagnostic approach. Clin Infect Dis [Internet]. 2001 May 1;32(9):1304–12. Available from: http://www.ncbi.nlm.nih.gov/pubmed/11303265 29. Oliveira CI De, Teixeira MJ, Gomes R, Barral A, Brodskyn C. Animal models for infectious diseases caused by parasites: Leishmaniasis. Drug Discov Today Dis Model [Internet]. 2004 Oct [cited 2013 Mar 10];1(1):81–6. Available from: http://linkinghub.elsevier.com/retrieve/pii/S1740675704000155 47 30. Silveira FT, Müller SR, De Souza AAA, Lainson R, Gomes CMC, Laurenti MD, et al. Revisão sobre a Patogenia da Leishmaniose Tegumentar Americana na Amazônia, com ênfase à doença causada por Leishmania (V.) braziliensis e Leishmania (L.) amazonensis. Rev Para Med [Internet]. 2008;22(1):9–20. Available from: http://scielo.iec.pa.gov.br/scielo.php?script=sci_arttext&pid=S010159072008000100002&lng=pt&nrm=iso 31. De Almeida LF. Estudo do polimorfismo em genes envolvidos no processo de cura de lesões e o desenvolvimento da leishmaniose tegumentar americana. [Dissertação]. Salvador: Universidade Federal da Bahia; 2012. p. 119. 32. Bhattacharya P, Ali N. Involvement and interactions of different immune cells and their cytokines in human visceral leishmaniasis. Rev Soc Bras Med Trop [Internet]. 2013;46(2):128–34. Available from: http://www.ncbi.nlm.nih.gov/pubmed/23559342 33. Carvalho LP, Passos ST, Jesus AR. Imunopatogênese da Leishmaniose Tegumentar. Gaz Médica da Bahia [Internet]. 2005;1:57–65. Available from: http://www.gmbahia.ufba.br/index.php/gmbahia/article/viewFile/351/340 34. Khader S a, Gopal R. IL-17 in protective immunity to intracellular pathogens. Virulence [Internet]. 2010;1(5):423–7. Available from: http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=2953849&tool=pmce ntrez&rendertype=abstract 35. Reiner SL, Locksley RM. The regulation of immunity to Leishmania major. Annu Rev Immunol [Internet]. 1995 Jan [cited 2013 Apr 9];13:151–77. Available from: http://www.ncbi.nlm.nih.gov/pubmed/7612219 36. Gonzalez-Lombana C, Gimblet C, Bacellar O, Oliveira WW, Passos S, Carvalho LP, et al. IL-17 mediates immunopathology in the absence of IL-10 following Leishmania major infection. PLoS Pathog [Internet]. 2013 Mar [cited 2014 Jun 12];9(3):e1003243. Available from: http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=3605236&tool=pmce ntrez&rendertype=abstract 37. Pereira BAS, Alves CR. Immunological characteristics of experimental murine infection with Leishmania (Leishmania) amazonensis. Vet Parasitol [Internet]. 2008 Dec 20 [cited 2013 Apr 5];158(4):239–55. Available from: http://www.ncbi.nlm.nih.gov/pubmed/18922635 38. Austin CP, Battey JF, Bradley A, Bucan M, Capecchi M, Collins FS, et al. The knockout mouse project. Nat Genet [Internet]. 2004 Sep [cited 2013 Apr 19];36(9):921–4. Available from: http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=2716027&tool=pmce ntrez&rendertype=abstract 39. Hezarjaribi HZ, Ghaffarifar F, Dalimi A, Sharifi Z, Jorjani O. Effect of IL-22 on DNA vaccine encoding LACK gene of Leishmania major in BALB/c mice. Exp 48 Parasitol [Internet]. Elsevier Inc.; 2013 Mar 26 [cited 2013 Apr 5]; Available from: http://www.ncbi.nlm.nih.gov/pubmed/23541883 40. DeKrey GK, Lima HC, Titus RG. Analysis of the immune responses of mice to infection with Leishmania braziliensis. Infect Immun [Internet]. 1998 Feb [cited 2013 Apr 17];66(2):827–9. Available from: http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=107977&tool=pmcen trez&rendertype=abstract 41. Courret N, Lang T, Milon G, Antoine JC. Intradermal inoculations of low doses of Leishmania major and Leishmania amazonensis metacyclic promastigotes induce different immunoparasitic processes and status of protection in BALB/c mice. Int J Parasitol [Internet]. 2003 Oct [cited 2013 May 24];33(12):1373–83. Available from: http://linkinghub.elsevier.com/retrieve/pii/S0020751903001796 42. Alter A, Grant A, Abel L, Alcaïs A, Schurr E. Leprosy as a genetic disease. Mamm Genome [Internet]. 2011 Feb;22(1-2):19–31. Available from: http://www.ncbi.nlm.nih.gov/pubmed/21585261 43. Birnbaum R, Craft N. Innate Immunity and Leishmania Vaccination Strategies. Dermatol Clin [Internet]. Elsevier Ltd; 2011;29(1):89–102. Available from: http://dx.doi.org/10.1016/j.det.2010.08.014 44. Ajdary S, Ghamilouie M-M, Alimohammadian M-H, Riazi-Rad F, Pakzad S-R. Toll-like receptor 4 polymorphisms predispose to cutaneous leishmaniasis. Microbes Infect [Internet]. Elsevier Masson SAS; 2011 Mar [cited 2012 Sep 8];13(3):226–31. Available from: http://www.ncbi.nlm.nih.gov/pubmed/21056683 45. Ramasawmy R, Menezes E, Magalhães A, Oliveira J, Castellucci L, Almeida R, et al. The -2518bp promoter polymorphism at CCL2/MCP1 influences susceptibility to mucosal but not localized cutaneous leishmaniasis in Brazil. Infect Genet Evol [Internet]. 2010 Jul 5 [cited 2012 Sep 8];10(5):607–13. Available from: http://www.ncbi.nlm.nih.gov/pubmed/2878927 46. Blackwell JM, Mohamed HS, Ibrahim ME. Genetics and visceral leishmaniasis in the Sudan: seeking a link. Trends Parasitol [Internet]. 2004 Jun [cited 2012 Aug 10];20(6):268–74. Available from: http://www.ncbi.nlm.nih.gov/pubmed/15147677 47. Bucheton B, Abel L, Kheir MM, Mirgani a, El-Safi SH, Chevillard C, et al. Genetic control of visceral leishmaniasis in a Sudanese population: candidate gene testing indicates a linkage to the NRAMP1 region. Genes Immun [Internet]. 2003 Mar [cited 2012 Aug 6];4(2):104–9. Available from: http://www.ncbi.nlm.nih.gov/pubmed/12618857 48. El-Safi S, Kheir MM, Bucheton B, Argiro L, Abel L, Dereure J, et al. Genes and environment in susceptibility to visceral leishmaniasis. C R Biol [Internet]. 2006 Nov [cited 2012 Sep 8];329(11):863–70. Available from: http://www.ncbi.nlm.nih.gov/pubmed/17067929 49 49. Mohamed HS, Ibrahim ME, Miller EN, White JK, Cordell HJ, Howson JMM, et al. SLC11A1 (formerly NRAMP1) and susceptibility to visceral leishmaniasis in The Sudan. Eur J Hum Genet [Internet]. 2004 Jan [cited 2012 Jul 24];12(1):66–74. Available from: http://www.ncbi.nlm.nih.gov/pubmed/14523377 50. Castellucci L, Jamieson SE, Almeida L, Oliveira J, Guimarães LH, Lessa M, et al. Wound healing genes and susceptibility to cutaneous leishmaniasis in Brazil. Infect Genet Evol [Internet]. Elsevier B.V.; 2012 Jul [cited 2012 Sep 8];12(5):1102–10. Available from: http://www.ncbi.nlm.nih.gov/pubmed/22554650 51. Cabrera M, Shaw MA, Sharples C, Williams H, Castes M, Convit J, et al. Polymorphism in tumor necrosis factor genes associated with mucocutaneous leishmaniasis. J Exp Med [Internet]. 1995 Nov 1 [cited 2012 Sep 8];182(5):1259– 64. Available from: http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=2192198&tool=pmce ntrez&rendertype=abstract 52. Castellucci L, Menezes E, Oliveira J, Magalhaes A, Guimaraes LH, Lessa M, et al. IL6 -174 G/C promoter polymorphism influences susceptibility to mucosal but not localized cutaneous leishmaniasis in Brazil. J Infect Dis [Internet]. 2006 Aug 15 [cited 2012 Sep 8];194(4):519–27. Available from: http://www.ncbi.nlm.nih.gov/pubmed/16845637 53. Fakiola M, Miller EN, Fadl M, Mohamed HS, Jamieson SE, Francis RW, et al. Genetic and functional evidence implicating DLL1 as the gene that influences susceptibility to visceral leishmaniasis at chromosome 6q27. J Infect Dis [Internet]. 2011 Aug 1 [cited 2012 Sep 8];204(3):467–77. Available from: http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=3132144&tool=pmce ntrez&rendertype=abstract 54. Frade AF, Oliveira LC De, Costa DL, Costa CHN, Aquino D, Van Weyenbergh J, et al. TGFB1 and IL8 gene polymorphisms and susceptibility to visceral leishmaniasis. Infect Genet Evol [Internet]. Elsevier B.V.; 2011 Jul [cited 2012 Aug 20];11(5):912–6. Available from: http://www.ncbi.nlm.nih.gov/pubmed/21376140 55. Mohamed HS, Ibrahim ME, Miller EN, Peacock CS, Khalil E a G, Cordell HJ, et al. Genetic susceptibility to visceral leishmaniasis in The Sudan: linkage and association with IL4 and IFNGR1. Genes Immun [Internet]. 2003 Jul [cited 2012 Jul 31];4(5):351–5. Available from: http://www.ncbi.nlm.nih.gov/pubmed/12847550 56. Ahir S, Chaudhari D, Chavan V, Samant-Mavani P, Nanavati R, Mehta P, et al. Polymorphisms in IL-1 gene cluster and its association with the risk of perinatal HIV transmission, in an Indian cohort. Immunol Lett [Internet]. Elsevier B.V.; 2013 Jun [cited 2014 Jun 18];153(1-2):1–8. Available from: http://www.ncbi.nlm.nih.gov/pubmed/23769826 50 57. Ferrero-Miliani L, Nielsen OH, Andersen PS, Girardin SE. Chronic inflammation: importance of NOD2 and NALP3 in interleukin-1beta generation. Clin Exp Immunol [Internet]. 2007 Feb [cited 2014 Sep 8];147(2):227–35. Available from: http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=1810472&tool=pmce ntrez&rendertype=abstract 58. Hollegaard M V, Bidwell JL. Cytokine gene polymorphism in human disease: online databases, Supplement 3. Genes Immun [Internet]. 2006 Jun [cited 2014 Jun 18];7(4):269–76. Available from: http://www.ncbi.nlm.nih.gov/pubmed/16642032 59. Haukim N, Bidwell JL, Smith a JP, Keen LJ, Gallagher G, Kimberly R, et al. Cytokine gene polymorphism in human disease: on-line databases, supplement 2. Genes Immun [Internet]. 2002 Sep [cited 2014 Jun 18];3(6):313–30. Available from: http://www.ncbi.nlm.nih.gov/pubmed/12209358 60. Silva MR, Sampaio A, Almeida A, Balseiro S, Santos P, Carvalho L. IDENTIFICAÇÃO DOS POLIMORFISMOS DOS GENES IL1B , IL1RN E TNFA NA GAS- TRITE CRÓNICA ASSOCIADA À INFECÇÃO POR HELICOBACTER PYLORI E NO. J Port Gastrenterol. 2008;15(1):8–14. 61. Walley AJ, Aucan C, Kwiatkowski D, Hill AVS. Interleukin-1 gene cluster polymorphisms and susceptibility to clinical malaria in a Gambian case-control study. Eur J Hum Genet [Internet]. 2004 Feb [cited 2014 Jun 19];12(2):132–8. Available from: http://www.ncbi.nlm.nih.gov/pubmed/14673470 62. Azad AK, Sadee W, Schlesinger LS. Innate immune gene polymorphisms in tuberculosis. Infect Immun [Internet]. 2012 Oct [cited 2014 May 28];80(10):3343– 59. Available from: http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=3457569&tool=pmce ntrez&rendertype=abstract 63. Fernández-Figueroa E a, Rangel-Escareño C, Espinosa-Mateos V, CarrilloSánchez K, Salaiza-Suazo N, Carrada-Figueroa G, et al. Disease severity in patients infected with Leishmania mexicana relates to IL-1β. PLoS Negl Trop Dis [Internet]. 2012 Jan [cited 2013 Mar 11];6(5):e1533. Available from: http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=3358333&tool=pmce ntrez&rendertype=abstract 64. Dinarello CA. Interleukin-1 beta, interleukin-18, and the interleukin-1 beta converting enzyme. Ann N Y Acad Sci [Internet]. 1998 Sep 29 [cited 2013 Mar 11];856:1–11. Available from: http://www.ncbi.nlm.nih.gov/pubmed/9917859 65. Weber A, Wasiliew P, Kracht M. Interleukin-1beta (IL-1beta) processing pathway. Sci Signal [Internet]. 2010 Jan [cited 2013 Mar 11];3(105). Available from: http://www.ncbi.nlm.nih.gov/pubmed/20086236 66. Lima-Junior DS, Costa DL, Carregaro V, Cunha LD, Silva ALN, Mineo TWP, et al. Inflammasome-derived IL-1β production induces nitric oxide-mediated 51 resistance to Leishmania. Nat Med [Internet]. 2013 Jul [cited 2014 Sep 9];19(7):909–15. Available from: http://www.ncbi.nlm.nih.gov/pubmed/23749230 67. Pétrilli V, Dostert C, Muruve D a, Tschopp J. The inflammasome: a danger sensing complex triggering innate immunity. Curr Opin Immunol [Internet]. 2007 Dec [cited 2013 Mar 5];19(6):615–22. Available from: http://www.ncbi.nlm.nih.gov/pubmed/17977705 68. Oikawa T, Yamada T. Molecular biology of the Ets family of transcription factors. Gene [Internet]. 2003 Jan 16;303:11–34. Available from: http://www.ncbi.nlm.nih.gov/pubmed/12559563 69. Asano Y, Markiewicz M, Kubo M, Szalai G, Watson DK, Trojanowska M. Transcription factor Fli1 regulates collagen fibrillogenesis in mouse skin. Mol Cell Biol [Internet]. 2009 Jan [cited 2013 Apr 5];29(2):425–34. Available from: http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=2612518&tool=pmce ntrez&rendertype=abstract 70. Castellucci L, Jamieson SE, Miller EN, de Almeida LF, Oliveira J, Magalhães a, et al. FLI1 polymorphism affects susceptibility to cutaneous leishmaniasis in Brazil. Genes Immun [Internet]. 2011 Oct [cited 2012 Oct 11];12(7):589–94. Available from: http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=3297968&tool=pmce ntrez&rendertype=abstract 71. De Moura TR, Novais FO, Oliveira F, Clarêncio J, Noronha A, Barral A, et al. Toward a novel experimental model of infection to study American cutaneous leishmaniasis caused by Leishmania braziliensis. Infect Immun [Internet]. 2005 Sep [cited 2013 May 6];73(9):5827–34. Available from: http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=1231065&tool=pmce ntrez&rendertype=abstract 72. Rovin BH, Lu L, Saxena R. A novel polymorphism in the MCP-1 gene regulatory region that influences MCP-1 expression. Biochem Biophys Res Commun [Internet]. 1999 Jun 7 [cited 2013 May 6];259(2):344–8. Available from: http://www.ncbi.nlm.nih.gov/pubmed/10362511 73. Ramasawmy R, Cunha-Neto E, Fae KC, Martello FG, Müller NG, Cavalcanti VL, et al. The monocyte chemoattractant protein-1 gene polymorphism is associated with cardiomyopathy in human chagas disease. Clin Infect Dis [Internet]. 2006 Aug 1 [cited 2013 May 6];43(3):305–11. Available from: http://www.ncbi.nlm.nih.gov/pubmed/16804844 74. Sambrook, J.; Fritsch, EF.; Maniatis, T. Molecular Cloning: A Laboratory Manual. Cold Spring Harbor Laboratory Press; Cold Spring Harbor, New York: 1989. 75. Quilez J, Martínez V, Woolliams J a, Sanchez A, Pong-Wong R, Kennedy LJ, et al. Genetic control of canine leishmaniasis: genome-wide association study and genomic selection analysis. PLoS One [Internet]. 2012 Jan [cited 2012 Sep 8];7(4):e35349. Available from: 52 http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=3338836&tool=pmce ntrez&rendertype=abstract 76. Benicio E de A, Gadelha EPN, Talhari A, Silva RM da, Ferreira LC, Santos MCC dos, et al. Combining diagnostic procedures for the management of leishmaniasis in areas with high prevalence of Leishmania guyanensis. An Bras Dermatol [Internet]. 2011 [cited 2014 Sep 26];86(6):1141–4. Available from: http://www.ncbi.nlm.nih.gov/pubmed/22281902 77. Romero GAS, Ishikawa E, Cupolillo E, Toaldo CB, Guerra MVF, Paes MG, et al. The rarity of infection with Leishmania (Viannia) braziliensis among patients from the Manaus region of Amazonas state, Brazil, who have cutaneous leishmaniasis. Ann Trop Med Parasitol [Internet]. 2002 Mar [cited 2013 Mar 30];96(2):131–6. Available from: http://www.ncbi.nlm.nih.gov/pubmed/12080973 78. Park BL, Kim YJ, Cheong HS, Kim LH, Choi YH, Lee H-S, et al. Association of common promoter polymorphisms of MCP1 with hepatitis B virus clearance. Exp Mol Med [Internet]. 2006 Dec 31;38(6):694–702. Available from: http://www.ncbi.nlm.nih.gov/pubmed/17202846 79. Flores-villanueva PO, Ruiz-morales JA, Song C, Flores LM, Jo E, Montaño M, et al. ART I CL E A functional promoter polymorphism in monocyte chemoattractant protein – 1 is associated with increased susceptibility to pulmonary tuberculosis. 2005;202(12):1649–58. 80. Frade AF, Pissetti CW, Ianni BM, Saba B, Lin-Wang HT, Nogueira LG, et al. Genetic susceptibility to Chagas disease cardiomyopathy: involvement of several genes of the innate immunity and chemokine-dependent migration pathways. BMC Infect Dis [Internet]. 2013 Jan;13:587. Available from: http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=3866603&tool=pmce ntrez&rendertype=abstract 81. Ksiaa Cheikh Rouhou L, Gorgi YL, Skhiri HA, Aouadi H, Ayed SJ, Sfar I, et al. Chemokine and chemokine receptor gene polymorphism in Tunisian hemodialysis patients with HCV infection. Arab J Nephrol Transplant [Internet]. 2011 Sep [cited 2014 Sep 23];4(3):117–24. Available from: http://www.ncbi.nlm.nih.gov/pubmed/22026334 82. Sakthianandeswaren A, Curtis JM, Elso C, Kumar B, Baldwin TM, Lopaticki S, et al. Fine mapping of Leishmania major susceptibility Locus lmr2 and evidence of a role for Fli1 in disease and wound healing. Infect Immun [Internet]. 2010 Jun [cited 2014 Sep 20];78(6):2734–44. Available from: http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=2876540&tool=pmce ntrez&rendertype=abstract 83. Castellucci LC, Almeida LF De, Jamieson SE, Fakiola M, Carvalho EM De, Blackwell JM. Host genetic factors in American cutaneous leishmaniasis: a critical appraisal of studies conducted in an endemic area of Brazil. Mem Inst Oswaldo Cruz [Internet]. 2014 May 27 [cited 2014 Sep 20];00(ahead):00–00. 53 Available from: http://www.scielo.br/scielo.php?script=sci_arttext&pid=S007402762014005040028&lng=en&nrm=iso&tlng=en 84. Moravej A, Rasouli M, Kalani M, Asaei S, Kiany S, Najafipour S, et al. IL-1β (511T/C) gene polymorphism not IL-1β (+3953T/C) and LT-α (+252A/G) gene variants confers susceptibility to visceral leishmaniasis. Mol Biol Rep [Internet]. 2012 Jun [cited 2014 Sep 24];39(6):6907–14. Available from: http://www.ncbi.nlm.nih.gov/pubmed/22311026 85. Pontillo A, Oshiro TM, Girardelli M, Kamada AJ, Crovella S, Duarte AJS. Polymorphisms in inflammasome’ genes and susceptibility to HIV-1 infection. J Acquir Immune Defic Syndr [Internet]. 2012 Feb 1;59(2):121–5. Available from: http://www.ncbi.nlm.nih.gov/pubmed/22227487 54 8. ANEXOS Anexo A – Fluxograma do Estudo Recrutamento de pacientes e indivíduos controle Entrevista e coleta de material Extração e quantificação de DNA Genotipagem de IL-1b Genotipagem de FLI-1 Processamento dos dados Análise Estatística Genotipagem de MCP-1 55 Anexo B – Termo de Consentimento Livre e Esclarecido Número do Prontuário:_____________ Número da Ficha:______________ TERMO DE CONSENTIMENTO LIVRE E ESCLARECIDO POLIMORFISMOS GENÉTICOS DOS GENES ENVOLVIDOS NA RESPOSTA IMUNE E NA CICATRIZAÇÃO DAS LESÕES EM PACIENTES COM LEISHMANIOSE CUTÂNEA EM UMA POPULAÇÃO CASO-CONTROLE DE MANAUS, AMAZONAS. Introdução: Você está sendo convidado para participar do projeto de pesquisa citado acima. Este estudo será coordenado pelo Dr. Rajendranath RAMASAWMY pesquisador visitante sênior e professor permanente do programa de Pós-Graduação em Doenças Tropicais e Infecciosas da UEA/FMT-HVD. Antes de tomar qualquer decisão, é importante que você leia e compreenda as seguintes explicações sobre o procedimento proposto. Esta declaração descreve o objetivo, procedimento, benefícios e riscos do estudo, e o seu direito de sair do estudo a qualquer momento. Estas informações estão sendo dadas para esclarecer quaisquer dúvidas sobre a pesquisa proposta, antes de obter o seu consentimento. Justificativa: Leishmaniose tegumentar ou ferida braba é uma doença causada por um pequeno parasita que fica na pele, mas existem diferentes espécies de parasito que podem causar a mesma doença, assim sendo, é importante saber qual o tipo de parasito que pode estar causando sua doença, ou se você não teve essa doença queremos saber se você tem mais possibilidade de contraí-la de que outra pessoa. Dessa forma, queremos que você participe deste estudo permitindo que seja retirada uma pequena amostra de sangue da sua veia pra fazermos testes que vão nos dizer se você tem mais chance de ter leishmaniose causada pela parasita Leismania guyanensis do que outra pessoa. Caso você tenha essa doença queremos saber se você tem no seu organismo (na parte genética) capacidade de boa cicatrização, após ter sido tratado com um medicamento específico para essa doença. Métodos: Caso você concordar em participar desse estudo, você será submetido a um exame médico, e uma coleta de 5 mL de sangue do seu antebraço, com uma agulha nova descartável, após a assepsia local (limpeza). Se o médico suspeitar que você tenha leishmaniose, você poderá ser submetido ainda a uma coleta para biópsia de pele realizada por um dos médicos (Dr. Jorge Guerra ou Dra Anette Talhari) integrantes da equipe. Os indivíduos não afetados por leishmaniose e sem histórico de leishmaniose, que procurem o ambulatório de dermatologia da FMT-HVD, serão selecionados para e, caso aceitem participar, comporão o grupo controle. Os indivíduos que, depois de examinados pelo dermatologista, forem caracterizados como não afetados por leishmaniose, e que não apresentarem história de infecções crônicas, inflamação e doenças auto-imunes, serão considerados candidatos. A biópsia será utilizada para identificação do parasita que está causando sua doença. A biópsia é um procedimento no qual se colhe uma pequena quantidade de pele, isto é, uma amostra, de tecido ou células, para posterior estudo em laboratório. A grande maioria dos pequenos procedimentos de biópsia é muito segura, tendo apenas um pequeno risco de sangramento ou infecção no local da biópsia. Ela é feita após anestesia local na borda local da ferida e depois de isso retirar um pequeno pedaço de pele com auxilio de instrumento cortante esterilizado chamado de punch. A amostra de sangue servirá para saber o que queremos através de testes feitos no laboratório, alem disso, com sua autorização, informações de prontuários clínicos também poderão ser lidas pelos participantes do estudo para comparar o resultado dos testes feitos em seu sangue e a resposta de seu tratamento. 1) Local do estudo Os procedimentos descritos acima serão realizados no ambulatório de Dermatologia (avaliação clínica e biópsia) sob a responsabilidade do Dr. Jorge Guerra ou Dra. Anette Talhari, médicos integrantes da equipe de pesquisa e nos laboratórios da FMT-HVD sob a responsabilidade do Dr. Rajendranath Ramasawmy. Rubrica do pesquisador:_________________ Rubrica do participante:__________________ 56 Número do Prontuário:___________________ Número da Ficha:_______________________ 2) Permissão para estocagem Está sendo solicitada a sua permissão para estocagem (guarda ou armazenamento) de sua amostra de biópsia, soro e de DNA na FMT-HVD sob a responsabilidade do Dr. Rajendranath Ramasawmy de acordo com a resolução CNS Nº441de 12 de maio de 2011 e da portaria Nº00212/2012-GDP/FMT-HVD. Se assinar esse termo de consentimento, você está autorizando estocagem de longo prazo das amostras para estudos futuros. Isso evitará procedimentos como nova coleta de sangue bem como a diminuição de recursos financeiros para novos procedimentos. A sua amostra de DNA será mantida indefinidamente. Isto significa que a sua amostra não será destruída após um determinado período de tempo, mas sim será estocada pelo tempo que ela durar. Amostras estocadas serão usadas exclusivamente para fins de pesquisas e poderão ser utilizadas para outros estudos a respeito da susceptibilidade à leishmaniose. O uso de sua amostra estocada terá como condição uma nova avaliação e aprovação do projeto de pesquisa pelo Comitê de Ética pertinente. Sua permissão será solicitada. Se você concorda, você será pedir de assinar um novo TCLE com as justificativas e especificidades. 3) Risco físico para saúde/desconfortos Os riscos físicos para a saúde na participação deste estudo são limitados ao procedimento de biópsia e coleta de sangue, ambos de rotina nos laboratórios clínicos. Em ambos os casos, você poderá sentir um desconforto temporário devido à introdução da agulha. A grande maioria dos procedimentos de biópsia é muito segura, tendo apenas um risco de sangramento ou infecção no local da biópsia. Ela é feita após anestesia local na borda local da ferida e depois de isso retirar um pequeno pedaço de pele com auxilio de instrumento cortante esterilizado chamado de punch. Durante a realização de biopsia, pode ocorrer pequeno sangramento que acostuma regredir em seguida. Pode também acontecer infecção se não haver cuidado de limpeza durante e depois desse procedimento. De qualquer maneira, você receberá uma pomada de antibiótico para evitar infecção. Entretanto, caso você tenha febre ou dor, "inchaço" (edema), rubor ou sangramento no local de biópsia deve procurar a equipe da pesquisa (Dr. Jorge Guerra ou Dra Anette Talhari). Existe também a possibilidade de risco de perda da confidencialidade tal como outras pessoas poderiam ter acesso aos seus dados e identificá-lo. Entretanto, serão tomados cuidados especiais para que isso não aconteça pois, sua identificação será feita por meio códigos. O seu nome não estará visível nos frascos contendo as amostras biológicas, elas serão codificadas para evitar o risco de perda da confidencialidade. 4) Indenização e compensação por danos Se você desenvolver uma infecção localizada devido ao procedimento de coleta de sangue ou biópsia, você será assistido pelo Dr. Jorge Guerra ou a Dra Anette Talhari, médicos integrantes da equipe de pesquisa no FMT-HVD. O custo desse tratamento será totalmente coberto pelo projeto. Qualquer dano decorrente de sua participação somente neste estudo referente a qualquer procedimento relacionado, você será assistido na FMT-HDV e terá direito a indenizações ou ressarcimentos em casos de danos decorrentes de sua participação no estudo. 5) Desligamento A sua participação neste estudo é voluntária e sua recusa em participar ou seu desligamento do estudo não envolverá penalidades ou perda de benefícios os quais você tenha direito. Se você estiver afetado pela leishmaniose, acesso a procedimentos médicos para diagnósticos e tratamento da doença será providenciado mesmo que não queira participar deste estudo. Se você não estiver afetado pela leishmaniose, você está sendo convidado a participar do estudo como parte do grupo controle ou como familiar do afetado. Neste caso, sua decisão de participar ou não, ou de cessar sua participação a qualquer momento, não irá interferir de nenhuma forma nos procedimento médicos para diagnóstico ou tratamento da leishmaniose que você possa necessitar no futuro. Da mesma forma, sua decisão não irá refletir no acesso a procedimentos médicos necessários a algum familiar ou contato afetado pela leishmaniose. Rubrica do pesquisador: __________________ Rubrica do participante:__________________ 57 Número do prontuário:____________________ Número da Ficha:_______________________ 6) Custo para os participantes No caso de você decidir participar do estudo, você não terá nenhum custo. Custos com testes laboratoriais e análises de suas amostras na pesquisa serão cobertos pelo estudo. 7) Benefícios Em longo prazo, os procedimentos médicos e laboratoriais aos quais você será submetido poderão facilitar a detecção de resistência à leishmaniose e seu tratamento, tornando possível evitar tratamentos inadequados. Além disso, espera-se que conhecimentos científicos adicionais sejam alcançados, com consequente melhoria do tratamento de pessoas afetadas pela leishmaniose. 8) Reembolso Já que não haverá gastos adicionais de transporte e alimentação devido a sua participação no estudo, você não será reembolsado por participar deste estudo. 9) Exclusividade de uso de material genético e biológico Amostras de DNA serão utilizadas apenas para pesquisa de susceptibilidade à leishmaniose. Todos os resultados obtidos no estudo, após análise do conjunto completo dos dados, serão publicados em artigos científicos. É importante reafirmar que o alvo de nossos estudos é a identificação do fator de risco genético à leishmaniose e contribuição no entendimento do mecanismo da doença 10) Confidencialidade dos dados Os registros de sua participação neste estudo serão mantidos confidencialmente até onde é permitido por lei e todas as informações estarão restritas à equipe responsável pelo projeto. Nenhuma informação genética individual será tornada pública. As informações serão codificadas e mantidas em local protegidas o tempo todo. Somente os pesquisadores envolvidos neste estudo terão acesso às informações. Após o término deste estudo, as informações serão transcritas dos questionários para os arquivos de computador, mantidos em local restrito com acesso permitido apenas aos mesmos pesquisadores. Os dados deste estudo poderão ser discutidos com pesquisadores de outras instituições, mas nenhuma identificação será fornecida. Você tenha direto de conhecer os resultados dos testes laboratoriais que serão realizados. Os resultados da pesquisa em nenhum momento irão interferir no tratamento já preconizado. POLIMORFISMOS GENÉTICOS DOS GENES ENVOLVIDOS NA RESPOSTA IMUNE E NA CICATRIZAÇÃO DAS LESÕES EM PACIENTES COM LEISHMANIOSE CUTÂNEA EM UMA POPULAÇÃO CASO-CONTROLE DE MANAUS, AMAZONAS. Você receberá uma cópia deste Termo de Consentimento para mantê-lo consigo. Se você tiver qualquer dúvida no futuro sobre sua participação neste estudo, você pode e deve utilizar os seguintes meios de contato com os pesquisadores responsáveis: Dra Anette TALHARI Dr Jorge GUERRA Dr Rajendranath RAMASAWMY Telefone: (92) 2127 3429 (92) 2127 3429 (92) 2127 3447 Email: [email protected] [email protected] [email protected] Para quaisquer informações, fica disponibilizado o endereço do CEP/FMT-HVD, sito à Av. Pedro Teixeira nº25 Dom Pedro I, Cep 69040-000, Manaus-AM, que funciona de 2ª a 6ª feira, das 08:00 às 14:00 horas, telefone (92)2127-3572, e-mail: [email protected] 58 Consentimento Li e entendi as informações precedentes. Tive a oportunidade de fazer perguntas e todas as minhas dúvidas foram respondidas a contento. Este formulário está sendo assinado voluntariamente por mim, indicando o meu consentimento em participar do estudo, até que eu decida o contrário. Assinatura ou impressão digital do voluntário Nome completo e nº do prontuário Assinatura do entrevistador Nome do entrevistador Assinatura testemunha 1 Data: ____/____/_____ Assinatura testemunha 2 59 Anexo C – Questionário FMT-HVD Fundação de Medicina Tropical Dr Heitor Vieira Dourado Instrumento de Coleta de Dados Projeto Genética da Leishmaniose Identificação do voluntário: 1. 2. 3. 4. 5. Nome: ___________________________________________________________ Gênero: Masc ( ) Fem ( ) D/N:___/___/_____ Idade:_____________ Etnia: Branco: ( ); Misto: ( ); Índio: ( ); Outro(qual):_____________________________ 6. Tipo e local de trabalho:_____________________________________________ Dados para contato: 1.Telefone:_______________________________________ 2. Endereço:______________________________________________________ ________________________________________________________________ 3. Referência:_____________________________________________________ 4. Tempo de Residência:______________________________ 5. Histórico de Residência (onde já morou e por quanto tempo):_____________ ________________________________________________________________ Dados Clínicos: 7. Já teve Leishmaniose? Sim ( ) Não ( ) a. Se Sim ( grupo caso): i. Caso ativo ou histórico? Ativo ( ) Histórico ( ) ii. Teve mais de uma vez (se sim, responder iii a viii para cada episódio): Sim ( ) Não ( ) iii. Data de diagnóstico:____/____/_______ iv. Forma clínica: cutânea ( ) Mucosa ( ) v. Local provável de infecção:____________________________________ vi. Tratamento:_________________________________________________ vii. Resposta ao tratamento: Sim ( ) Não ( ) viii. Se caso histórico – cicatriz? Sim ( ) Não ( ) b. Se não (grupo controle) - confirmado por ausência de cicatriz 8. Já teve malária? Sim ( ) Não ( ) a. Se Sim quantas:_______________________ 9. Já teve outras doenças? _____________________________________________ 10. Prontuário da FMT-HVD: __________________________ 60 Anexo D – Protocolo de Extração a) Após centrifugação das amostras de sangue total para separação do plasma, o “buffy-coat” será retirado e transferido para microtubo de 2,0 mL. b) Ao “buffy-coat” serão adicionados 160 µL de tampão de proteinase K 5X (NaCl 5M; EDTA 0,5M pH 8,0), 40 µL de proteinase K, 40 µL de SDS 20% e 300 µL de H 2O destilada. Em seguida, os tubos serão incubados em banho-maria a 37°C durante toda a noite. Retirar os tubos do banho-maria e esperar resfriar a temperatura ambiente. c) Adicionar 200 µL de NaCl 6M para precipitação de proteínas, centrifugando-se em seguida a 13.000 rpm por 20 minutos, e após, transferir o sobrenadante para um microtubo de 1,5 mL. Centrifugações adicionais (13.000 rpm durante 10 e 5 minutos respectivamente) serão realizadas até a remoção completa das proteínas residuais, sendo o sobrenadante dividido em dois novos tubos de 1,5 mL. d) Adicionar em cada tubo 900 µL de etanol 99,5% (temperatura ambiente). Deixar o DNA precipitar invertendo o tubo gentilmente por, no máximo, 2 minutos. Após esta etapa, o DNA precipitado será lavado mais uma vez com etanol a 70%. e) Após remover o etanol, com as paredes do tubo já secas, ressuspender o DNA em 200 µL de H 2O destilada. f) Em seguida, realizar leitura para quantificação do DNA, e aliquotar as amostras, armazenando-as em temperatura de -70 °C. 61 ANEXO E – Minuta de Artigo Associação Genética de Polimorfismos em IL-1b e FLI-1 com Susceptibilidade para Leishmaniose Cutânea numa População Caso-Controle do Estado do Amazonas Karolina da Costa Sabino1, Luciane Macedo de Souza1, Priscila Bentes Sousa1, Rajendranath Ramasawmy2. 1Mestre em Doenças Tropicais e Infecciosas - Programa de Pós-Graduação em Medicina Tropical; Universidade do Estado do Amazonas/ Fundação de Medicina Tropical Dr. Heitor Vieira Dourado. 2Doutor em genética, Pesquisador da Fundação de Medicina Tropical Doutor Heitor Vieira Dourado. Resumo Nas áreas onde a leishmaniose é considerada endêmica, somente uma fração da população exposta ao parasita desenvolve a doença, sugerindo que fatores genéticos tenham um papel fundamental nesse contexto. Esse estudo teve como objetivo verificar o papel de polimorfismos genéticos em IL-1b e FLI-1 associados com leishmaniose cutânea (LC) em uma amostra populacional do estado do Amazonas. Amostras de 774 indivíduos, sendo 392 indivíduos com leishmaniose cutânea e 382 indivíduos sem a doença foram genotipadas para os SNPs IL-1b_rs1143634, IL1b_rs16944, FLI1_rs619521, FLI1_rs531894 e FLI1_rs7930515 pela técnica de PCR-RFLP. Não foram encontradas diferenças significativas nas frequências alélicas e genotípicas para os SNPs IL-1b_rs1143634, FLI1_rs619521 e FLI1_rs531894 entre os grupos estudados. No entanto, para o SNP IL1b_rs16944, foram detectadas diferenças significativas na distribuição alélica e genotípica do polimorfismo entre os grupos caso e controle. Para o SNP rs7930515 de FLI-1 foi observada uma tendencia à associação para LC (P=0.036). Os resultados obtidos no presente estudo reforçam o papel dos genes IL1-b e FLI-1 associados com resistência ou susceptibilidade a LC. Introdução A leishmaniose, causada por um parasita celular obrigatório pertencente ao gênero Leishmania, representa um grupo de doenças com um amplo espectro clínico e diversidade epidemiologica (1,2). As manifestações variam de úlceras cutâneas leves à forma letal, com acometimento visceral (2–4). No estado do Amazonas, as formas mais comuns de manifestação são leishmaniose cutânea (LC) e mucosa (LM), que são causadas principalmente por Leishmania guyanensis. Em áreas endêmicas para leishmaniose, apenas uma pequena fração dos indivíduos expostos desenvolve a doença, sugerindo que fatores 62 genéticos tenham um papel fundamental nesse contexto, somado a a fatores ambientais, genética do hospedeiro e cepa do parasita possam estar envolvidos na susceptibilidade à doença (5–7). Nos últimos anos, muito se tem focado na função dos polimorfismos de nucleotídeo único (SNPs) em citocinas e sua associação com doenças em humanos (8). A interleucina-1 (IL-1) pertence ao grupo de citocinas pro-inflamatórias (IL-1) do tipo Th1, e tem papel chave na resposta inflamatória com efeitos pleiotrópicos em muitas células e vias de sinalização, tendo um papel importante no desenvolvimento da patologia. IL-1 consiste de um grupo de três moléculas nomeadas IL-1 alfa, IL-1 beta e IL-1 receptor antagonista (9). Polimorfismos genéticos em IL-1b estão associados com a susceptibilidade a doenças autoimunes graves, inflamatórias e infecciosas tais como HIV, TB, gastrite crônica associada com Helicobacter pylori, esclerose múltipla, lupus eritematoso sistêmico, malária e leishmaniose (8,10–14). Polimorfismo em IL-1b -511 C/T foi associado com risco para desenvolver a doença em pacientes infectados por L. mexicana (14). Outro importante gene envolvido no controle da susceptibilidade da leishmaniose e que apresenta participação no processo de cura das lesões é o FLI-1. Pertencente à família ETs de fatores de transcrição, é preferencialmente expresso em linhagens de células hematopoiéticas e células endoteliais vasculares (15). Resultados apresentados por Asano et al. estabelecem o gene FLI-1 como um regulador chave na homeostase da expressão de colágeno na pele, e, portanto, pode atuar na via de cicatrização das lesões. Além disso, o estudo demonstra que o domínio C-terminal do FLI-1 tem um papel essencial no controle da expressão de colágeno in vivo (16). Ainda, Castellucci et al. demonstraram associação entre polimorfismos em FLI-1 e susceptibilidade para leishmaniose cutânea causada por L. braziliensis (17). A importância de IL-1b e FLI-1 na resposta imune sugere que polimorfismos nesses genes podem influenciar na resistência ou susceptibilidade de um indivíduo para desenvolver a doença. Dessa maneira, SNPs em IL-1b e FLI-1 foram investigados com associação para LC em uma população caso-controle do estado do Amazonas. Metodologia 63 - Pacientes e controles Para análise dos SNPs IL-1b_rs1143634, IL1b_rs16944, FLI1_rs619521, FLI1_rs531894 e FLI1_rs7930515, um total de 392 indivíduos com a manifestação clínica da doença para compor o grupo caso, recrutados exclusivamente no ambulatório de dermatologia da FMT-HVD por demanda espontânea. Foram admitidos apenas pacientes com leishmaniose cutânea que tiveram seu diagnóstico confirmado através do exame de escarificação da lesão; e 382 indivíduos sem sinais e sintomas de leishmaniose cutânea para o grupo controle, provenientes de áreas endêmicas adjacentes às rodovias AM-010 e BR-174, de onde procedeu a maioria dos indivíduos do grupo caso. - Coleta da amostra e extração de DNA Amostras de sangue periférico (5 mL) foram coletadas de pacientes e controles em tubo contendo ácido etilenediaminetetraacetico (EDTA) como anticoagulante. O DNA genômico foi extraído a partir do creme leucocitário com o kit comercial de extração de DNA (WIZARD®, Promega Corporation, EUA), de acordo com as instruções sugeridas pelo fabricante ou pela técnica de “salting-out”, conforme protocolo de Sambrook et al. com algumas modificações(18). - Genotipagem A genotipagem foi realizada pela técnica de PCR-RFLP (Reação em Cadeia da Polimerase - Análise clássica de comprimento do fragmento de restrição) para descriminação alélica. Foi realizada a amplificação das sequências gênicas que incorporam as regiões de mutação utilizando-se a técnica de PCR Convencional. Os pares de iniciadores usados, bem como as enzimas de restrição, os produtos de PCR, Temperatura de anelamento e concentração de MgCl2 estão descritos na Tabela 1. A PCR foi realizada num volume final de 25 µL contendo 50 ng de DNA genômico, 1X PCR buffer, MgCl2, 40uM de dNTP, 0,2 µM de cada iniciador e 1U de Taq polimerase. As condições de PCR incluíram um passo inicial de desnaturação por 5 min a 94ºC, seguido de 40 ciclos de desnaturação por 30s a 94ºC, anelamento por 30s à temperatura específica, extensão por 30s a 72ºC; e uma extensão final por 7 min. Um 64 volume de 10µL de PCR foi digerido com a enzima de restrição correspondente. Os fragmentos foram identificados em gel de agarose 2% contendo brometo de etídio sob luz ultravioleta. - Análise Estatística Foi realizado o cálculo das frequências de cada polimorfismo nos grupos caso e controle e analisou-se o valor de P pelo teste exato de Fisher e X2 através do programa estatístico SPSS, considerando um valor de p<0.05 como estatisticamente significativo. Tabela 1. Condições gerais usadas na reação de PCR-RFLP. MARCADOR INICIADORES Ta (°C) MgCl2 (mM) PRODUTO FRAGMENTOS ENZIMA DE PCR DA DIGESTÃO IL1b rs16944 (C/T) F: 5' TGG CAT TGA TCT GGT TCA TC 3' R: 5' GTT TAG GAA TCT TCC CAC TT 3' 53 2,0 305 pb AvaI C: 190/115 T: 305 IL1b rs1143634 (C/T) F: 5' GTT GTC ATC AGA CTT TGA CC 3' R: 5' TTC AGT TCA TAT GGA CCA GA 3' 53 2,0 250pb TaqαI C: 136/114 T: 250 FLI1 rs7930515 (A/C) F: 5' GGG TAA GTG GCC TCG TTG CAC GAG 3' R: 5' GGC GCT AGG GAT CTG TGC GGA CGG 3' 62 4,0 243 pb BfaI A: 139/97/7 C: 236/7 FLI1 rs619521 (A/G) F: 5' AGC TAG GCA CAG GTG CAC CTA GAC 3' R: 5' CCT GAA GGT GGG TGC CTT GGT TTC 3' 62 4,0 324pb Pvu II G: 180/144 A: 324 FLI1 rs531894 (A/G) F: 5' TAA GGA AGT GAA TAT CCC AGT CCT GTT 3' R: 5' ATG AGG AAG GTG TCT ACC TTC CTG 3' 62 3,2 286pb Alu I G: 147/97/42 A: 244/42 Resultados - Características Gerais da População em Estudo Foram recrutados 392 indivíduos com a manifestação clínica da doença para compor o grupo caso. Desse total, 311 (79%) eram do masculino e 80 (20%) do sexo feminino. A idade teve uma variação de 5-70 anos de idade com média de 32,0 anos. Adicionalmente, analisou-se a média de idade de acordo com o sexo, tendo-se 65 observado a mesma média (32,0 anos) tanto para o gênero masculino quanto feminino. Para compor o grupo controle, foram recrutados 382 indivíduos sem sinais e sintomas de leishmaniose cutânea, sendo 225 (59%) do sexo masculino e 157 (41%) pertencente ao feminino. A idade dos indivíduos do grupo controle variou de 5-89 anos de idade, com uma média de 36,0 anos. Novamente, analisou-se a média de idade associada ao gênero, obtendo-se média de 38,0 anos para o gênero masculino e 34,0 anos para o gênero feminino. A tabela 2 sumariza a análise descritiva da população caso-controle quanto ao sexo e idade. Tabela2. Características gerais da população caso-controle em estudo. Sexo (%) Masculino Feminino Dado faltante Média de idade – Anos (± DP) Quanto ao sexo Masculino Feminino CASO CONTROLE n= 392 311 (79) 80 (20) 1 (3) n= 382 225 (59) 157(41) ... 32 ± 14.1 36 ± 17.7 32 ± 13.7 32 ± 15.5 38 ± 17.6 34 ± 17.5 DP = Desvio Padrão - Genotipagem, HWE e DL Os marcadores analisados apresentaram-se em HWE, com exceção do marcador FLI1_rs145675082, que por não estar em equilíbrio foi excluído da análise. Quanto à genotipagem, esta foi realizada por PCR-RFLP conforme ilustrado nas figuras 1 e 2; e apresentou êxito ≥90% para todos os marcadores testados. 66 1 2 Figura 1. Padrão eletroforético da PCR-RFLP para os SNPs IL1-B +3953 C/T e IL1-B -511 C/T respectivamente. M: marcador de 100 pb; Figura 2. Padrão eletroforético da PCR-RFLP para os SNPs FLI1_rs7930515, FLI1_rs619521 e FLI1_rs531894 respectivamente. M: marcador de 100 pb; Análise para verificar o DL entre os SNPs dos genes IL1B e FLI-1 foi realizada utilizando-se para cálculo o parâmetro r2, conforme ilustra a figura 3. A análise do DL confirmou independência para todos os SNPs testados. As frequências alélicas e genotípicas para os marcadores IL1b_rs1143634 e IL1b_16944 do gene IL-1b estão descritas na tabela 3. Conforme análise, não foi observada evidência de associação do marcador IL1b_rs1143634 com LC na amostra populacional estudada, pois não foram observadas diferenças estatisticamente significativas nas frequências alélicas e genotípicas quando foram comparados os grupos caso e controle para os três modelos de comparação que foram aplicados (genotípico, alélico e de dominância), todos com valores superiores a 0.05. 67 A B r2 r2 Figura 3. Análise de desequilíbrio de ligação entre os SNPs dos genes IL-1b e FLI1 respectivamente. Para o SNP IL1b_16944, análise da distribuição das frequências alélica, genotípica e de dominância foram aplicadas entre os grupos caso e controle, observando-se evidência positiva de associação desse SNP (modelo aditivo P=0.026 e dominante P=0.027) com LC na amostra estudada. Tabela 3. Comparação das distribuições das frequências genotípicas e alélicas para os marcadores IL1b_rs16944 e IL1b_rs1143634. GRUPO CASO n= 374 (%) GRUPO CONTROLE n= 352 (%) 81 (22) 54 (15) CT 183 (49) 167 (48) TT 110 (29) 131 (37) CC + CT TT 264 (71) 110 (29) 221 (63) 131 (37) CT + TT CC Alelo C T IL1b_rs1143634 Genótipo CC 293 (78) 81 (22) IL1b_rs16944 Genótipo CC P X2 OR (IC 95%) 0.026 ... ... Dominância CT TT Alelo C T 0.027 1.42 [1.04 – 1.94] 298 (85) 54 (15) 0.036 0.66 [0.45 – 0.96] 345 (46) 403 (54) n= 388 (%) 275 (39) 429 (61) n= 371 (%) 0.007 1.34 [1.084-1.646] 296 (76) 282 (76) 85 (22) 7 (2) 78 (21) 11 (3) 677 (87) 99 (13) 642 (87) 100 (13) 1.15 0.56 ... 0.17 0.68 ... 68 Para o gene FLI1, não foram observadas evidências de associação dos marcadores FLI1_rs531894 e FLI1_rs619521 com LC na amostra populacional estudada (P>0.05) para os três modelos de comparação que foram aplicadas (genotípico, alélico e de dominância). Tabela 4. Comparação das distribuições das frequências genotípicas e alélicas para os marcadores FLI1_rs7930515, FLI1_rs531894 e FLI1_rs619521. GRUPO CASO GRUPO CONTROLE n= 347 (%) n= 335 (%) AA 9 (3) 20 (6) AC 144 (41) 142 (42) CC 194 (56) 173 (52) Dominância AA + AC CC 153 (44) 194 (56) 162 (48) 173 (52) 338 (97) 9 (3) 315 (94) 20 (6) 345 (46) 403 (54) n= 354 (%) 275 (39) 429 (61) n= 316 (%) 131 (37) 169 (48) 54 (15) 114 (36) 160 (51) 42 (13) 0.77 0.68 ... 431 (61) 277 (39) 388 (61) 244 (39) 0.19 0.66 ... n= 337 (%) n= 318 (%) 87 (26) 156 (46) 94 (28) 76 (24) 154 (48) 88 (28) 0.40 0.82 ... 330 (49) 344 (51) 306 (48) 330 (52) 0.09 0.76 ... FLI1_rs7930515 P X2 OR (IC 95%) Genótipo AC + CC AA Alelo A C FLI1_rs531894 Genótipo AA AG GG Alelo A G FLI1_rs619521 Genótipo AA AG GG Alelo A G 0.075 ... 0.28 0.84 [0.62 – 1.14] 0.036 2.38 [1.07 – 5.32] 0.10 1.23 [0.96 – 1.56 ] 69 Para o SNP FLI1_rs7930515 também foram aplicadas análises da distribuição das frequências alélica, genotípica e de dominância entre os grupos caso e controle, podendo-se observar valores de P “boderline” (modelo aditivo P=0.075 e dominante P=0.036), com OR de 2.38 [IC 95% 1.07 – 5.32], conforme tabela 4. Discussão As leishmanioses ainda são consideradas um grande problema de saúde pública, representando um complexo de doenças com importante espectro clínico e diversidade epidemiológica. Nas áreas onde a leishmaniose é considerada endêmica, somente uma fração da população exposta ao parasita desenvolve a doença (4,19,20). Como a leishmaniose é uma doença infecciosa cuja susceptibilidade é em parte controlada geneticamente, entende-se que esforços no sentido de se elucidar os mecanismos moleculares de controle e interação patógeno/hospedeiro e os mecanismos de desenvolvimento da doença tendem a contribuir de forma crítica para um maior entendimento de sua patogênese, levando ao desenvolvimento de melhores estratégias de diagnóstico, tratamento e prevenção. Para avaliar o impacto de fatores genéticos em leishmaniose cutânea, neste estudo optou-se por analisar SNPs em IL-1b e FLI-1 associados à LC, provenientes da literatura, para replicação. A população estudada neste projeto foi composta por pacientes com LC, recrutados no ambulatório de Dermatologia da FMT-HVD por demanda espontânea, provenientes de áreas endêmicas para leishmaniose no Amazonas, com destaque para o ramal da Cooperativa e ramal do Pau-Rosa, com suas respectivas vicinais; e indivíduos sem a doença, provenientes das mesmas áreas endêmicas dos casos. De acordo com os achados no presente estudo, dos indivíduos acometidos com LC, 79% eram do sexo masculino e 20% do sexo feminino. A média de idade para os casos foi de 32 anos de idade. Nossos dados estão de acordo com os achados de Benicio et al., onde foram observados 78% de indivíduos do sexo masculino e 22% do sexo feminino, com média de idade de 29 anos (21). Segundo o Manual de Vigilância da Leishmaniose Tegumentar Americana, a doença atinge indivíduos do sexo masculino principalmente, em fase produtiva, o que caracteriza o acometimento de indivíduos cuja principal atividade ocupacional está 70 associada à penetração em áreas de mata, desflorestamento, agropecuária ou exercícios militares, tornando essa população altamente exposta à infecção por Leishmania sp (19). Para o grupo controle, procurou-se realizar emparelhamento por sexo e idade de acordo com o grupo caso, obtendo-se 59% de indivíduos do sexo masculino e 41% do sexo feminino, com idade média de 36 anos. Ter um grupo controle com idade média superior ao caso é encarado como fator positivo, pois dessa maneira tem-se uma maior segurança em trabalhar com um grupo que ultrapassou a idade média do grupo caso, sob as mesmas condições de risco, no entanto não desenvolveu a doença. Polimorfismos em FLI-1 foram descritos pela primeira vez em associação com L. major baseado em modelo murino (22). Sakthianandeswaren et al. validaram a importância do FLI-1 na resolução e cicatrização das lesões e identificaram três polimorfismos na região promotora deste gene, indicando-o como novo candidato influenciando na resistência à L. major. Nesse estudo, observou-se que em camundongos resistentes, o pico na expressão de FLI-1 em linfonodos na sexta semana de infecção correspondia ao observado no pico fenotípico no mesmo período, em que camundongos resistentes apresentaram estabilização e cicatrização da lesão. Em contraste, as lesões presentes em camundongos susceptíveis, seis semanais após infecção continuavam a crescer em tamanho e gravidade, possivelmente devido à ausência de uma expressão aumentada de FLI-1. Posteriormente, Castellucci et al., baseado no estudo citado anteriormente, examinaram FLI-1 como um gene controlando LC causada por L. braziliensis num estudo de associação baseado em família (17). No estudo de Castelluci et al., os SNPs de FLI-1 foram genotipados inicialmente em 168 famílias nucleares compostas por 250 casos de LC e 87 casos de LM, todos provenientes de Corte de Pedra, área de endemicidade para L. braziliensis, Bahia. Foi demonstrado associação do alelo C (P=0.003; OR=1.64) no SNP rs7930515 e risco para LC, que após análise primária, foi replicada numa segunda amostra de 402 casos de LC em 157 famílias nucleares (P=0.014;OR=1.60) coletadas na mesma região. No presente estudo, dentre os quatro SNPs genotipados para FLI-1, apenas verificou-se tendência para associação com LC no SNP rs7930515 (modelo aditivo p=0.075 e dominante p=0.036). Esta tendência à associação pode ser observada 71 entre o alelo C e susceptibilidade para leishmaniose cutânea pelo modelo de dominância [p=0.036 OR=2.38 (IC 95% 1.07 – 5.32)]. Os dados observados no presente estudo corroboram com os achados de Castellucci et al., em que o alelo C estava associado com risco para LC, no entanto nossos achados podem apenas ser considerados como sinal positivo de associação, uma vez que o valor de P apresentou-se “borderline”. Esses dados, somados a outros estudos evidenciam a importância do gene FLI1 como candidato influenciando resistência/susceptibilidade na leishmaniose, além de ter sido correlacionado com uma resposta de cura de lesão em modelo murino (17,22,23). Interleucina 1 (IL-1) pertence ao grupo de citocinas pro-inflamatórias do tipo Th1, e tem papel chave como mediador da resposta inflamatória com efeitos pleiotrópicos numa variedade de células e vias de sinalização, e tem função importante no desfecho da condição patológica (9). IL-1b é uma citocina com atividade pirogênica que é normalmente ativada em resposta a uma infecção ou injúria (24). Os inflamassomas tem papel um importante nesse contexto, devido sua função de promover a ativação de IL-1b através de vários estímulos durante a infecção por micro-organismos (25). Polimorfismos genéticos em IL-1b estão associados com susceptibilidade a diversas doenças autoimunes, inflamatórias e infecciosas, dentre elas tuberculose, gastrite crônica associada a Helicobacter pylori, HIV, esclerose múltipla, lupus eritematoso sistêmico, malária e leishmaniose (8,10–14). Existem dois polimorfismos bastante comuns no gene IL-1b influenciando a expressão da citocina: um localizado no exon 5 (IL-1b 3953 C/T) e outro localizado na região promotora (IL1-b -511 C/T) (26). Fernández-Figueroa et al. demonstraram que o polimorfismo em IL-1b (-511 C/T) está associado com um alto risco de contrair a doença em pacientes infectados com L. mexicana. Também foram capazes de demonstrar que pacientes com a forma mais grave da doença e que albergam um maior número de parasitas de L. mexicana, apresentam uma produção aumentada de IL-1b por monócitos in vitro, uma acentuada expressão sérica de IL-1b e uma distribuição difusa de IL-1b nas lesões (14). Estudo realizado para verificar associação do polimorfismo IL-1b -511 C/T com LV, demonstrou que o alelo C pareceu conferir susceptibilidade a LV, enquanto a 72 presença do alelo T atuou como possível fator influenciador para resistência à doença (26). Esses achados corroboram com os do presente estudo, em que se observou associação dos alelos C e T do polimorfismo IL-1b -511 C/T com susceptibilidade e resistência, respectivamente, à LC. Pelo modelo de dominância, pode-se sugerir a presença do alelo C conferindo risco [P = 0.027, OR=1.43 (IC 95% 1.04 – 1.94)] e o contrário, do alelo T conferindo resistência [P = 0.036, OR = 0.66 (IC 95% 0.45 – 0.96)]. Moravej et al., que também obtiveram o mesmo perfil de associação desse polimorfismo com LV, sugerem que indivíduos portadores do alelo IL-1b -511 C produzem mais IL-1b do que os portadores do alelo T, resultando no desenvolvimento da doença e resolução da lesão (26). Ainda, segundo dados de Fernández-Figueroa et al., a distribuição de IL-1b nos tecidos está associada à carga parasitária no hospedeiro, assim indivíduos com a forma mais grave da doença, e que, portanto possuíam carga parasitária maciça, apresentavam maiores níveis celulares de IL-1b. O oposto também foi observado (14). Associação de IL1b 3953 C/T foi observada conferindo risco para desenvolvimento da forma grave de malária em uma população de Gambia (12). Da mesma forma, associação desse polimorfismo foi observada com susceptibilidade para infecção por HIV (27). Mas não para leishmaniose visceral em uma população iraniana (26). Dados de Moravej et al. corroboram com os achados deste estudo, em que também não foi possível encontrar associação do polimorfismo em IL-1b 3953 C/T com LC (Modelo aditivo, P=1.15, X2=0.56). Conclusão Não foram observadas associações genéticas com a susceptibilidade/resistência à LC para os SNPs IL-1b_rs1143634, FLI1_rs619521 e FLI1_rs531894 na população estudada. Foram detectadas diferenças estatisticamente significativas na distribuição alélica e genotípica do polimorfismo em IL-1b -511 C/T (rs16944) entre os grupos caso e controle, sugerindo associação do alelo C com susceptibilidade, e do alelo T com resistência para LC. 73 O SNP rs7930515 de FLI-1 foi considerado como sinal positivo de associação, podendo-se sugerir que o alelo C estava associado com risco para LC. Os resultados obtidos no presente estudo reforçam o papel dos genes IL1-b e FLI-1 associados com resistência a LC. Referências 1. Stockdale L, Newton R. A review of preventative methods against human leishmaniasis infection. PLoS Negl Trop Dis [Internet]. 2013 Jan [cited 2014 Jul 14];7(6):e2278. Available from: http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=3688540&tool=pmcentrez&rendertyp e=abstract 2. Murray HW, Berman JD, Davies CR, Saravia NG. Advances in leishmaniasis. Lancet [Internet]. 2005;366(9496):1561–77. Available from: http://www.ncbi.nlm.nih.gov/pubmed/16257344 3. Sharma U, Singh S. Immunobiology of leishmaniasis. Indian J Exp Biol [Internet]. 2009 Jun;47(6):412–23. Available from: http://www.ncbi.nlm.nih.gov/pubmed/19634705 4. Desjeux P. Leishmaniasis: current situation and new perspectives. Comp Immunol Microbiol Infect Dis [Internet]. 2004 Sep [cited 2013 Feb 27];27(5):305–18. Available from: http://www.ncbi.nlm.nih.gov/pubmed/15225981 5. Guerra JA de O, Talhari S, Paes MG, Garrido M, Talhari JM. Aspectos clínicos e diagnósticos da leishmaniose tegumentar americana em militares simultaneamente expostos à infecção na Amazônia. Rev Soc Bras Med Trop [Internet]. 2003 Oct [cited 2013 Mar 11];36(5):587–90. Available from: http://www.scielo.br/scielo.php?script=sci_arttext&pid=S003786822003000500008&lng=pt&nrm=iso&tlng=pt 6. Romero G a, Vinitius De Farias Guerra M, Gomes Paes M, de Oliveira Macêdo V. Comparison of cutaneous leishmaniasis due to Leishmania (Viannia) braziliensis and L. (V.) guyanensis in Brazil: clinical findings and diagnostic approach. Clin Infect Dis [Internet]. 2001 May 1;32(9):1304–12. Available from: http://www.ncbi.nlm.nih.gov/pubmed/11303265 7. Ajdary S, Ghamilouie M-M, Alimohammadian M-H, Riazi-Rad F, Pakzad S-R. Toll-like receptor 4 polymorphisms predispose to cutaneous leishmaniasis. Microbes Infect [Internet]. Elsevier Masson SAS; 2011 Mar [cited 2012 Sep 8];13(3):226–31. Available from: http://www.ncbi.nlm.nih.gov/pubmed/21056683 8. Hollegaard M V, Bidwell JL. Cytokine gene polymorphism in human disease: on-line databases, Supplement 3. Genes Immun [Internet]. 2006 Jun [cited 2014 Jun 18];7(4):269–76. Available from: http://www.ncbi.nlm.nih.gov/pubmed/16642032 9. Ahir S, Chaudhari D, Chavan V, Samant-Mavani P, Nanavati R, Mehta P, et al. Polymorphisms in IL-1 gene cluster and its association with the risk of perinatal HIV transmission, in an Indian cohort. Immunol Lett [Internet]. Elsevier B.V.; 2013 Jun [cited 2014 Jun 18];153(1-2):1–8. Available from: http://www.ncbi.nlm.nih.gov/pubmed/23769826 10. Haukim N, Bidwell JL, Smith a JP, Keen LJ, Gallagher G, Kimberly R, et al. Cytokine gene polymorphism in human disease: on-line databases, supplement 2. Genes Immun [Internet]. 2002 Sep [cited 2014 Jun 18];3(6):313–30. Available from: http://www.ncbi.nlm.nih.gov/pubmed/12209358 74 11. Silva MR, Sampaio A, Almeida A, Balseiro S, Santos P, Carvalho L. IDENTIFICAÇÃO DOS POLIMORFISMOS DOS GENES IL1B , IL1RN E TNFA NA GAS- TRITE CRÓNICA ASSOCIADA À INFECÇÃO POR HELICOBACTER PYLORI E NO. J Port Gastrenterol. 2008;15(1):8–14. 12. Walley AJ, Aucan C, Kwiatkowski D, Hill AVS. Interleukin-1 gene cluster polymorphisms and susceptibility to clinical malaria in a Gambian case-control study. Eur J Hum Genet [Internet]. 2004 Feb [cited 2014 Jun 19];12(2):132–8. Available from: http://www.ncbi.nlm.nih.gov/pubmed/14673470 13. Azad AK, Sadee W, Schlesinger LS. Innate immune gene polymorphisms in tuberculosis. Infect Immun [Internet]. 2012 Oct [cited 2014 May 28];80(10):3343–59. Available from: http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=3457569&tool=pmcentrez&rendertyp e=abstract 14. Fernández-Figueroa E a, Rangel-Escareño C, Espinosa-Mateos V, Carrillo-Sánchez K, SalaizaSuazo N, Carrada-Figueroa G, et al. Disease severity in patients infected with Leishmania mexicana relates to IL-1β. PLoS Negl Trop Dis [Internet]. 2012 Jan [cited 2013 Mar 11];6(5):e1533. Available from: http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=3358333&tool=pmcentrez&rendertyp e=abstract 15. Oikawa T, Yamada T. Molecular biology of the Ets family of transcription factors. Gene [Internet]. 2003 Jan 16;303:11–34. Available from: http://www.ncbi.nlm.nih.gov/pubmed/12559563 16. Asano Y, Markiewicz M, Kubo M, Szalai G, Watson DK, Trojanowska M. Transcription factor Fli1 regulates collagen fibrillogenesis in mouse skin. Mol Cell Biol [Internet]. 2009 Jan [cited 2013 Apr 5];29(2):425–34. Available from: http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=2612518&tool=pmcentrez&rendertyp e=abstract 17. Castellucci L, Jamieson SE, Miller EN, de Almeida LF, Oliveira J, Magalhães a, et al. FLI1 polymorphism affects susceptibility to cutaneous leishmaniasis in Brazil. Genes Immun [Internet]. 2011 Oct [cited 2012 Oct 11];12(7):589–94. Available from: http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=3297968&tool=pmcentrez&rendertyp e=abstract 18. Sambrook, J.; Fritsch, EF.; Maniatis, T. Molecular Cloning: A Laboratory Manual. Cold Spring Harbor Laboratory Press; Cold Spring Harbor, New York: 1989. 19. Ministério da Saúde. Secretaria de Vigilância em Saúde. Departamento de Vigilância Epidemiológica. Manual de Vigilância da Leishmaniose Tegumentar Americana [Internet]. 2 a ed. Brasília: Ministério da Saúde; 2010. Available from: http://bvsms.saude.gov.br/bvs/publicacoes/manual_vigilancia_leishmaniose_tegumentar_ameri cana.pdf 20. Quilez J, Martínez V, Woolliams J a, Sanchez A, Pong-Wong R, Kennedy LJ, et al. Genetic control of canine leishmaniasis: genome-wide association study and genomic selection analysis. PLoS One [Internet]. 2012 Jan [cited 2012 Sep 8];7(4):e35349. Available from: http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=3338836&tool=pmcentrez&rendertyp e=abstract 21. Benicio E de A, Gadelha EPN, Talhari A, Silva RM da, Ferreira LC, Santos MCC dos, et al. Combining diagnostic procedures for the management of leishmaniasis in areas with high prevalence of Leishmania guyanensis. An Bras Dermatol [Internet]. 2011 [cited 2014 Sep 26];86(6):1141–4. Available from: http://www.ncbi.nlm.nih.gov/pubmed/22281902 75 22. Sakthianandeswaren A, Curtis JM, Elso C, Kumar B, Baldwin TM, Lopaticki S, et al. Fine mapping of Leishmania major susceptibility Locus lmr2 and evidence of a role for Fli1 in disease and wound healing. Infect Immun [Internet]. 2010 Jun [cited 2014 Sep 20];78(6):2734–44. Available from: http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=2876540&tool=pmcentrez&rendertyp e=abstract 23. Castellucci LC, Almeida LF De, Jamieson SE, Fakiola M, Carvalho EM De, Blackwell JM. Host genetic factors in American cutaneous leishmaniasis: a critical appraisal of studies conducted in an endemic area of Brazil. Mem Inst Oswaldo Cruz [Internet]. 2014 May 27 [cited 2014 Sep 20];00(ahead):00–00. Available from: http://www.scielo.br/scielo.php?script=sci_arttext&pid=S007402762014005040028&lng=en&nrm=iso&tlng=en 24. Dinarello CA. Interleukin-1 beta, interleukin-18, and the interleukin-1 beta converting enzyme. Ann N Y Acad Sci [Internet]. 1998 Sep 29 [cited 2013 Mar 11];856:1–11. Available from: http://www.ncbi.nlm.nih.gov/pubmed/9917859 25. Weber A, Wasiliew P, Kracht M. Interleukin-1beta (IL-1beta) processing pathway. Sci Signal [Internet]. 2010 Jan [cited 2013 Mar 11];3(105). Available from: http://www.ncbi.nlm.nih.gov/pubmed/20086236 26. Moravej A, Rasouli M, Kalani M, Asaei S, Kiany S, Najafipour S, et al. IL-1β (-511T/C) gene polymorphism not IL-1β (+3953T/C) and LT-α (+252A/G) gene variants confers susceptibility to visceral leishmaniasis. Mol Biol Rep [Internet]. 2012 Jun [cited 2014 Sep 24];39(6):6907–14. Available from: http://www.ncbi.nlm.nih.gov/pubmed/22311026 27. Pontillo A, Oshiro TM, Girardelli M, Kamada AJ, Crovella S, Duarte AJS. Polymorphisms in inflammasome’ genes and susceptibility to HIV-1 infection. J Acquir Immune Defic Syndr [Internet]. 2012 Feb 1;59(2):121–5. Available from: http://www.ncbi.nlm.nih.gov/pubmed/22227487
Download