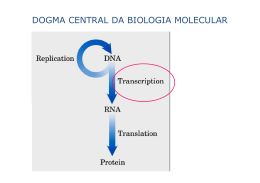
Processamento pós-transcricional Capeamento Remoção de introns Adição da cauda de poliA Ligação a proteínas ligantes ao RNA Capeamento Remoção de introns Adição da cauda de poliA Varios tipos de capeamento: extensão de reações de metilação Capeamento: ocorre no primeiro minuto Todos os mRNAs eucariotos são capeados Proteção contra degradação: decapeamento ligado a degradação Ligação de fatores que facilitarão ligação do ribossomo Disjunção: splicing Introns Definidos por sequências GT (5’) e AG (3’) Presença de códons de terminação em ordem de leitura (ORF) Mecanismo da disjunção: complexa interação entre proteínas Splicing alternativo Poliadenilação - Presente em eucariotos: enzima Poli-A polimerase Sequência AAUAAA em mamíferos: Sinal de poliadenilação e de terminação da transcrição - Altamente conservada em mamíferos Função: transporte, estabilidade, eficiência da tradução - Presente em região 11 a 30 nt antes do sítio de adição do poliA - cordycepin (3’deoxyadenosine): impede o transporte para o citosol Em animais somente uma sequência é presente. Em plantas, várias: diferentes tamanhos para um mRNA: diferente estabilidade e tradução Sinal de poliadenilação e de terminação da transcrição Tamanho da cauda de poli-A pode variar de gene para gene poli-A: estabilidade e/ou eficiência de tradução Instabilidade do RNA em plantas Experimentos com actinomicina D (inibidor da transcrição): presença desse Elemnto reduz a vida do RNA Instabilidade do RNA em plantas Erro de leitura da RNA polimerase II. Ex: gene da fitohemaglutinina Região responsável pelo aumento da estabilidade do RNA Domínios II e III,aricos em (AU)n instabilidade em I,resposta presença de luzresponsáveis no mRNA dapela ferredoxina na presença de açúcares Instabilidade e degradação do RNA Proteínas ligantes internamente ao RNA Elemento IRE Degradação do RNA em levedura Degradação do RNA em plantas Degradação do RNA em plantas
Baixar