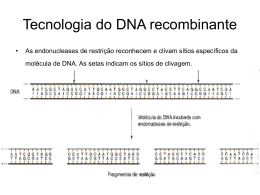
Área de Ciências da Saúde – Curso de Medicina Módulo: Saúde do Adulto e Idoso II – GENÉTICA HUMANA Professora: Dra. Juliana Schmidt SEPARAÇÃO ELETROFORÉTICA DE DNA A eletroforese em gel de agarose consiste no método mais usado para separar, identificar, analisar, caracterizar e purificar fragmentos de DNA. Uma molécula de DNA, quando exposta a um campo elétrico, migra para o eletrodo na velocidade ou mobilidade eletroforética, proporcional a força do campo e a carga líquida da molécula. A mobilidade eletroforética é também inversamente proporcional ao coeficiente friccional da molécula, que, por sua vez, é função do tamanho e forma da molécula, e da viscosidade do meio. A eletroforese separa fragmentos de DNA de acordo com seus tamanhos relativos. Fragmentos de DNA são carregados em um gel agarose, que é colocado em uma câmara preenchida com um solução tampão condutora. A corrente contínua é passada entre os eletrodos de arame em cada extremidade da câmara. Fragmentos de DNA são carregadas negativamente, e quando colocado em um elétrico campo serão atraídos para o pólo positivo e repelidos pelo pólo negativo. A matriz do gel agarose age como uma peneira molecular através da qual pequenos fragmentos de DNA podem se mover mais facilmente que os maiores. Durante um determinado período de tempo, fragmentos menores vão viajar mais longe do que maiores. Fragmentos do mesmo tamanho vão ficar juntos e migrar como uma "banda" de DNA no gel. No exemplo abaixo (Figura 13), é mostrado um gel de eletroforese com bandas de amplificados de PCR, com tamanhos de 941 bp e 641 bp, separadas com base em seus tamanhos. O genoma humano contém pequenos elementos de DNA repetitivo que tornaram-se aleatoriamente inseridos nele ao longo de milhões de anos. Um desses elementos repetitivos é chamado de "seqüência Alu" . Esta é uma seqüência de DNA de cerca de 300 pares de bases que se repete quase 500 mil vezes ao longo do genome. Algumas dessas seqüências Alu têm características que as tornam muito úteis para geneticistas. Quando presentes dentro de íntrons dos genes certos, eles podem ser associados com uma doença ou ser usado para estimar parentesco entre os indivíduos. Nesta atividade prática a análise de uma repetição Alu única é usado para estimar sua freqüência na população e como uma simples medida da variação genética molecular - sem nenhuma referência à doença ou parentesco entre os indivíduos. Você irá analisar um elemento Alu na região da PV92 cromossomo 16, o qual foi amplificado na aula anterior de PCR. Este elemento Alu particular é dimórfico, o que significa que o elemento é presentes em alguns indivíduos e outros não. Algumas pessoas têm em uma cópia do cromossomo 16 (um alelo), outros podem ter a inserção em ambas as cópias do cromossomo 16 (dois alelos), enquanto alguns podem não ter a inserção em nenhuma cópia do cromossomo (Figura 12). Os primers neste kit são projetados para amplificar uma seqüência dentro da região PV92 com 641 pares de base se o intron não contém a inserção Alu, ou 941 pares de base Alu se está presente. Este aumento no tamanho é devido à seqüência de 300 pares de bases da inserção Alu. Quando seus produtos de PCR são analisados por eletroforese em gel de agarose, três distintos resultados são possíveis. Se ambos os cromossomos contêm inserções Alu, cada produto amplificado de PCR produto terá 941 pares de base, eles vão migrar como uma banda que corresponde a 941 pares de bases. Se nenhum cromossomo contém o elemento Alu, cada produto amplificado de PCR terá 641 pares de base e eles vão migrar como uma banda que corresponde a 641 pares de bases. Se houver uma inserção Alu em um cromossomo, mas não no outro, haverá um produto de PCR de 641 pares de base e um de 941 pares de bases. O gel vai revelar duas bandas para tal amostra. Em humanos, existem centenas de segmentos de DNA como o Alu, que podem ser selecionados e utilizados para análise de impressão digital de DNA. Dependendo de fatores demográficos, como etnia e isolamento geográfico, algumas populações mostram variação muito menor em segmentos de DNA do que outros. Um menor grau de variação aumenta as chances de mais de um indivíduo ter a mesma seqüência. Se 33% (1 de três indivíduos) de uma dada população tem o mesmo padrão de impressões digitais para um certo segmento de DNA, identificar um indivíduo. então pouca Nesse informação caso, um será resultado obtida positivo utilizando só esse identificará segmento uma sozinho pessoa com para 33% precisão. Ao analisar as provas incriminatórias baseadas em testes de DNA é preciso fazer a pergunta: Estatisticamente, quantas pessoas em uma população terão o mesmo padrão de DNA que as tomadas de uma cena de crime: 1 em 1.000.000? 1 em 10.000? 1 em 10? Para uma impressão digital de DNA poder identificar de modo preciso um suspeito em um caso criminal ou um pai em uma identificação de paternidade são necessárias não uma chance de 1 de 3 (1 / 3) em uma população, mas mais perto de um 1 em 10 milhões de chances (1 / 107). A freqüência de um padrão de DNA particular em uma população diminui drasticamente quando segmentos múltiplos de DNA são selecionados e amplificados, ao invés de apenas um segmento. Para identificação de DNA ser admissível como prova em tribunal, deve-se analisar 30-40 segmentos diferentes de DNA em cromossomos múltiplos da mesma pessoa. A inserção Alu usada neste exercício tem sido utilizada para estudar padrões de migração de populações humanas ao longo do tempo. Teoricamente, em populações geograficamente isoladas, todos os indivíduos podem ser homozigotos + / +. Em outros, os indivíduos podem ser todos homozigotos - / -. Em população não isoladas, os três genótipos (+/+, + / -, - / -) podem existir em equilíbrio. As freqüências dos genótipos e alelos são características básicas que geneticistas usam para descrever e analisar populações. Os resultados obtidos neste exercício oferecem uma oportunidade real para calcular as freqüências genotípicas e alélicas da inserção Alu em sua turma e usar a equação de Hardy-Weinberg. A equação de HardyWeinberg, p2 + q2 + 2pq = 1, é um dos fundamentos da genética de populações. É a expansão algébrica de (p + q) 2 = 1, onde p + q = 1. A equação descreve as freqüências de genótipos em uma população que está em "equilíbrio genético", o que significa que as freqüências são estáveis de geração em geração. A teoria de Hardy-Weinberg afirma que para grande, os membros uma população devem acasalar alcançar e este produzir equilíbrio, descendentes a população aleatoriamente deve com ser igual bastante sucesso, e não deve haver a migração de pessoas para dentro ou fora da população, ou uma excessiva frequencia de mutações convertendo um alelo para outro. Dadas essas condições, e as freqüências alélicas p e q, a equação de Hardy-Weinberg diz que: p2 = freqüência esperada do genótipo (+/+) na população 2pq = freqüência esperada do genótipo (+/-) na população q2 = a freqüência esperada do genótipo (-/-) na população É importante compreender que p2, 2pq e q2 são as frequencias teóricas dos genótipos esperados de uma população e elas não podem ser calculadas em amostras de uma população, se uma das condições não for atendida. Essas frequencias teóricas são calculadas usando os valores observados para p e q, e eles podem ou não ser o mesmo que as freqüências genotípicas observadas na população real. Se as frequencias genotípicas observadas e as esperadas são as mesmas, isso indica que a população está em equilíbrio genético de acordo com Hardy-Weinberg. Área de Ciências da Saúde – Curso de Medicina Módulo: Saúde do Adulto e Idoso II – GENÉTICA HUMANA Professora: Dra. Juliana Schmidt SEPARAÇÃO ELETROFORÉTICA DE DNA OBJETIVO Promover a separação de fragmentos de DNA produzidos nas aulas anteriores MATERIAIS Fragmentos de DNA obtidos anteriormente (PCR) - micropipetas de 20µL - Tampão de carregamento de amostra - ponteiras de 200µL - Tampão de eletroforese TAE 1X - cuba de eletroforese e fonte ddp - gel de Agarose 1% previamente preparado - Isopor com gelo picado - solução Fast Stain diluida 100X (~120mL) - Câmara UV - EZ load molecular mass (1000, 700, 500, 200, 100bp) - Solução de brometo de etídio - Transparências e papel A4 branco - PROCEDIMENTO 1. Preparar um gel de agarose a 1% em tampão TAE 1X; 2. Montar a forma para receber o gel de agarose; 3. Despejar o gel na forma e aguardar sua solidificação; 4. Colocar o tampão TAE 1X na cuba de eletroforese e retirar o pente do gel; 5. Misturar as amostras de DNA com tampão de carregamento e aplicar nos poços; 6. Ligar os eletrodos da cuba a uma fonte geradora de diferença de potencial e ajustar a voltagem para 100V; 7. Acompanhar o deslocamento dos corantes indicadores ao longo do gel durante a corrida; 8. Desligar a fonte quando o primeiro corante tiver se deslocado por 2/3 do comprimento do gel (aprox. 30 min); 9. Retirar o gel da forma e coloca-lo em um recipiente para proceder à coloração; 10. Corar o gel com FastStain por 2 minutos e depois descorar com água aquecida (40-50°C) por 10 minutos com agitação. Pode-se corar o gel com solução de brometo de etídio por aproximadamente 20 minutos, depois retirar o excesso de corante lavando o gel em água deionizada. (CUIDADO!!! O BROMETO DE ETÍDIO É MUTAGÊNICO – USAR LUVAS PARA MANIPULAR O GEL) ; 11. Visualizar o gel corado na câmara de luz UV acoplada ao computador. Obs: se o gel for corado com FastStain, não pode ser corado posteriormente com brometo de etídeo. O contrário pode ser feito. Área de Ciências da Saúde – Curso de Medicina Módulo: Saúde do Adulto e Idoso II – GENÉTICA HUMANA Professora: Dra. Juliana Schmidt SEPARAÇÃO ELETROFORÉTICA DE DNA Relatório de aula prática – Data:____/____/____ - Turma:____ Nota: Nome:_____________________________________________________________________________ Nome:_____________________________________________________________________________ Nome:_____________________________________________________________________________ Nome:_____________________________________________________________________________ Nome:_____________________________________________________________________________ QUESTÃO 1) Analise o resultado da eletroforese comparando sua amostra com as faixas de controle, utilizando o marcador de tamanho DNA como uma referência. Ao comparar o seu padrão de migração de DNA com os controles, determine se são homozigotos + / +, homozigotos - / -, ou heterozigoto + / -. Lembre-se que a interpretação deste gel permite a você determinar sua composição genética apenas no locus que está sendo estudado. Faça um esboço explicativo do resultado obtido (visualização do gel corado), explicando por que os dois produtos possíveis PCR diferem em tamanho por 300 pares de base e como isso pode ser usado para determinar o genótipo homo ou heterozigoto.
Download