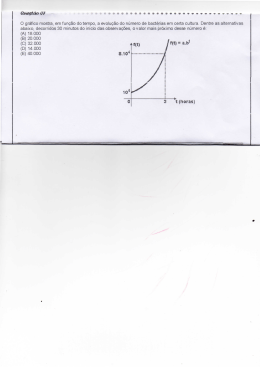
Detecção de resistência antimicrobiana em bactérias isoladas de animais será tema de palestra O uso indiscriminado de antimicrobianos na produção animal tem favorecido a disseminação da resistência antimicrobiana entre humanos, um problema com sérias consequências para a saúde pública. Por essa razão, é grande o interesse no desenvolvimento de métodos que permitam detectar a resistência antimicrobiana em bactérias isoladas de animais. A professora Miliane Moreira Soares de Souza, da Universidade Federal Rural do Rio de Janeiro (UFRRJ), falará sobre esse assunto durante uma palestra no XXVIII Congresso Brasileiro de Microbiologia, que será realizado em Florianópolis entre 18 e 22 de outubro. Nesta entrevista à Micro in foco, a professora falou sobre os desafios metodológicos à identificação da resistência antimicrobiana em micro-organismos oriundos do gado e das aves de granja. Um levantamento recente de pesquisadores da Universidade de Princeton detectou que 80% dos antibióticos consumidos a cada ano nos Estados Unidos são empregados na pecuária. Quais os perigos dessa prática? A utilização de antimicrobianos como promotores de crescimento em produção animal tem o objetivo de controlar doenças infecciosas que poderiam comprometer a conversão alimentar e o índice de produtividade. Esta prática acarreta a concentração de resíduos desses fármacos no ambiente, na carne e derivados desses animais, como o leite, levando a um aumento da pressão de seleção positiva o que contribui para a expressão da denominada resistência antimicrobiana ativa, e consequente dispersão de genes de resistência. Por esse motivo, a União Europeia promove embargo a produtos que apresentem resíduos de determinados antimicrobianos. Quais as alternativas para tornar a criação de gado e as granjas menos dependentes do uso de antibióticos? Essa é uma pergunta com inúmeros vieses, porque depende do tipo de produção e da realidade local. Mas, de modo geral, é preciso atuar de modo permanente na capacitação profissional das pessoas diretamente envolvidas no manejo dos animais, para que a adoção de boas práticas seja inerente ao trabalho e não apenas um conjunto de ações registradas, mas que não são observadas no cotidiano da produção. Além disso, envolve aspectos como qualidade da água, limpeza do ambiente do entorno, controle de dípteros e outros possíveis agentes carreadores de agentes patogênicos, entre outros. O que as pesquisas mais recentes têm revelado sobre a disseminação da resistência antimicrobiana nos diferentes ambientes de produção animal? Em linhas gerais, o que os estudos apontam é que, em decorrência dos diferentes tipos de criação e modelos de produção, da circulação de espécies que se inter-relacionam, dos distintos agravos e desafios enfrentados, esses ambientes promovem intensa circulação de agentes bacterianos e, consequentemente, elevada circulação de genes de resistência. Ao agregar a esse conjunto o uso indiscriminado de antimicrobianos, gerando aumento da pressão de seleção no meio, estabelece-se um ambiente propício à recombinação gênica e à propagação dessas informações de resistência. Exemplos desse modelo de propagação foram o surgimento das cepas VRE em ambiente de suinocultura e das cepas MARSA-LA. Quais são os principais desafios envolvidos na detecção de resistência antimicrobiana em bactérias isoladas de animais? Em primeiro lugar, é necessária a identificação precisa das espécies. Se consideramos um gênero como Staphylococcus spp, extremamente abundante no cotidiano da produção animal, temos um maior número de espécies coagulase-positivas e perfis de resistência distintos – diferentemente do que ocorre na medicina humana. Outro aspecto que foi observado é que alguns testes baseados em resistência que funcionam para diferenciação de bactérias provenientes de humanos não necessariamente serão eficientes para aquelqs provenientes de animais. É o caso, por exemplo, da resistência à bacitracina, de baixo uso no ambiente de produção e mesmo na clínica veterinária de animais de companhia. Quanto à detecção gênica de resistência, um estudo reporta que mudanças pontuais na região de anelamento do gene mecA em bactérias oriundas de hospedeiros bovinos impediam o anelamento do iniciador desenvolvido com base na seqüência gênica de bactéria de hospedeiro humano, gerando resultado falso-negativo. Esse gene é correlacionado com a predição de resistência a beta-lactâmicos. Atualmente, trabalhamos na construção de um iniciador universal, para sua ampliação. Além disso, foram descobertos genes homólogos ao mecA em animais. Essas observações revelam que a transposição direta de metodologias da microbiologia clínica humana não permite a acurácia e a especificidade necessárias.
Baixar