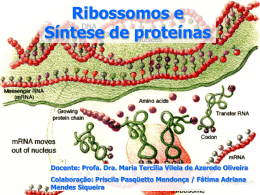
Programa de Pós-Graduação em Genética RIBOSSOMOS E SÍNTESE PROTÉICA Doutoranda: Leiza V. Penariol RIBOSSOMOS • 20-30 nm de diâmetro • observados em microscopia eletrônica • E. coli 20.000 ribossomos (25% peso seco) • céls. mamíferos em proliferação: 10 milhões ribossomos ____________________________________________________Ribossomos Composição e estrutura • constituídos por moléculas de RNAr e proteínas • formado por duas subunidades: maior e menor • subunidade menor se liga ao RNAm e ao RNAt durante a síntese de proteínas • a subunidade maior catalisa a ligação peptídica entre os aminoácidos durante a síntese de proteínas ____________________________________________________Ribossomos ____________________________________________________Ribossomos Procariotos x Eucariotos ____________________________________________________Ribossomos Localização • livres no citoplasma • associados à membrana do retículo endoplasmático rugoso • associados à membrana externa do envoltório nuclear • no interior das mitocôndrias e dos cloroplastos ____________________________________________________Ribossomos Polissomos ou Polirribossomos Formações constituídas por uma molécula de RNA mensageiro + vários ribossomos Polissomos podem estar: a) Livres no citoplasma b) Aderidos à membrana ____________________________________________________Ribossomos a) Polissomos livres no citoplasma • sintetizam proteínas destinadas à célula • ocorrem em células que sintetizam grande quantidade de proteínas usadas internamente no crescimento rápido • ex: células reticulócitos cancerosas, células embrionárias e (precursores das hemáceas, polissomos sintetizam hemoglobina) ____________________________________________________Ribossomos a) Polissomos aderidos à membrana • sintetizam proteínas que serão secretadas Ex: glândula mamária: em repouso → polissomos livres em lactação → ligados a memb. do retículo • sintetizam proteínas que compõem estruturas celulares membranosas Ex: memb. plasmática, Golgi, lisossomos, vacúolos Síntese de proteínas a transcrição e a tradução são os meios pelos quais as células expressam suas instruções genéticas (seus genes) Gene A Os genes podem ser Gene B Transcrição expressos com RNA diferentes RNA Tradução eficiências A A A A A A A A A A A A A A A A B As informações nas células DNA Gene: sequência linear nucleotídeos NÚCLEO RNA RNAm: sequência códons CITOPLASMA PROTEÍNAS TRANSCRIÇÃO TRADUÇÃO Formação da cadeia polipeptídica ________________________________________________________Síntese protéica DNA • todas as células armazenam a informação hereditária em moléculas de DNA • dupla hélice, formada nucleotídeos • nucleotídeo: açúcar (desoxirribose) + gr. fosfato + base nitrogenadas (A,T, C, G) • produz cópias de si mesmo durante a divisão celular • capaz de expressar sua informação, usando-a para guiar a síntese de outras moléculas na célula ________________________________________________________Síntese protéica RNA • fita simples (hélice) • açúcar ≠ DNA: ribose • bases: A, C, G e U • cadeia pode dobrar-se • conformação tridimensional permite executar diversas funções ________________________________________________________Síntese protéica Tipos de RNA • RNAm: codifica proteínas (transcrito DNA) • Outros RNAs não mensageiros (produto final do gene) - componentes estruturais e enzimáticos das céls - traduzem a mensagem genética para a proteína RNAr: forma os ribossomos, síntese protéica RNAt: adaptadores entre RNAm e aa ________________________________________________________Síntese protéica RNA mensageiro (RNAm) • transcrito complementar a uma das fitas do DNA (fita molde) • especifica a sequência de aminoácidos (aa) __________________________________________________________________Síntese protéica Transcrição • copia do gene (DNA) em uma seq. nucleotídeos do RNA • informação escrita na mesma linguagem: nucleotídeos • abertura e desenrolamento pqna região DNA • formação da cadeia de RNA: transcrito (5’ 3’) • liberação fita RNA (processo rápido) • enzima atuante: RNA polimerase __________________________________________________________Síntese protéica RNA polimerase • desenrola a hélice de DNA • catalisa as ligações que unem os nucleotídeos • adiciona nucleotídeos, a cd 3 bases (trinca): códon • não possui atividade de revisão e correção nucleotídica • 1 erro a cd 104 nucleotídeos (107 DNA polimerase) __________________________________________________________Síntese protéica Transcrito primário: molécula de RNAm • processado antes de sair do núcleo • íntros são removidos • restam apenas os éxons • RNAm maduro contém apenas as sequências que codificam proteínas • migra para citoplasma (através do complexo de poros), onde vai ocorrer a tradução Transcrição: Vídeo ________________________________________________________Síntese protéica RNA transportador (RNAt) • formado por uma pqna cadeia de nucleotídeos dobrada sobre si mesma (forma de trevo) • produzido no núcleo e migra p/ citoplasma • adaptador entre aa e RNAm • captura aa e o transporta até o RNAm no ribossomo • primeira base, G modificada em inosina (I), “oscilante”: pareia com C, A e U • por isso pouco mais de 30 tipos RNAt e não 61 ________________________________________________________Síntese protéica Duas regiões importantes no RNAt Ligação com aa Ligação com RNAm RNA transportador: Braço TC Braço do aminoácido Braço DHU (resíduos 10-25) Braço do anticódon Anticódon ________________________________________________________Síntese protéica RNA ribossômico (RNAr) + proteínas Ribossomos • fornecem o meio para controlar a interação RNAm e RNAt • grande complexo formado por mais de 50 proteínas • movem-se ao longo da cadeia do RNAm • em células eucarióticas: 1 ribossomo adiciona 2 aa/segundo • em células procarióticas: 20 aa/segundo ________________________________________________________Síntese protéica 4 sítios de ligação nos ribossomos • um para se ligar ao RNAm • três para se ligar ao RNAt (A, P, E) ________________________________________________________Síntese protéica Proteínas • dirigem › dos processos químicos das células (enzimas) • outras funções: estruturais, movimentação, sensorial, etc • colocam em AÇÃO a informação genética da célula • polímeros constituídos por 20 tipos ≠ aminoácidos (aa) • cada tipo aa codificado por 3 nucleotídeos (trincas), chamadas de CÓDONS • 64 códons possíveis: 3 stop codons, 61 codificam aa (desses, 1 tb inicia síntese) • › aa são codificados por mais de 1 códon ________________________________________________________Síntese protéica Ligação entre os aa Os aminoácidos são unidos por ligações covalentes ligações peptídicas ________________________________________________________Síntese protéica Somente 20 aminoácidos dos conhecidos ocorrem em proteínas ________________________________________________________Síntese protéica Os aa, os seus códigos e códons correspondentes Proteína: codificada pela seqüência de códons (trincas de bases nitrogenadas presentes na molécula de RNAm) Códon de início: AUG (MET) Códon de terminação: UAA, UAG e UGA (STOP) ________________________________________________________Síntese protéica Características do Código Genético 1. LINEAR: bases ribonucleotídeos do RNAm LETRAS 2. TRIPLO: Códon 3 nucleotídeos: 1 aminoácido 3. NÃO AMBÍGUO: cd trinca nucleot. especifica apenas um aa 4. DEGENERADO: um aa pode ser especificado por + 1 trinca 5. SINAIS DE INICIAÇÃO E TERMINAÇÃO 6. SEM INTERRUPÇÕES 7. NÃO SOBREPOSTO 8. UNIVERSAL As informações nas células DNA Gene: sequência linear nucleotídeos NÚCLEO RNA RNAm: sequência códons CITOPLASMA PROTEÍNAS TRANSCRIÇÃO TRADUÇÃO Formação da cadeia polipeptídica _________________________________________________________Síntese protéica Tradução • processo complexo • tradução da informação genética a partir do alfabeto de 4 letras dos nucleotídeos (C, G, U, A) do RNAm em letras das proteínas (aa) _________________________________________________________Síntese protéica Maquinaria da tradução Ocorre no citoplasma • RNAm • RNAt • ribossomos • enzimas • cofatores _________________________________________________________Síntese protéica Etapas da tradução Ativação dos aa INICIAÇÃO ELONGAÇÃO TERMINAÇÃO Processamento pós-tradução _________________________________________________________Síntese protéica Ativação dos aminoácidos • ligação do aa ao seu RNAt • aminoacil-tRNA sintetase: ativação dos aa e ligação aa-RNAt Reação: Mg2+ aa + RNAt + ATP aminoacil-tRNA + AMP +2 Pi _________________________________________________________Síntese protéica Ligação do aa ativado ao RNAt _________________________________________________________Síntese protéica Ativação do aa Para cada aminoácido uma enzima diferente para realizar a ativação deste com o seu RNAt _________________________________________________________Síntese protéica Ativação do aa 1. Enzima reconhece o aa (lisina) e seu RNAt com o anticódon UUU _________________________________________________________Síntese protéica Ativação do aa 2. A energia para a ligação do aa ao RNAt provém da hidrólise do aminoacilAMP _________________________________________________________Síntese protéica Ativação do aa 3. O aminoacilRNAt é liberado 4. Enzima disponível p/ ativar outro aa _________________________________________________________Síntese protéica Pareamento do complexo aa – RNAt (aminoacil-RNAt) ao códon do RNAm A base presente na primeira posição (3’ 5’) do anticódon é oscilante e pode emparelhar-se com mais de um tipo de base na terceira posição do códon (5’ 3’) _________________________________________________________Síntese protéica Complexo de iniciação: componentes 1. ribossomos: percorrem a molécula de RNAm e promovem a união dos aa transportados pelos RNAt 2. RNAm 3. fMet-tRNAfMet:: (Met) 1º aa de qualquer cadeia polipeptídica 4. fatores de iniciação: IF-1, IF-2 e IF-3 5. GTP _________________________________________________________Síntese protéica Iniciação a) Acoplamento do RNAm à subunidade menor do ribossomo b) União do 1º RNAt (sítio P) ao códon de início da proteína (AUG) c) Junção das duas subunidades do ribossomo _________________________________________________________Síntese protéica Elongação: Microciclo 1. Complexo de Iniciação 2. Aminoacil-tRNA 3. Fatores de Elongação: EF-TU, EF-TS e EF-G 4. GTP Reações desde a síntese da 1º degradação peptídica até a última _________________________________________________________Síntese protéica Elongação a) entrada do 2º aminoacil-RNAt no sítio A do ribossomo (livre) b) Formação da ligação peptídica c) Movimentação do ribossomo ao longo do RNAm d) Sítio A vazio: nova seqüência de procedimentos 3 a 5 aa/s Proteína com 100 a 200 aa menos 1minuto _________________________________________________________Síntese protéica Terminação: Componentes: 1. Um dos códons de terminação: UAA, UAG, UGA 2. Fatores de Terminação (liberação): RF1, RF2 e RF3 3. GTP _________________________________________________________Síntese protéica Terminação a) Prosseguimento da síntese é interrompido quando o ribossomo alcança um dos códons de terminação: UAA, UAG, UGA b) Fatores de liberação reconhecem códons de terminação c) Peptidil do sítio A passa para o sitio P d) Liberação - Subunidades > e < - RNAt - RNAm Diferenças na síntese de Procariotos e Eucariotos Característica Ribossomos Aminoácido iniciador Códon de iniciação Identificação do códon Procariotos Eucariotos 70S 80S n-formilmetionina AUG GUG Shine-Dalgarno de iniciação Fatores de iniciação metionina AUG Ribossomo + Cap 1º AUG IF-1, IF-2 e IF-3 eIF-2, eIF-3, eIF-4c, CBPI, eIF-4ª, eIF4B, eIF-4F, eIF-5, eIF-6 mRNA Policistrônico Monocistrônico Fatores de elongação EF-TU, EF-TS e EF-1 α, EF-1 β e Fatores de terminação EF-G EF-2 RF-1, RF-2 e RFe RF-3 _________________________________________________________Síntese protéica Sequência da síntese protéica _________________________________________________________Síntese protéica _________________________________________________________Síntese protéica _________________________________________________________Síntese protéica _________________________________________________________Síntese protéica _________________________________________________________Síntese protéica Obrigada!!! Dúvidas???
Baixar