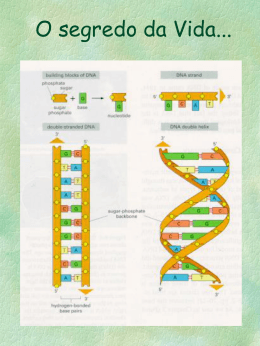
Estrutura dos Ácidos Nucléicos Aspectos Moleculares & Enfoques Conceituais HISTÓRICO DA BIOLOGIA MOLECULAR 1866: Gregor Mendel HISTÓRICO DA BIOLOGIA MOLECULAR 1869 → Miescher # Johann Friedrich Buscava determinar os componentes químicos do núcleo celular. # Usava os glóbulos brancos contidos no pus para suas pesquisas (células que apresentam núcleos grandes e fáceis de serem isolados do citoplasma). # Descobriu a presença de um composto de natureza ácida desconhecido até o momento (rico em fósforo e em nitrogênio, desprovido de enxofre e resistente à ação da pepsina - enzima proteolítica)) → nucleína HISTÓRICO DA BIOLOGIA MOLECULAR 1880 → Albrecht Kossel # Demonstrou que a nucleína continha bases nitrogenadas em sua estrutura. 1889 → Richard Altmann # Obteve nucleína com alto grau de pureza, comprovando sua natureza ácida e dandolhe o nome de ácido nucléico. HISTÓRICO DA BIOLOGIA MOLECULAR 1900: Hugo de Vries, Erich von Tschermak e Carl Correns Redescoberta de Mendel Leis da Hereditariedade Leis de Mendel HISTÓRICO DA BIOLOGIA MOLECULAR 1928: Frederick Griffith:Transformação HISTÓRICO DA BIOLOGIA MOLECULAR HISTÓRICO DA BIOLOGIA MOLECULAR 1944: Avery, MacLeod e MacCarty: Princípio transformante HISTÓRICO DA BIOLOGIA MOLECULAR 1953: Alfred Hershey e Martha Chase DNA 32P Proteína 35S HISTÓRICO DA BIOLOGIA MOLECULAR 1953: James Watson e Francis Crick Maurice Wilkins e Rosalind Franklin DNA → INTRODUÇÃO • Entre todas as propriedades dos organismos vivos, a capacidade de auto-replicação é fundamental. • Conter a informação genética significa armazená-la, transmiti-la ao longo das gerações, e expressá-la na forma de proteínas. • Avanços significativos tem sido alcançados na área da Biologia Molecular a partir do isolamento, análise e síntese de seqüências de DNA. • DNA recombinante → estudos de função e dos mecanismos que controlam a expressão gênica ÁCIDO NUCLÉICO → PROTEÍNA DNA → INTRODUÇÃO DNA → INTRODUÇÃO GENOMA HUMANO Autossomos x Sexuais 99% 1% GENOMA HUMANO Nucleotídeo DNA Genoma Cromossomo DEFINIÇÃO DE GENE rRNA Ribossomos tRNA RNP snRNA AA mRNA Proteína ESTRUTURA PRIMÁRIA DO DNA Pentose (Açúcar) R OH Ribose -D-Ribofuranose H 2’-Desoxirribose -D-2-desoxirribofuranose ESTRUTURA PRIMÁRIA DO DNA Bases Nitrogenadas = Anéis Aromáticos Heterocíclicos PURINAS Bicíclicas Purina Adenina Guanina PIRIMIDINAS Monocíclicas Pirimidina Citosina Adenina = Timina Uracil Uracil Timina Guanina Citosina ESTRUTURA PRIMÁRIA DO DNA Curiosidade sobre as Bases Nitrogenadas PIRIMIDINAS Uracil Timina 5-Metiluracil ESTRUTURA PRIMÁRIA DO DNA Posições dos Átomos Grupamento Fosfato Bases Nitrogenadas Pentose ESTRUTURA PRIMÁRIA DO DNA Nucleotídeo (2’-) Desoxirribonucleotídeo Nucleosídeo Ribonucleotídeo Nucleosídeo ESTRUTURA PRIMÁRIA DO DNA Grupamento Fosfato = Ésteres de Fosfato Monofosfato - 5’-Monofosfato de Adenosina - AMP 5’-Monofosfato de Desoxiadenosina - dAMP Difosfato - 5’-Difosfato de Adenosina - ADP 5’-Difosfato de Desoxiadenosina - dADP Trifosfato - 5’-Trifosfato de Adenosina - ATP 5’-Trifosfato de Desoxiadenosina - dATP ESTRUTURA PRIMÁRIA DO DNA Grupamento Fosfato = Ésteres de Fosfato ESTRUTURA PRIMÁRIA DO DNA Bases Nitrogenadas ESTRUTURA PRIMÁRIA DO DNA Sentido da Fita de DNA Base Base Base 5’ 3’ 5’AACGTTGCTATCGT3’ ESTRUTURA PRIMÁRIA DO DNA ESTRUTURA PRIMÁRIA DO DNA Bases Nitrogenadas Grupo Ceto (C=O) e Amino (C-NH2) Relação Molar (1949) (A+G) = (T+C) Chargaff A = 1,0 T C = 1,0 G AT = CG (?) ESTRUTURA PRIMÁRIA DO DNA Ligações Importantes Ligações Fosfodiéster Ligação (-) Glicosídica (Glicosílica) DUPLA-HÉLICE DO DNA Estabilidade do DNA: Integridade e Flexibilidade Ligações Covalentes Forças de Van der Walls Interações Iônicas (Mg+2) Sítios Hidrofóbicos Sítios Hidrofílicos Pontes de Hidrogênio DESNATURAÇÃO E RENATURAÇÃO • Fenômenos físicos que ocorrem com o DNA dupla-hélice fundamentais para os processos de replicação, transcrição e recombinação. • DESNATURAÇÃO → rompimento das pontes de hidrogênio entre as cadeias complementares do DNA. • RENATURAÇÃO → ligamento das pontes de hidrogênio entre as cadeias complementares do DNA. • Esses processos podem ser observados in vitro DESNATURAÇÃO E RENATURAÇÃO A desnaturação da estrutura secundária do DNA pode ser obtida através dos seguintes mecanismos: → aumento de temperatura → titulação com ácidos ou álcalis (protonizam ou desprotonizam os anéis aromáticos) → agentes desnaturantes (formamida) # Tais tratamentos geram grupos carregados no interior da dupla-hélice (levando ao rompimento das pontes de hidrogênio entre as bases complementares). DESNATURAÇÃO E RENATURAÇÃO A desnaturação do DNA pode ser acompanhada pela medida em espectrofotômetro de absorbância de luz ultravioleta (UV). Efeito Hipercrômico DESNATURAÇÃO E RENATURAÇÃO A temperatura necessária para a desnaturação de um dado DNA está diretamente relacionada com sua seqüências de pares de bases. Adenina = Timina Uracil Guanina Citosina Tm (oC) = 69,3 + 0,41(GC%) • Em condições fisiológicas, a dupla-hélice é muito estável. • Para que estes processos ocorram há a necessidade da participação de enzimas especializadas (DNA-helicases e SSBs) DESNATURAÇÃO E RENATURAÇÃO Rompimento das Pontes de Hidrogênio Tm (oC) = 69,3 + 0,41(GC%) T (anelamento) (oC) = Tm – 25oC DESNATURAÇÃO E RENATURAÇÃO • Mesmo quando as duas fitas do DNA estão completamente separadas, o processo pode ser revertido. • Se uma solução contendo DNA desnaturado por calor for lentamente resfriada, as fitas complementares reassociam-se. T (anelamento) (oC) = Tm – 25oC • No início, a renaturação ocorre lentamente. Porém, à medida que as bases complementares se associam, a velocidade do processo aumenta. • Resfriamentos abruptos colapsam a renaturação # Quanto maior a complexidade do genoma, maior será o tempo de sua renaturação. DESNATURAÇÃO E RENATURAÇÃO DUPLA-HÉLICE DO DNA As ligações glicosídicas no DNA, por não estarem diretamente opostas na dupla-hélice, geram duas cavidades desiguais em seu contorno: → cavidade maior → cavidade menor # Nestas regiões, as bases estão expostas ao meio solvente. # Moléculas que agem com seqüências específicas de bases (proteínas) podem identificar estas seqüências sem romper a estrutura da duplahélice. DUPLA-HÉLICE DO DNA Antiparalelas DUPLA-HÉLICE DO DNA Cavidades Maior e Menor Cavidade Menor Cavidade Maior Volta 0.34 nm DUPLA-HÉLICE DO DNA Cavidades Maior e Menor DUPLA-HÉLICE DO DNA Cavidades Maior e Menor TIPOS DE DNA O DNA pode assumir diferentes conformações, dependendo da sua composição de bases e do meio em que se encontra → DNA A → DNA B → DNA Z Tipo A → forma mais abundante encontrada na célula (forma de duplahélice clássica) Tipo B → formado a partir da desidratação ou diminuição do teor de sal no meio em que se encontra o Tipo A. TIPOS DE DNA Tipo Z → encontrado, aparentemente, em apenas algumas regiões do DNA Tipo B ou Tipo A. # Fatores que estabilizam sua formação: → metilação ou bromação de bases → estresse torcional → ligação de proteínas específicas ao DNA • Alterações nas conformações podem facilitar ou dificultar a interação do DNA com proteínas. TIPOS DE DNA TIPOS DE DNA DNA B DNA A DNA Z TIPOS DE DNA DNA B DNA A DNA Z DESNATURAÇÃO E RENATURAÇÃO Conformação anti e syn Umidade Relativa (92%) Dupla RNA DNA B Etanol 75% [Sal] C2’-endo DNA A C3’-endo DNA Z Metilação ou Bromação DESNATURAÇÃO E RENATURAÇÃO Principais características dos DNAs A, B e Z CARACTERÍSTICAS FORMA A FORMA B FORMA Z Sentido helicoidal (Giro) Direita Direita Esquerda Diâmetro (nm) ~2,6 ~2,0 ~1,8 Pb por giro (n) 11 10 12 Espaço entre as bases (nm) 0,26 0,34 0,37 Inclinação da base 20o 6o 7o Sulco maior Estreito/Profundo Largo/Profundo Achatado Sulco menor Largo/Raso Estreito/Profundo Estreito/Profundo Ligação glicosídica anti anti anti(pir) sin(pur) RNA x DNA H H H H RNA x DNA RNA mensageiro (mRNA) Sinal de ligação ao Ribossomo CAU AUG Códon de Iniciação (Start Códon) AGGAGGU Início da Transcrição AAAAA Hairpin Fim da mensagem RNA transportador (tRNA) RNA ribossomal (rRNA) OUTROS RNAs hnRNA snRNA snoRNA RNA DE INTERFERÊNCIA (RNAi) Premio Nobel de Medicina 2006 Andrew Fire e Craig Mello FORMAS DE DNA E SUPERTORÇÃO O DNA nem sempre está na forma de um bastão de dupla-hélice (linear). # Quando as duas extremidades de um DNA ligam-se covalentemente, formam-se as chamadas estruturas circulares. FORMAS DE DNA E SUPERTORÇÃO Linear x Circular Plamídeos e Vírus FORMAS DE DNA E SUPERTORÇÃO • Além da estrutura secundária da dupla-hélice, o DNA assume uma conformação tridimensional supertorcida. # A dupla-hélice enrola-se sob si mesma (extremamente importante nos processos de replicação, transcrição, recombinação e expressão gênica). FORMAS DE DNA E SUPERTORÇÃO Supertorções FORMAS DE DNA E SUPERTORÇÃO Estrutura Supertorcida, Suprerenrolada ou Super-hélice B C Topoisômeros A Grau de superenrolamento crescente A: Relaxada B: Superenrolamentos parciais C: Totalmente Superenrolado FORMAS DE DNA E SUPERTORÇÃO Tipos de Superenrolamentos • Plectonêmico → DNA em solução. • Toroidal → DNA enrolado em proteínas (histonas), para formar os nucleossomos. # O desenrolamento das hélices pode induzir à formação de estruturas criciformes, triplex ou DNA Tipo Z. FORMAS DE DNA E SUPERTORÇÃO Tipos de Superenrolamentos Plectonêmico Solução Toroidal Histonas FORMAS DE DNA E SUPERTORÇÃO TOPOISOMERASES Topoisômeros → moléculas com seqüências e tamanhos idênticos, que diferem apenas na sua topologia. Topoisômerases → enzimas que, quando presentes, promovem a quebra transitória nas pontes fosfodiéster adicionando ou removendo superenrolamentos. • A enzima permanece ligada covalentemente ao DNA e permite que as fitas passem umas sobre as outras. • Estas enzimas mostram-se presentes tanto em células procarióticas como eucarióticas. TOPOISOMERASES Topoisômerase I → quebram apenas uma das fitas de DNA Topoisômerase II → quebram as duas fitas da dupla-hélice Participam de importantes etapas nos processos de replicação, transcrição e recombinação # o grau de superenrolamento, sua geração e remoção em moléculas de DNA pode ser medido de diferentes maneiras. TOPOISOMERASES Topoisomerase do tipo I Relaxamento de Superenrolamento ATP Ligação Fosfotirosina Restauração do DNA Ligação e Desnaturação E. coli Topo I: Monômero de 100 kDa (+ e –) Mutação no Gene topA Inviável Topo III: Monômero de 74 kDa (+) Mutação no Gene topB Viável TOPOISOMERASES Topoisomerase do tipo II E. coli Topo II Tetrâmero 400 kDa DNA-Girase Sub A e B Adiciona Supertorções – Gene gyrA e gene gyrB Topo IV Catenadas Antibióticos e Antitumorais 105 a 140 pb 5’-Tyr(122)A Central: 20 pb (AT) Lateral: Rica em GC FORMAS DE DNA E SUPERTORÇÃO Métodos de aferição do grau de superenrolamento • Eletroforese em gel de agarose → separa as moléculas de DNA em função da sua forma e compactação (funções diretas do superenrolamento) → o DNA migra em géis com diferentes velocidades (dependendo de seu tamanho e forma) # Moléculas de DNA de mesmo tamanho migram com diferentes velocidades se estiverem na forma circular relaxada, linear ou supertorcida. FORMAS DE DNA E SUPERTORÇÃO Métodos de aferição do grau de superenrolamento Velocidade de Sedimentação Gel Agarose Microscopia Eletrônica FORMAS DE DNA E SUPERTORÇÃO Tratamento com Topoisomerase 5 Min 30 Min CONSIDERAÇÕES FINAIS Ácidos Nucléicos maiores representantes do genótipo (DNA) Proteínas maiores representantes do fenótipo DNA Envolvido com todo metabolismo do organismo Molécula da Hereditariedade: Seqüências de bases nitrogenadas Informações DNA Moléculas principais RNA Moléculas intermediárias Proteína Resultado final da informação
Download