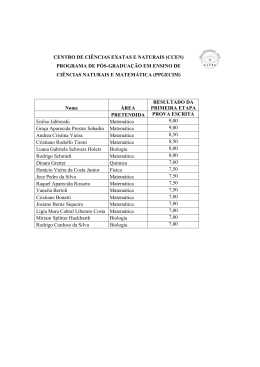
Bioinformática e Sistemas Complexos em Biologia Florencio Pazos Cabaleiro. Centro Nacional de Biotecnología (CNB-CSIC). Madrid [email protected] A aproximação reducionista aplicada a Biologia tem sido, como em outras áreas da ciência, a que mais êxitos tem proporcionado a esta disciplina. Esta aproximação baseia-se em estudar um sistema separando-o em componentes mais simples e fácies de estudar, supondo-se de que as propriedades desse sistema se podem-se obter a partir das propriedades dos componentes individuais. No limite, este método pressupõe que qualquer característica de um sistema poderá em princípio explicar-se a partir dos átomos ou inclusive partículas subatómicas que o compõem e que, nos casos em que este não se tenha podido fazer, o problema está na falta de conhecimento detalhado destes componentes, o que realimenta a abordagem reducionista pois implica que a solução passa por estudar mais a fundo as propriedades destes componentes. No caso da Biologia esta perspectiva reducionista levada ao limite pretenderá, por exemplo, explicar fenómenos tão complexos como a consciência ou as funções cerebrais superiores baseando-se em reacções químicas ou fenómeno atómicos quânticos. A crença na capacidade ilimitada da abordagem reduccionista para resolver problemas científicos é muito lógico se pensarmos no tremendo êxito que tem tido e continua a ter. Na física, a descoberta da estrutura atómica da matéria e o feito de que propriedades de níveis superiores (reacções químicas, propriedades macroscópicas de materiais, etc.) se pudessem explicar tomando como base as propriedades dos átomos fez-me pensar que tudo se poderia explicar seguindo esta aproximação. No caso da Biologia, o advento da Biologia Molecular durante o século XX e seus tremendos êxitos explicando muitas características dos sistemas vivos a partir dos seus componentes moleculares levou a pensar que todos os fenómenos biológicos poderiam chegar a explicar-se assim. Da mesma maneira, dado que as moléculas biológicas estão formadas por átomos, o conhecimento detalhado destas permitiria explicar fenómenos biológicos a qualquer escala. Desta maneira a Biologia, como qualquer outra ciência, convergiria face a Física. Ainda hoje há quem defenda estas posições extremas. As primeiras críticas à abordagem reducionista vêem dos estudos de sistemas complexos. Esses primeiros estudos são muito recentes, dos anos 70, se bem que estão baseados em dados e observações muito anteriores que ninguém se atreveu a interpretar como um erro da aproximação reduccionista. Nestes estudos descobriu-se que certas propriedades de alguns sistemas não poderiam nunca ser preditas a partir de seus componentes já que dependiam de certos tipos de interacções entre eles (retroalimentação, etc.) que requeriam um modelo matemático não-linear. As equações não lineares só podiam ser resultantes por aproximação ou simulação, o que fazia que a mais mínima imprecisão nos dados se amplifique (pela própria alta interdependência do sistema) até resolve-lo. Certas propriedades (emergentes) simplesmente apareciam e nunca se poderia chegar a deduzi-las por muito avançado que estivesse o nosso conhecimento dos componentes do sistema e suas interacções. Não era um problema de falta de conhecimento detalhado ou de falta de ferramentas matemáticas, esta impossibilidade de predizer propriedades do sistema a partir dos componentes era algo intrínseco a própria natureza do sistema. Até então, fenómenos tão quotidianos e de tanta importância prática como turbulências em fluidos, fenómenos atmosféricos, estruturas biológicas, que resistiam à abordagem reduccionista, eram caracterizados de “curiosidades”. A Teoria do Caos e de Sistemas Complexos chegou quando a abordagem molecular a Biologia estava em seu apogeu e isto fez com que se incorporassem mais tarde ás ciências da vida. No entanto, desde o princípio se viu que os sistemas vivos eram um dos protótipos de sistemas complexos. Agora estamos começando a ver claramente, e de maneira dramática em muitos casos, os limites do reducionismo em Biologia. Hoje está claro que muitíssimos fenómenos biológicos nunca poderão ser explicados a partir de componentes moleculares 1,2. De todo não é uma simples soma das partes senão uma função muito mais complexa de estas 3,4. Sabemos agora que as propriedades dos componentes individuais significam muito pouco e só no contexto das complexas interacções com outros adquirem significado biológico. A obtenção do repertório de “componentes” de vários organismos (sequenciação de genomas, proteómica) e a realização de que estes repertórios não pareciam estar, nem em quantidade nem em qualidade, relacionados com a tremenda diferença entre organismos (similar número de genes entre C elegans e humano, quase uma identidade nas sequências de proteínas de humano e ratos) não parece tirar força à ideia de que estes componentes moleculares podiam explicar as características últimas dos organismos 5. Uma característica dos sistemas complexos (entre eles os biológicos) é a “emergência”. Uma propriedade emergente é aquela que surge das intrincadas relações entre componentes pelo que se resiste totalmente a ser deduzida a partir de estes. As propriedades emergentes diferenciam-se das “resultantes”, que podem ser extrapoladas a partir dos componentes. Propriedades emergentes dos sistemas vivos (inclusivamente a um nível muito baixo) são, por exemplo, as características topológicas das redes biológicas (modularidade, centralidade)6-8. A níveis superiores (fisiológicos, mentais) quase todas as propriedades são emergentes e resistem a uma abordagem reducionista: por exemplo, estruturas fisiológicas como veias, folhas de plantas, alvéolos pulmonares, etc., não podem ser deduzidas a partir da informação genética, pois surgem de complicados processos autoorganizativos que implicam inevitavelmente interacções com o ambiente 9. Curiosamente, nestes casos as propriedades de níveis superiores determinam as características dos inferiores 1,10,11: por exemplo: a necessidade de que as redes biológicas sejam modulares e tenham pontos centrais (hubs) determina as características dos componentes moleculares que a formam (modulando-os evolutivamente). Isto é precisamente o contrário da visão reducionista em que, são os componentes de níveis baixos os que determinam as propriedades de níveis superiores, e nunca o contrário. Outra característica destes sistemas é a “robustez”, no sentido de que não se vêem irreversivelmente afectados por certas perturbações que são capazes de se recuperar 5. A robustez consegue-se basicamente por redundância e modularidade12. Isto é curioso dado que a alta interdependência nestes sistemas tende a amplificar qualquer efeito (ver acima), incluídas perturbações, pelo que se esperaria que fossem muito instáveis. A explicação está no conceito de atractor da Teoria do Caos: em muitos sistemas complexos a sua evolução leva-os a um estado dinâmico em que o seu comportamento, ainda que complexo e imprevisível nos detalhes, os faz passar um número limitado e finito de estados (as Perante qualquer perturbação que os tire daí, evoluem regressando a esse atractor. Em Biologia, os atractors têm-se relacionado com conceitos tão importantes como a homeostasia, ou inclusivamente, com os diferentes tipos celulares (tecidos), que se têm interpretado com atractors no espaço de expressões génicas 13, teoria que se está confirmando experimentalmente com dados de DNA arrays 14. Um aspecto onde dramaticamente se manifesta a falha da aproximação reducionista em Biologia, é no tratamento de certas doenças complexas como o cancro. Muitos componentes moleculares implicados no cancro têm-se estudado exaustivamente à escala molecular (sequências, estruturas, mutantes), sem que isso tenha ajudado a apontar soluções globais a esta doença. Isto deve-se, como se disse acima, a que as características moleculares destes componentes significam muito pouco por si só, e só no contexto das suas complexas interacções com outros adquirem significado para esta doença. Outro exemplo, é o desenho de drogas, que também até ao momento se tem baseado primordialmente numa aproximação reducionista. A falha desta perspectiva reflecte-se no decréscimo constante do número de novas drogas que saem no mercado, apesar da crescente inversão da matéria. Outras técnicas terapêuticas prometedoras, que também estão a dar resultados muito por abaixo dos esperados, como a terapia genética, RNA “no-sense”, etc. 15, coincidem com a primeira na perspectiva reducionista (“uma droga – uma “diana”) que não valoriza o suficiente as complexas relações da droga e da “diana” com outros componentes celulares, o que leva a que esta não funcione ou produza efeitos secundários indesejados. A base reducionista destas técnicas terapêuticas, também deveria ter feito com que se beneficiassem enormemente dos projectos de “catalogação” de componentes (sequenciação de genomas, proteómica, genómica estrutural) quando se tem visto claramente que não tem sido assim 16. O desenho racional de vacinas também segue uma abordagem reducionista, reduzindo o complexo processo de “protecção de um organismo contra uma infecção” a um problema de interacção entre duas moléculas (antigenio/vacina - anticorpo) 2. Esta é uma das causas de não se ter encontrado ainda vacina para certos vírus como o HIV a pesar de todo o dinheiro e esforço investidos 17. No caso das vacinas, a aproximação reducionista chegou mais longe com as vacinas de péptidos, cujo fracasso é muito conhecido 18. A própria metodologia em Biologia é altamente reducionista. Um exemplo são as experiências de knockout genético para revelar o papel de um determinado gene. Estas experiências têm dado em muitos casos resultados falsos: genes que se sabe têm funções essenciais cuja função não produz efeito, funções com efeitos totalmente diferentes aos esperados ou aos que se dão em organismos próximos e também estirpes 19. Em muitos casos isto se deve a subvalorizar as características mencionadas de robustez do sistema e o feito com que, muitas vezes, a função biológica de um gene só adquire sentido em contexto de suas complexisimas relações com outros. Também a nível metodológico, a ilimitada confiança nas aproximações in-vitro, e mais recentemente in-silico, a Biologia tem levado a pensar que estas aproximações podiam chegar até ao último conhecimento biológico prescindindo da aproximação in-vivo. A Teoria de Sistemas Complexos diz-nos que, dado que são aproximações, os pequenos detalhes que podemos perder nestes passos podem levar a que muitas das conclusões obtidas não tenham nada a ver com o que ocorre in-vivo. Todas estas novas visões não-reducionistas estão relacionadas com a chamada Biologia de Sistemas 4,5, que estuda os fenómenos biológicos como formados por componentes em complexíssimas relações, onde muitas propriedades do sistema só se podem entender estudando estas relações e as propriedades emergentes que possui (aproximação de alto baixo), e não estudando em detalhe as propriedades dos componentes e tentando predizer as do sistema a partir deles (visão reduccionista). Nesta aproximação a Bioinformática é fundamental já que se requer do tratamento de grandes quantidades de informação (componentes e relações). Esta nova disciplina também necesita de novas ferramentas experimentais (quase todas as actuais estão desenhadas pensando numa abordagem reducionista), como os DNA microarrays, a nanotecnología, microfluidos 20, etc. Os DNA array são o exemplo perfeito de estudo biológico desde o punto de vista de sistemas já que nesta técnica se perde precisão en la monitorización de los valores de expresión de los genes individuales (componentes) para obtener “fotografías” globales del “espacio de expresión génica” del sistema y sus cambios. A visão anti-reduccionista da Biologia é recente e, lamentavelmente, os seus esforços tem-se centrado mais em demostrar as limitações da perspectiva reducionista (ás vezes demasiado criticamente 11) que em dar realmente soluções. No entanto, a aproximação não-reducionista no tratamento de problemas biológicos também está a dar resultados, tanto de conhecimento básico como aplicado. Entre o conhecimento básico que esta aproximação tem produzido, destaca-se o obtido do estudo de redes biológicas 12,21. Da rede de interacção entre proteínas tem-se estudado a sua topologia, distribuição e número de interacções, localização de pontos críticos, taxa evolutiva dos distintos tipos de pontos, tolerância da rede a perturbações e erros, e comparação da organização das redes em distintos organismos 6-8,22-26. Sobre as redes metabólicas, os numerosos estudos incluem descrições da sua topologia e conectividade 23, carácter modular e a relação destes módulos com vias metabólicas “clássicas” 27, evolução da rede 28,29, características e evolução da rede de metabolismo secundário 30. Sobre as redes de regulação génica tem-se estudado a sua composição em motivos 31 donde activações e repressões se combinam em complexas funções para modular e resultado final 32,33. Também se tem visto que estes motivos têm surgido independentemente partindo de componentes distintos, por evolução convergente e em distintos organismos, mercê a sua importância 34,35. Enquanto as aplicações práticas também houve várias aproximações desde este novo ponto de vista a doenças: Um modelo sobre a rede de interacções metabólicas e genéticas relacionadas com a utilização de galactose em levedura permitiu formular hipóteses sobre doenças humanas complexas 22. Algo tão complexo como o cancro de próstata, está a começar a estudar com DNA arrays de milhões de sequências 20 e tem-se visto que meio milhar de factores de transcrição estão relacionados com este processo, tornando impossível qualquer tratamento ou aproximação reduccionista ao problema (baseada num só target). Nesta doença também se tem visto que, dada a tremenda complexidade do processo, o uso de um grupo grande de moléculas e das suas relações como marcadores tumorais (em análises de sangue, etc.) é mais eficaz que o uso de marcadores simples 20. Em geral está-se passando de uma visão em que os targets são proteínas ou genes, a uma em que são subredes biológicas, dado que a doença perturba a subrede inteira 20,22. A abordagem nãoreducionista não tenciona acabar com a abordagem reducionista, mas sim, complementá-lo, de maneira que se aplique a aproximação adequada a cada problema biológico e a cada nível de complexidade. A visão diametralmente oposta ao reduccionismo, holismo extremo, que considera que tudo depende de tudo e que portanto nunca poderemos predizer nada, também não nos é útil. Esta visão anti-reduccionista da Biologia também implica que esta deve manter-se como una disciplina à parte com as suas próprias vocabulário e conceitos distintos da Física ou da Química 2. Referencias 1. Oltvai, Z. N. & Barabási, A.-L. (2002). Life’s complexity pyramid. Science. 298, 763–764. 2. Van Regenmortel, M. H. (2004). Reductionism and complexity in molecular biology. Scientists now have the tools to unravel biological and overcome the limitations of reductionism. EMBO Rep. 5, 1016-1020. 3. Nurse, P. (2003). Systems biology: understanding cells. Nature. 424, 883. 4. Kitano, H. (2002). Computational systems biology. Nature. 420, 206-210. 5. Kitano, H. (2002). Systems Biology: A Brief Overview. Science. 295, 1662-1664. 6. Jeong, H., Mason, S. P., Barabási, A. L. & Oltvai, Z. N. (2001). Lethality and centrality in protein networks. Nature. 411, 41-42. 7. Guelzim, N., Bottani, S., Bourgine, P. & Képès, F. (2002). Topological and causal structure of the yeast transcriptional regulatory network. Nature Genet. 31, 60-63. 8. Wuchty, S., Oltvai, Z. N. & Barabasi, A. L. (2003). Evolutionary conservation of motif constituents in the yeast protein interaction network. Nat Genet. 35, 176-179. 9. Cho, A. (2004). Life's Patterns: No Need to Spell It Out? Science. 303, 782-783. 10. Kim, J. (1999). Making sense of emergence. Philos Stud. 95, 3-36. 11. Lewontin, R. (2000). The Triple Helix. Gene, Organism and Environment, Harvard University Press, Cambridge, MA, USA. 12. Alm, E. & Arkin, A. P. (2003). Biological networks. Curr Opin Struct Biol. 13, 193-202. 13. Kauffman, S. A. (1993). The Origins of Order, Oxford University Press, New York. 14. Huang, S., Eichler, G., Bar-Yam, Y. & Ingber, D. E. (2005). Cell Fates as High-Dimensional Attractor States of a Complex Gene Regulatory Network. Phys Rev Letters. 94, 128701. 15. Glassman, R. H. & Sun, A. Y. (2004). Biotechnology: identifying advances from the hype. Nat Rev Drug Discov. 3, 177-183. 16. Drews, J. (2003). Strategic trends in the drug industry. Drug Discov Today. 8, 411-420. 17. Burton, D. R. & Moore, J. P. (1998). Why do we not have an HIV vaccine and how can we make one? Nat Med. 4, 495-498. 18. Van Regenmortel, M. H. (2001). Pitfalls of reductionism in the design of peptide-based vaccines. Vaccine. 19, 2369-2374. 19. Pearson, H. (2002). Surviving a knockout blow. Nature. 415, 8-9. 20. Hood, L., Heath, J. R., Phelps, M. E. & Lin, B. (2004). Systems biology and new technologies enable predictive and preventative medicine. Science. 306, 640-643. 21. Barabasi, A. L. & Oltvai, Z. N. (2004). Network biology: understanding the cell's functional organization. Nat Rev Genet. 5, 101-113. 22. Ideker, T., Thorsson, V., Ranish, J. A., Christmas, R., Buhler, J., Eng, J. K., et al. (2001). Integrated genomic and proteomic analyses of a systematically perturbed metabolic network. Science. 292, 929-934. 23. Jeong, H., Tombor, B., Albert, R., Oltvai, Z. N. & Barabási, A. L. (2000). The large scale organisation of metabolic networks. Nature. 407, 651-653. 24. Fraser, H. B., Hirsh, A. E., Steinmetz, L. M., Scharfe, C. & Feldman, M. W. (2002). Evolutionary rate in the protein interaction network. Science. 296, 750-752. 25. Kelley, B. P., Sharan, R., Karp, R. M., Sittler, T., Root, D. E., Stockwell, B. R., et al. (2003). Conserved pathways within bacteria and yeast as revealed by global protein network alignment. Proc Natl Acad Sci U S A. 100, 11394-11399. 26. Aldana, M. & Cluzel, P. (2003). A natural class of robust networks. Proc Natl Acad Sci U S A. 100, 8710-8714. 27. Ravasz, E., Somera, L., Mongru, D. A., Oltvai, Z. N. & Barabási, A. L. (2002). Hierarchical organization of modularity in metabolic networks. Science. 297, 1551-1555. 28. Schmidt, S., Sunyaev, S., Bork, P. & Dandekar, T. (2003). Metabolites: a helping hand for pathway evolution? Trends Biochem Sci. 28, 336-341. 29. Alves, R., Chaleil, R. A. G. & Sternberg, M. J. E. (2002). Evolution of enzymes in metabolism: a network perspective. J Mol Biol. 320, 751-770. 30. Pazos, F., Valencia, A. & De Lorenzo, V. (2003). The organization of the Microbial Biodegradation Network from a Systems-Biology perspective. EMBO Rep. 4, 994-999. 31. Milo, R., Shen-Orr, S. S., Itzkovitz, S., Kashtan, N., Chklovskii, D. & Alon, U. (2002). Network motifs: simple building blocks of complex networks. Nature. 298, 824-827. 32. Shen-Orr, S. S., Milo, R., Mangan, S. & Alon, U. (2002). Network motifs in the transcriptional regulation network of Escherichia coli. Nature Genet. 31, 64-68. 33. Setty, Y., Mayo, A. E., Surette, M. G. & Alon, U. (2003). Detailed map of a cis-regulatory input function. Proc Natl Acad Sci U S A. 100, 7702-7707. 34. Conant, G. C. & Wagner, A. (2003). Convergent evolution of gene circuits. Nat Genet. 34, 264266. 35. Babu, M. M. & Teichmann, S. (2003). Evolution of transcription factors and the gene regulation network in Escherichia coli. Nucleic Acids Res. 31, 1234-1244.
Download