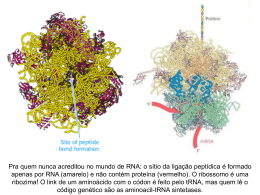
TRADUÇÃO PROTEICA Tradução é o processo de leitura da seqüência de mRNA e sua conversão em uma seqüência de aminoácidos. A tradução ocorre no citoplasma e ocorre em organelas citoplasmáticas chamadas ribossomos. A seq de nucleotídeos de uma molécula de mRNA é “lida” da ponta 5’ para 3’ em grupo de três (códons). Código Genético 5’ ... ... AUU CCG UAC GUA AAU UUG ... ... 3' códon códon códon códon códon códon Como existem 4 nucleotídeos, existem 4x4x4 = 64 códons diferentes, cada um codificando um AA ou um sinal de término da tradução (stop-codons). Código Genético 20 AAs + 3 stopcodons Número de letras no código O código genético possui 20 AAs comumente encontrados nas ptns celulares. 1 base = 4 AAs possíveis X 2 bases = 16 AAs possíveis X 3 bases = 64 AAs D--- excesso de “palavras” – código genético é redundante, isto é, alguns AAs devem ser especificados por 2 ou + codons diferentes, pois se apenas 20 codons fossem usados (sendo os outros 44 stop-codons), então a maioria das mudanças de matriz de leitura deve produzir palavras sem sentido, o q pararia o processo de síntese de ptns. Matriz de Leitura (Open Read Frame - ORF) Uma seq de nucleotídeos pode, em princípio, ser lida em qualquer de suas 3 janelas de leitura, mas a maioria das seqs de DNA codifica um produto protéico em apenas uma janela de leitura. Na janela de codificação não pode haver nenhum stop-codon e cada códon deve corresponder ao AA apropriado. Mas algumas vezes um AA pode ser substituído por outro em outra janela e ainda reter a fç da ptn, tornando plausível a utlização de outras janelas de leitura. Há exceções, como por exemplo em alguns vírus. 5' 3' atgcccaagctgaatagcgtagaggggttttcatcatttgaggacgatgtataa 1 atg ccc aag ctg aat agc gta gag ggg ttt tca tca ttt gag gac gat gta taa M P K L N S V E G F S S F E D D V * 2 tgc cca agc tga ata gcg tag agg ggt ttt cat cat ttg agg acg atg tat C P S * I A * R G F H H L R T M Y 3 gcc caa gct gaa tag cgt aga ggg gtt ttc atc att tga gga cga tgt ata A Q A E * R R G V F I I * G R C I O Código Genético é Universal Os códons têm o mesmo significado, com pequenas exceções, em todas as espécies. As exceções mais importantes à essa universalidade ocorrem nas mitocôndrias dos mamíferos, nas leveduras e em várias outras espécies. Nas mitocôndrias dos mamíferos: - UGA especifica Trp - AUA especifica uma Met e não Ile - AGA e AGG são códons de término e não códons de Arg tRNA Cada AA é trazido ao ribossomo por uma molécula específica de RNA transportador (tRNA) que se pareia a um códon do mRNA. O pareamento do segmento de 3 nucleotídeos de tRNA , chamado anticódon, é feito com o códon. O tRNA tem dois sítios importantes: o anticódon e o sítio de ligação do aminoácido (extremidade 3’). Os outros dois braços ajudam a ligação do tRNA ao ribossomo. As moléculas de tRNA tem similaridades estruturais, mas cada uma tem uma forma 3D única, que permite o reconhecimento pela sintetase correta, a qual catalisa a união de um tRNA com seu aminoácido específico para formar um aminoacil-tRNA (tRNA + AA). Amino Acid Activation by Aminoacyl-tRNA Synthetase In two chemical steps, this enzyme catalyzes the formation of an ester bond between the carboxyl group of an amino acid and the 3 foot OH of the appropriate tRNA. (1) The amino acid and a molecule of ATP enter the active site of the enzyme. Simultaneously, ATP loses pyrophosphate, and the resulting AMP bonds covalently to the amino acid. The pyrophosphate is hydrolyzed to 2P(sub)i. (2) The tRNA covalently bonds to the amino acid, displacing the AMP. The aminoacyl tRNA is then released from the enzyme. Degeneração do Código Genético Algumas espécies de tRNA podem levar seus AAs específicos em resposta a vários códons, e não apenas por um, por um tipo frouxo de pareamento em uma ponta do códon e anticódon. Este pareamento é chamado de oscilante. A oscilação é causada pelo 3º nucleotídeo de um anticodon q não fica bem alinhado. Este nucleotídeo desalinhado às vezes pode formar pontes de H não só com seu nt complementar normal na 3ª posição do codon, mas também com um nucleotídeo diferente nesta posição. SÍNTESE DE PROTEÍNAS Resumo: 1- Cada AA é ligado a uma molécula específica de tRNA por uma ligação de alta energia derivada do ATP. O processo é catalisado por uma enzima específica chamada de sintetase. Há uma sintetase específica para cada AA. 2- A energia do tRNA carregado é convertida em uma ligação peptídica que une o AA a outro no ribossomo. 3- Novos AAs são ligados por meio de ligações peptídicas à cadeia em crescimento 4- Este processo continua até AAn (o AA final) ser adicionado. O processo total só funciona na presença de mRNA, ribossomos, vários fatores protéicos adicionais, enzimas e íons inorgânicos. Ribossomo Consiste de 2 subunidades que nos procariontes, se sedimentam como partículas 50S (subunidade maior) e 30S (subunidade menor) e se associam para formar uma partícula 70S. Nos eucariotos são as subunidades 60S e 40S que formam a partícula 80S. Os ribossomos contém sítios específicos que os possibilitam se ligar ao mRNA, aos tRNAs e aos fatores proteicos específicos necessários à síntese de ptns. O mRNA liga-se à subunidade 30S. Os tRNAs se ligam a dois sítios no ribossomo: Sítio A (aceptor) – sítio de entrada de um aminoacil-tRNA. Sítio P (peptidil) – o peptidil-tRNA se liga levando a cadeia polipeptídica em crescimento para o novo aminoacil-tRNA, formando uma nova ligação peptídica Sítio E ? (saída) – onde o tRNA desacilado é liberado. Estágios da Síntese Proteíca 3 etapas: Iniciação - ligação do ribossomo ao mRNA e ligação do 1º tRNA. Alongamento - translocação do ribossomo e formação de todas as ligações peptídicas. Término - liberação da cadeia peptídica, dissociação do ribossomo ao mRNA. Iniciação Componentes: Iniciação Procariotos: Além do mRNA, dos ribossomos e das moléculas específicas de tRNA, a iniciação necessita da participação de vários fatores,chamados fatores de iniciação IF1, IF2 e IF3. Na maioria dos procariotos o primeiro AA em qq novo polipeptídeo sintetizado é uma N-formilmetionina. Ele é inserido por um tRNA iniciador chamado tRNAfMet. Portanto o códon AUG na maioria das vezes é o códon iniciador (pode ser tb GUG e em raras vezes UUG). Esse códon é reconhecido pelo tRNAfMet, e a Metionina aparece como o 1º AA da cadeia. 1. Ligação do mRNA à subunidade 30S, estiulada pelo IF3. Qdo não estão participando da síntese de ptns,as subunidades ribossomais existem sob a forma livre. 2. IF2 se liga a GFP e ao fMet-tRNA e estimula sua ligação ao complexo de iniciação, levando o fMet-tRNA para o sítio P. 3. GFP é quebrado por um ptn ribossomal, liberando energia para ativar a montagem das 2 subunidades ribossomais. Então IF2 e IF3 são liberados. IF1: seu papel não está claro, parece participar na reciclagem do ribossomo. Como o códon correto de iniciação é selecionado dentre os muitos códons AUG e GUG no mRNA? Os verdadeiros códons de iniciação são precedidos de seqs que se pareavam bem com a ponta 3’ do rRNA 16S (Seqüência de Shine-Delgarno) rica em purina. Há uma separação curta variável entre a seq de Shine-Delgarno e o códon de iniciação Structure of a typical bacterial mRNA molecule. Unlike eucaryotic ribosomes, which typically require a capped 5 end, procaryotic ribosomes initiate transcription at ribosome-binding sites (Shine-Dalgarno sequences), which can be located anywhere along an mRNA molecule. This property of ribosomes permits bacteria to synthesize more than one type of protein from a single mRNA molecule Eucariotos: - Shine-Delgarno não está presente em eucariotos, e esta é uma diferença significativa entre os 2 sistemas de tradução. - Em eucariotos, a sub-unidade menor do ribossomo se associa à extremidade 5’ do mRNA. (reconhecimento do 5’Cap) e “escaneia” este até achar o primeiro AUG, onde então a subunidade maior se associa. O AUG de eucariotos deve estar num contexto específico GCCA/GCCAUGG. - Em alguns vírus que infectam eucariotos (ex. picornavirus), existem sequências internas para iniciação de tradução (IRES) Æ destrói Cap dos mRNAs. - O tRNA-Met inicial não é formilado, mas sua estrutura tambem é diferente da do tRNA-Met do meio da cadeia, responsável pela especificidade da ligação. AIongamento As etapas do alongamento são auxiliadas por 3 fatores proteicos: EF-Tu, EF-Ts e EFG. 1- EF-Tu medeia a entrada do aminoaciltRNA no sítio A. Para isso EF-Tu se liga a GTP e se liga ao tRNA. GTP é quebrado e ativa a ligação do aminoacil-tRNA no sítio A e EF-TU então é liberado. 2- EF-Ts medeia a ligação EF-Tu – GDP do ribossomo e a regeneração de EF-Tu-GTP. 3- Na etapa de translocação a cadeia polipeptídica do peptidil-tRNA do sítio P é transferida para o aminoacil-tRNA no sítio A, em uma reação catalisada pela peptidiltransferase. O ribossomo então se move um códon ao longo do mRNA, indo no sentido 5’3’. Esta etapa é mediada pelo ET-G e é necessária a quebra de GTP para liberar o tRNA descarregado do sítio P e para a transferência do peptidil-tRNA para o sítio A. A cadeia polipeptídica cresce no sentido N-terminal l C-terminal, pois a ligação peptídica ocorre com a ligação da região N-terminal do AAn+1 à região C-terminal do AAn . Eucariotos Em eucariotos o alongamento é semelhante ao dos procariotos. Terminação 3 códons de término: UAG, UAA e UGA. Não são reconhecidas por um tRNA, mas por fatores proteicos (fatores de liberação). Em procariotos os fatores de liberação são RF1 e RF2. RF1: reconhece as trincas UAA e UAG. RF2: reconhece as trincas UAA e UGA. RF3: ajuda a catalisar o término da cadeia, não reconhece os stop-codons. Em eucariotos um único fator de liberação (eRF) reconhece todos os 3 códons de término. Quando o peptidil-tRNA está no sítio P, os fatores de liberação, em resposta aos códons de término, ligam-se ao sítio A. O polipeptídeo é então liberado do sítio P, e o ribossomo se dissocia em duas subunidades, em uma reação ativada pela hidrólise de uma molécula de GTP.
Baixar