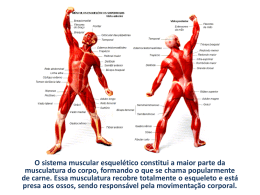
Pergunta 1 Parte A Você está estudando um receptor chamado Receptor do Fator de Tamanho ou SF-R em um eucariota haplóide. Abaixo encontra-se um diagrama esquemático da proteína SF-R: A fim de ser localizado na membrana plasmática, o SF-R deve primeiro passar por várias etapas dentro da célula. a) Desenhe uma configuração do SF-R na forma como ele iria aparecer na vesícula de transporte (após deixar o RER) e na membrana plasmática abaixo. Não se esqueça de rotular os domínios apropriados e as terminações N e C da proteína. 8 PONTOS b) A Partícula de Reconhecimento de Sinal (SRP) direciona o peptídeo nascente SF-R para ... (circule uma). Complexo de Golgi 2 PONTOS Retículo Endoplasmático Núcleo Lisossoma 1 Nome ______________________________ Pergunta 1 (cont.) Parte B O SF-R foi identificado como um receptor da tirosina-quinase. Quando o ligando SF se liga a SF-R, uma cascata de transdução de sinal é ativada. A sinalização através de SF-R leva a uma expressão de proteínas necessária para um tamanho normal de célula. Se a sinalização através de SF-R sofre ruptura, o tamanho da célula é menor. Para estudar os domínios da proteína SF-R com mais detalhes, você isola três mutantes SF-R. Você cultiva cada um dos mutantes na presença de SF e mede duas propriedades: a localização SF-R e o tamanho da célula. c) Indique onde cada uma das mutações em SF-R, [(S), (L), (T), (K), descritas abaixo], seria consistente com os fenótipos observados relacionados para os mutantes no gráfico a seguir. (S) deleção de domínio de seqüência de sinal apenas (L) deleção de domínio de ligação a ligando apenas (T) deleção de domínio transmembrana apenas (K) deleção de domínio quinase apenas 12 PONTOS Fenótipos Linhagem Localização SF-R Tamanho da Célula Mutação(ões) Possível(is) Tipo Selvagem membrana plasmática normal nenhuma Mutante 1 membrana plasmática pequeno L, K Mutante 2 citoplasma pequeno S Mutante 3 fora da célula / secretado pequeno T 2 Pergunta 1, Parte B (cont.) A figura a seguir representa a cascata de sinalização na célula que leva à expressão de genes que determinam o tamanho da célula. d) Se você acidionar uma forma de SF que se liga irreversivelmente a SF-R a células de tipo selvagem, que efeito isso terá na trajetória de sinalização abaixo do SF-R? (Circule uma das seguintes alternativas). 3 PONTOS Nenhum Efeito Sempre Ativa Sempre Inativa e) Você isola DNA de outra linhagem mutante e determina a seqüência de seu gene SF-R. Você descobre que há mutações que mudam os dois aminoácidos tirosina (Y) no domínio plasmático para alanina (você pode assumir que essas mutações não afetam a dobra ou a localização do SF-R). Que efeito isso terá na trajetória de sinalização abaixo do SF-R? Nenhum Efeito Sempre Ativa Sempre Inativa 3 Nome ___________________________ Pergunta 2 Interessado em descobrir por que os Red Sox apresentam uma performance tão insatisfatória, você analisa amostras de sangue de jogadores atuais do Red Sox. Você descobre uma proteína única, à qual dá o nome de “YysSk”, encontrada apenas nos jogadores do Red Sox, que pode explicar sua performance decepcionante. A caracterização completa dessa proteína iria exigir a retirada de litros de sangue dos jogadores (o que dificilmente iria melhorar sua performance), então você decide expressar a proteína YysSk nas bactérias. Você faz a triagem de uma biblioteca de cDNA e identifica um clone positivo ao qual dá o nome de pYS. Você conhece a seqüência do plasmídeo de DNA adjacente ao inserto de cDNA (mostrado abaixo). A fim de determinar a seqüência desse inserto, você usa os seguintes primers. Primer 1: 5’ TCAGTC 3’ Primer 2: 5’ CGGCGC 3’ a) Qual dos seguintes itens seria uma seqüência válida para o filamento superior de seu inserto de cDNA, conforme determinado nos géis seqüenciadores acima? Circule um. 4 PONTOS 4 Pergunta 2 (cont.) Agora, você deseja expressar essa proteína em bactérias. O clone cDNA, pYS, que você isolou é apresentado abaixo. Esse plasmídeo atua como um promotor bacteriano, então você planeja clonar o gene YysSk em pUC19, que possui um promotor bacteriano em si (ver abaixo). Você digere pYS e pUC19 com Bg1II, executa as reações em gel de agarose juntamente com os plasmídeos não-digeridos (não-cortados). Depois de fazer a coloração para DNA, você vê a imagem abaixo. b) Quais fragmentos você purificaria para misturar para ligação? Circule os fragmentos corretos no gel acima. 4 PONTOS c) Você transforma células com sua mistura de ligação. O que você deve acrescentar ao meio em placa de Petri para isolar somente bactérias que tomaram um plasmídeo? 3 PONTOS AMPICILINA 5 Nome _________________________________ Pergunta 2 (cont.) Você pega três colônias (X, Y e Z), isola o DNA de seu plasmídeo e realiza uma análise de restrição. O gel tingido é apresentado abaixo. d) Indique o plasmídeo contido em cada colônia. Escolha entre os plasmídeos mostrados ao fim desta página (p1, p2 ou p3). 5 PONTOS Colônia X contém p1 Colônia Y contém p3 Colônia Z contém p2 e) Qual dos plasmídeos abaixo você conseguiu encontrar por “clonagem por hibridização” usando o DNA YksSk como sonda? 2 PONTOS p1 e p3 f) Qual plasmídeo você usaria para purificar a proteína YksSk? 3 PONTOS p3 6 Pergunta 2 (cont.) Para fins de purificação de proteína, você gostaria de adicionar quatro histidinas à terminação N de YksSk, já que essas histidinas se ligam fortemente ao níquel, permitindo a você separar YksSk de proteínas bacterianas. g) Dadas as seguintes terminações de código no gene YksSk, quais dois primers PCR você escolheria para amplificar o gene YksSk e adicionar quatro histidinas na terminação N de YksSk? (Obs.: O códon para histidina é CAU). Gene YksSk 5’ ATG CGG ATA CGT …..… CGG CTT CCC TAA 3’ 3’ TAC GCC TAT GCA …..… GCC GAA GGG ATT 5’ 6 PONTOS Circule um primer de cada coluna para amplificação PCR. h) Você escolhe seus primers e os adiciona a seu tubo PCR, assim como um modelo de DNA YksSk e polimerase Taq. Você programa sua máquina de PCR para realizar 30 seqüências de amplificação. Após executar sua reação em gel de agarose e tingir o gel para DNA, você vê... nada! Seu parceiro de laboratório, que viu você fazer tudo isso, dá uma risadinha e aponta seu grande erro. Qual foi ele? 3 pontos Você se esqueceu de adicionar nucleotídeos. 7 Nome __________________________________ Pergunta 3 O gene lacZ codifica a enzima β-galactosidase, que metaboliza a lactose em E. coli. O óperon lac em E. coli é transcricionalmente regulado pela proteína repressora Lac I, cuja ação é inibida pela ligação da lactose. A região cromossômica do óperon é mostrada abaixo. PI lacI PIAC O lacZ é o promotor para o gene lacI codifica a proteína repressora é a seqüência promotora para lacZ é a seqüência operadora codifica β-galactosidase, a enzima que quebra a lactose Os mutantes a seguir são feitos e examinados por seus níveis de β-galactosidase na presença e ausência de lactose. “+” “-““OC” significa tipo selvagem significa uma mutação que reduz a função significa um operador que confere expressão constitutiva de lacZ a) Indique o nível de β-galactosidase para cada genótipo, usando os termos ALTO ou BAIXO. 6 PONTOS genótipo I+ Plac+ O+ Z+ I- Plac+ O+ Z+ I+ Plac+ Oc Z+ I+ Plac- O+ Z+ - lactose baixo alto alto baixo + lactose alto alto alto baixo b) Preencha a tabela para os seguintes diplóides baterianos. 8 pontos Plasmídeo F’ Cromossomo I+ Plac+ O+ Z+ I+ Plac+ O+ Z+ I+ Plac+ O+ Z+ I+ Plac+ O+ Z+ I+ Plac+ O+ Z+ I+ Plac+ O+ Z+ I+ Plac+ O+ Z+ I+ Plac+ O+ Z+ I+ Plac+ O+ Z+ I+ Plac+ O+ Z+ 1. 2. 3. 4. 5. - lactose + lactose baixo alto alto alto baixo alto baixo baixo baixo alto c) Circule os componentes na lista abaixo que atuam APENAS em cis. lacI Plac O lacZ 8 Pergunta 4 O gráfico a seguir ilustra uma ação potencial medida em um único ponto ao longo de um axônio. Quatro pontos são realçados ao longo da curva, ã, ~, ♥, ö. a) Na tabela abaixo, identifique qual íon (Na+, K+, Ca++, Cl-) está passando pelo maior fluxo líquido através da membrana nos pontos indicados e afirme a direção em que o íon está se movendo (para o interior ou exterior da célula). 6 pontos Ponto ~ ♥ Íon Na+ Direção (dentro/fora) dentro K+ fora b) O potencial de membrana é de 70 mV nos pontos ã e ö no gráfico acima. Qual dos canais iônicos ativados por voltagem é fechado no ponto ã, mas aberto no ponto ö? 3 pontos canal k+ ativado por voltagem c) O que determina o fechamento do canal ativado por voltagem que é aberto no ponto ~? 3 pontos TEMPO d) Há pelo menos três estados em que existem canais Na+ dependentes de voltagem. No ponto ♥ do gráfico acima, a maioria dos canais Na+ dependentes de voltagem estaria em que estado? Circule a melhor resposta. 3 pontos Aberto Fechado Inativado 9 Nome _________________________ Pergunta 4 (cont.) Dois neurônios pré-sinápticos diferentes, neurônio 1 e neurônio 2, fazem sinapse para a célula W, conforme mostrado abaixo. Quando o neurônio 1 é estimulado, a membrana da célula W é localmente despolarizada. Quando o neurônio 2 é estimulado, a membrana da célula W é localmente despolarizada exatamente na mesma extensão que a observada com o neurônio 1. e) Circule a única afirmação verdadeira abaixo. 3 pontos • Se a estimulação é igual, o neurônio 1 apresenta mais probabilidade de resultar em uma ação potencial na célula W que o neurônio 2. • Se a estimulação é igual, o neurônio 2 apresenta mais probabilidade de resultar em uma ação potencial na célula W que o neurônio 1. • Se a estimulação é igual, o neurônio 1 e o neurônio 2 apresentam a mesma probabilidade de resultar em uma ação potencial na célula W. f) Se você fosse exposto a uma toxina que bloqueasse de forma irreversível os canais de Ca++ ativados por voltagem, indique se as afirmações a seguir seriam VERDADEIRAS ou FALSAS. 6 pontos V F A sinapsina amarra as vesículas secretórias. V F Seus músculos terminam em uma contração rígida. V F As vesículas secretórias preenchidas com neurotransmissores iriam se fundir com a membrana plasmática. 10 11
Download