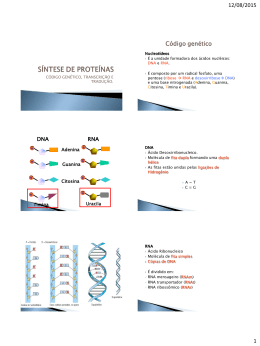
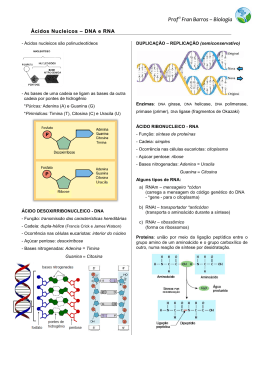
CENTRO DE CIÊNCIAS NATURAIS E EXATAS – CCNE DEPARTAMENTO DE BIOLOGIA DISCIPLINA DE GENÉTICA AGRONOMIA Unidade 1 – Genética Molecular 1. Introdução Ao se analisar um indivíduo, seja uma planta, seja um animal, o que se vê é o conjunto de fatores que ao agirem, cada um há seu tempo, produzem o que se denomina fenótipo. Esse conjunto é composto pelos componentes celulares, sobretudo pelo núcleo, além de um componente chamado ambiental. O núcleo é o que age de forma decisiva na expressão do fenótipo, ou aparência do indivíduo, pois ele contém o que se denomina a molécula da vida, ou DNA. Mas o que tem esse DNA que faz com que as ervilhas de Mendel sejam amarelas ou verdes, lisas ou rugosas? Que o feijão tenha flores roxas ou brancas e que suas sementes sejam pretas, marrons ou brancas? O que tem esse DNA que faz o animal engordar mais rápido num bom pasto, em relação a outros animais que não engordam tanto com a mesma forragem? Que estruturas moleculares contribuem para fazer com que esse fenótipo se manifeste diferentemente em épocas específicas de desenvolvimento dos indivíduos? O que faz que tecidos de crescimento vegetativo se transformem em reprodutivos e, por último, como isso passa através das gerações? A Genética Molecular consegue responder a essas questões, inclusive estabelecendo relações com a Fisiologia Vegetal. Como se trabalha apenas com exemplos vegetais tentar-se-á usá-los, em sua maioria, para explorar essas questões e evidenciar a importância de se conhecer intimamente o DNA, sua composição e sua transmissão através das gerações. Na década de 50 a estrutura da molécula de DNA foi descoberta por Watson e Crick que estabeleceram um modelo de conformação dessa molécula que se encontra no núcleo das células dos vegetais e animais, nos seres humanos e procariontes (há vírus que tem o RNA como material genético no lugar do DNA). O modelo da estrutura do DNA atualmente é muito divulgado, dada sua grande importância em todas as áreas relacionadas com a Biologia, como a Física, a Química, a Bioquímica e a Fisiologia Vegetal. Pode-se nesse momento dizer que genes e enzimas estabelecem um par perfeito para o funcionamento celular, pois um está em estreita relação com o outro. O presente capítulo tem por objetivo descrever a funcionalidade do DNA do núcleo das células e como os genes, que estão no DNA, se transformam em proteínas para o funcionamento das plantas. 2. O gene e a Enzima Ao se cruzar cultivares de feijão (Phaseolus vulgaris L.) que têm flores brancas, que sejam homozigotas, obtém-se a primeira geração filial, a F1, com flores de cor púrpura. Sabe-se que a geração F1 é, por excelência, heterozigota, derivada do cruzamento entre paternais homozigotos, portanto possuem em seu genótipo as duas formas alélicas em todos seus genes. Ao se cruzar as plantas da F1 entre si obtêm-se a geração F2, onde se percebe que a proporção de flores púrpuras para brancas é de 9:7. Com essa proporção chega-se a concluir que são dois genes que estão agindo para a manifestação do fenótipo e que o produto proteico resultante possui uma interação do tipo Epistasia (Ver Unidade 5). É de conhecimento que na epistasia um alelo de um gene pode inibir o outro, ou que, quando os dois alelos ou apenas um de um gene não está presente no genótipo, o fenótipo fica alterado. Para melhor se entender a proporção epistática mencionada o tabela 1.1 demonstra a segregação fenotípica e a relação com a proporção epistática dos genes. 1 Unidade 1 Genética Molecular Tabela 1.1 – Demonstração do genótipo, fenótipo e proporção epistática em F2 com dois genes interagindo entre si. Número 9 3 3 1 Genótipo A- BA- bb aa Baa bb Fenótipo Púrpura Branca Branca Branca Proporção Epistática 9 7 Portanto para a produção da cor púrpura os dois alelos dominantes A e B deverão estar nos mesmos indivíduos. Na falta de um deles a cor passa a ser branca. O que têm estão esses dois alelos para produzirem a cor púrpura? A cor das flores depende dos pigmentos que são produzidos e esses derivam de rotas metabólicas específicas de transformações de substratos. E a transformação dos substratos depende de enzimas. As enzimas são proteína com atividade catalítica constituídas de sequências de aminoácidos. Para que a sequência de aminoácidos funcione como uma enzima é necessária ter uma informação prévia que dite onde se colocará a alanina ou a serina, se a metionina deve ou não ficar na sequência. E quem determina isso tudo é o DNA que é constituído de um conjunto ordenado de nucleotídeos, que são os precursores para a formação das cadeias de proteínas. Então para se responder como a cor púrpura é produzida deve-se levar em consideração que um alelo dominante de um gene A deve estar presente. Esse alelo, no DNA, possui a informação, codificada na sequência e bases nucleotídicas, que produz uma enzima que transforma o substrato 1 em 2. Assim como, no outro gene B, o alelo dominante também deve estar presente para haver a transformação do substrato 2 em 3. O substrato 3 é o que produz a cor púrpura. Em resumo: Caso um desses alelos não esteja presente no indivíduo à cor será branca, porque haverá bloqueio na rota metabólica, conforme esquema abaixo: 2 Unidade 1 Genética Molecular Se for considerada uma planta, em princípio, pode-se dizer que todas as suas células possuem o mesmo conteúdo genético, portanto o mesmo número de cromossomos e genes. Esta informação é válida, porém várias alterações cromossômicas são passíveis de acontecer. Por exemplo, as células dos vasos condutores não possuem mais núcleos e as células das folhas podem ter mais de 2 genomas, entretanto quando se refere às reprodutoras, aí sim elas possuem o mesmo número cromossômico (são haploides), salvo algum problema provocado pro mutagênicos Mas como pode cada geração possuir a mesma informação genética contida no DNA? 3. Constituição do DNA O DNA é constituído pelo açúcar (desoxirribose), o fósforo (H3PO4) e bases nitrogenadas (Púricas – Adenina e Guanina e Pirimídicas – Citosina e Timina). O açúcar e o fósforo constituem o que se chama de corrimão e as bases nitrogenadas ligadas entre si, duas a duas, os degraus de uma escada imaginária enrolada de forma helicoidal. A molécula de DNA possui filamento duplo. 3.1. As Ligações no DNA As ligações entre a molécula de fósforo e o açúcar são do tipo fosfodiéster. O fósforo (-PO4) liga-se ao carbono 5 do açúcar de um nucleotídeo e ao carbono 3 do nucleotídeo subsequente. Portanto, ao longo de um dos filamentos do DNA, a ligação é 5’ >>> 3’. Sendo o DNA uma molécula dupla o outro filamento possui a ligação, entre o fósforo e o açúcar, na sequência 3’ >>> 5’. Diz-se então que os filamentos são Antiparalelos. Para manter ambos os filamentos unidos os degraus da escala, que são as bases nitrogenadas, estão ligadas entre si por pontes de hidrogênio. As bases nitrogenadas possuem uma ordenação específica de ligação. A Adenina liga-se com 2 pontos de hidrogênio a Timina e a Citosina com 3 pontes a Guanina. Esse pareamento é constante, apenas as quantidades se alteram. Toda molécula de DNA, num organismo, encerra a informação para o desenvolvimento desse mesmo organismo. A cor do hipocótilo, a posição e a pigmentação das folhas, das flores, o tamanho das vagens ou das espigas, o peso das sementes, a produtividade são características determinadas pelos genes que estão no DNA. E, além disso, possui também a informação para a produção de enzimas que irão desdobrar os substratos no interior das células, para que essas características possam se manifestar. Portanto, a molécula de DNA tem o que se denomina de gene. Se o gene está presente à característica que ele determina aparecerá. Se a sua forma alélica estiver presente no genótipo, outra característica se manifestará, dependendo do tipo de interação que estiver envolvido esse gene. Uma das propriedades funcionais do DNA é a sua duplicação. Essa propriedade permite que uma cópia do DNA já existente na célula sirva de molde para que outra seja formada. Esse processo de duplicação do DNA ocorre numa fase do ciclo celular vegetal chamado de interfase. 4. A Duplicação do DNA As pesquisas sobre o comportamento do DNA foram elaboradas inicialmente em bactérias, principalmente em Echerichia coli, mas devido ao comportamento celular dos eucariontes serem semelhantes, inferiu-se o modelo de conformação e de duplicação do DNA para todos os organismos. A própria conformação da estrutura da molécula de DNA pressupõe sua duplicação, segundo seus descobridores. Para entender como e porque o DNA se duplica é necessário dividir-se o ciclo de vida de uma célula em duas partes: a interfase e a divisão celular, conforme esquema abaixo: 3 Unidade 1 Genética Molecular É na interfase que os genes se expressam, pois ocorre a diferenciação celular. O período de interfase pode ser subdividido em três subperíodos: G1, S e G2. Ambos subperíodos, G1 e G2, derivam da primeira letra da palavra gap, que, em inglês significa parada. No período G1 são produzidas enzimas para o crescimento e diferenciação celular, e enzimas que atuarão sobre o DNA no subperíodo seguinte. Neste estágio o DNA recebe o nome de cromatina. Ela está desespiralizada permitindo a expressão fenotípica do gene, através de outra macromolécula denominada RNA. No período S (síntese) é onde ocorre a duplicação de toda molécula do DNA. Enzimas específicas já produzidas agem sobre o DNA fazendo sua duplicação. Esse processo, como não poderia deixar de ser, é de forma ordenada. Nas extremidades e ao longo do DNA, ao mesmo tempo, proteínas começam a agir desenrolando os filamentos, são as chamadas DNAtopoisomerases. A DNA-helicase provavelmente quebre as pontes de hidrogênio no local de origem da duplicação. Com o afrouxamento dos fios de DNA a principal enzima de duplicação pode agir. É a DNA polimerase. 4.1. A Duplicação do DNA é semiconservativa A DNA polimerase coloca novos nucleotídeos apenas diante de um molde de DNA. Portanto um dos filamentos dos novos DNA’s será velho e outro será novo, por isso a denominação semiconservativa. Para a DNA-polimerase iniciar sua atividade necessita de uma extremidade livre 3’OH, que é gerada por uma enzima chamada primase. Essa enzima é responsável pela colocação de um primer (pequeno segmento de RNA ou de DNA, também chamado de “disparador”) no filamento cuja polaridade é 5’ >>> 3’. Esse primer é colocado na extremidade 3’ da cadeia molde. A partir daí a DNA-polimerase sintetiza novos nucleotídeos. Esse primer posteriormente é retirado pelo processo enzimático de correção, feito pela própria DNA-polimerase. A DNA-polimerase só funciona diante de um molde, cuja polaridade é 3’ >>> 5’. Se os fios do DNA são antiparalelos como agiria a DNA polimerase no molde 5’ >>> 3’? Vários modelos de duplicação foram propostas pelos pesquisadores moleculares. O modelo “faca” e o “descontínuo” foram os primeiros, entretanto o modelo descontínuo ganhou maiores evidências (GARDNER, 1975). O modelo descontínuo prevê que o filamento original da polaridade 3’ >>> 5’ seja duplicado continuamente (filamento leading) e o filamento 5’ >>> 3’ de forma descontínua (filamento leaging). Para isso, conforme já descrito a primase sintetiza um primer no local de origem da duplicação, junto ao filamento 5’ >>> 3’ e a partir daí a DNA polimerase sintetiza o novo filamento dirigindo-se para o lado oposto da origem da duplicação. Esse modelo tem se mantido até então, desde que R. Okazaki o concebeu. Os fragmentos no fio leaging formados receberam o nome do descobridor – Fragmentos de Okazaki. Após a duplicação, formando o fragmento de Okazaki e a retirada da molécula de primer pela ação de correção da DNA polimerase, a enzima ligase promove a ligação dos fragmentos, completando toda a duplicação. Além dessa importante ação enzimática sobre o DNA para sua duplicação, no período S da interfase, salienta-se que no final de todo o processo a quantidade de DNA fica duplicada. Portanto, essas moléculas agora duplicadas, possuem a mesma informação genética. E quando ocorre a condensação para a divisão igualitária dos genes entre as células, os cromossomos se formam já com as cromátides-irmãs. Pode-se afirmar, então, que as cromátides-irmãs, dos cromossomos homólogos são produzidas neste subperíodo. Essas 4 Unidade 1 Genética Molecular cromátides-irmãs dividir-se-ão nas fases de anáfase e anáfase II, da mitose e meiose, respectivamente, levando para as gerações seguintes à mesma informação. (Ver Unidade 2). Essa geração pode ser celular, no mesmo tecido, no mesmo indivíduo, por exemplo, tecido meristemático, como geração populacional. Essa descrição referiu-se a primeira funcionalidade da molécula de DNA. A segunda funcionalidade é a transferência da informação do gene para formação das proteínas. 5. O Processo de Expressão Fenotípica O DNA pode ser copiado em novo DNA, como processo acima descrito, ou ser copiado para uma nova macromolécula chamada RNA (ácido ribonucleico). Se se entendesse o gene como uma conta, o DNA pode ser entendido como um “colar de contas”. Os genes desta forma estariam dispostos linearmente ao longo de todo DNA. Hoje se sabe que nem todos os genes se transformam em proteínas para originarem fenótipos. Há genes nos eucariontes que não funcionam. Esses já funcionaram durante o processo de evolução da espécie ou poderão funcionar, permitindo sua readaptação a ambientes modificados, como tem acontecido nos últimos tempos. Há no genoma sequências de genes não repetidas e sequências altamente repetitivas decorridas de processo de duplicação ao longo da evolução. A maioria dos genes estruturais está nas sequências não repetitivas produzindo as proteínas. Em ervilhas, cerca de 15% do DNA é constituído de cópias únicas ou com pouca repetição (MANTELL et al, 1994). Esses autores citam que o DNA altamente repetitivo forma a heterocromatina nos centrômeros dos cromossomos. Ao longo do DNA existem genes que controlam genes. São chamados de controladores ou reguladores. Esses genes produzem proteínas que se ligam ou se desligam do DNA permitindo a produção ou não de proteínas pelos genes estruturais. Os genes estruturais são os que realmente produzem enzimas que entrarão nas rotas metabólicas para a transformação de substratos e, por consequência, a caracterização do fenótipo. Por isso pode-se afirmar que esses são os genes que se transformam em fenótipos. Entretanto, para a manifestação do fenótipo, outras estruturas são necessárias. São os RNA’s. Três RNA’s são os mais salientados para que os genes estruturais possam funcionar, o RNA mensageiro, o RNA ribossômico e o RNA transportador. Todos esses RNA’s são copiados do DNA pelo processo enzimático, entretanto cada um tem forma e função especifica. Os RNA’s ribossômicos (RNAr) são produzidos na região organizadora do nucléolo (RON) e se constitui na maior quantidade de RNA celular. Originam os ribossomos através do enrolamento da fita de RNA e pode ser encontrado, a nível celular, no reticulo endoplasmático formando o reticulo endoplasmático rugoso. Possuem a função de reunir o RNAm e o RNAt e os aminoácidos no processo de tradução. Os RNA’s transportadores (RNAt) são estruturas mais simples de RNA e possuem a forma de trevo. Sua função é carregar os aminoácidos livres no citoplasma para os ribossomos. O RNAt possui o que se denomina de anticódon. São três bases ribonucleotídicas numa das extremidades da molécula que tem estreita relação com o aminoácido que será carregado numa das alças do trevo. O RNA mensageiro (RNAm) é de forma linear e é um transcrito de uma das fitas do DNA, ou mais especificamente, do(s) gene(s) estrutural(is). Por um processo semelhante o da duplicação do DNA, o RNA é produzido sendo mediado pela enzima RNA polimerase DNA dependente e esse processo chama-se transcrição. 5.1. A Transcrição 5 Unidade 1 Genética Molecular A transcrição é o processo de copiar o DNA para o RNA. Isto é um processo normal na célula, porque o RNA é uma molécula pequena, em relação ao DNA, e, portanto, pode-se locomover através dos poros da carioteca, indo do núcleo para o citoplasma, mais especificamente, para o retículo endoplasmático rugoso. Na transcrição pode-se dizer que os genes são “escolhidos” para serem transcritos, dependendo do órgão em que estiverem e do estágio de desenvolvimento do vegetal. Genes da raiz não se manifestarão nas folhas e vice-versa, dada à especificidade do tecido vegetal. No ponto onde o gene ou grupo de genes se encontram os filamentos do DNA se afrouxam e a RNA polimerase liga-se ao sítio promotor. Esse sítio permite a síntese, porém, quando o gene não deve ser transcrito o sítio promotor não permitirá a ação a RNA polimerase. Essas regiões, ditas promotoras, possuem sequências de bases constante em todos os organismos, apresentando somente pequenas variações e são chamadas de TATA box porque são ricas em adenina e timina (TATAATG em bactérias e TATAAAT em eucariontes). Elas podem ser chamadas, respectivamente de Pribnow box e Hogness box, lembrando os pesquisadores que as encontraram. As regiões promotoras encontram-se sempre antes do trecho que será copiado do DNA para o RNAm, é nesse local que a RNA polimerase se liga. O local dessa região é variável; pode estar de 5 a 10 bases antes da região codificante, para alguns autores. Outros citam, no caso da zeína no milho, estar à região promotora, cerca de 20 a 30 bases antes do gene estrutural (MANTELL et al, 1994). Outra sequência também conhecida que participa do controle da síntese de proteínas é a região chamada CATA box. Está localizada, no caso da zeína, no milho, cerca de 70 a 80 bases antes do local da síntese, conforme abaixo. Elemento de Controle GCCCAATCT Região Promotora TATAAAA -70 Gene Estrutural TACTGCGATCGAAATTTCCCTATATG A partir desse sítio a RNA polimerase inicia o processo de transcrição da fita de DNA copiando-a de forma complementar, apenas com duas alterações: uma nas bases, a base nitrogenada que irá parear com a adenina será a uracila e a outra é no açúcar, que é uma ribose. Dessa forma simples o RNA mensageiro vai se formando, até que a RNA polimerase encontre o ponto de término da transcrição. A direção dessa síntese é da extremidade 5’ >>> 3’ da molécula de DNA. Guilfoyle e Malcolm em 1980 (citado por MANTELL et al, 1994) isolaram a enzima RNA polimerase em embriões de soja, enquanto Jendrirak (1980), citado pelos mesmos autores, a isolou em trigo. Isto foi à confirmação da analogia do que ocorre entre os processos de transcrição em organismos diferentes, no caso a soja e o trigo. Deve-se aqui por uma ressalva: nos eucariontes o RNA produzido da forma acima descrita recebe, atualmente, o nome de pré-RNA, pois ele contém partes que irão se transformar em proteínas e partes que não fazem parte das proteínas, naquele momento. Após a produção do pré-RNA algumas transformações devem ocorrer para que ele possa atravessar a carioteca e ir até os ribossomos. Essas modificações são necessárias porque grande parte dos genes eucariontes é interrompida. Entretanto, a RNA polimerase copia todo segmento, indiscriminadamente, do DNA para formar o pré-RNA. As transformações posteriores são para que passe somente cheguem ao citoplasma, partindo do núcleo, as informações na forma de bases nucleotídicas que codificarão a proteína. As estruturas no pré-RNA que se transformarão em proteínas se chamam exons e segmentos que não são traduzidos que se denominam de introns. (Figura 1.1) 6 Unidade 1 Genética Molecular Figura 1.1 – Formas de produção de RNAm a partir de diferentes exons que ocorre em diferentes tecidos vegetais (Alternative splicing) (Fonte: http://pandasthumb.org/imagens/altsplice.jpg) 5.2. Transformações no pré-RNAm Quatro etapas são importantes para a transformação do pré-RNA em RNAm: a) Retirada dos introns - Os introns não deverão passar para o citoplasma, porque não irão ser traduzidos em proteínas. Isto é o que se denomina de economia celular. b) Ligação dos exons – Os exons ligados originarão a sequência correta de nucleotídeos que é a informação para originar a cadeia polipeptídica. c) A adição do CAP – Uma molécula de 7-metil-guanosina é adicionada na extremidade 5’ do préRNA com a finalidade de direcionar o RNAm até os ribossomos. d) A adição da cauda de Poli A – Várias sequências de adenina são adicionadas na extremidade 3’. Após essas transformações, que ocorrem ainda dentro do núcleo, o RNAm está pronto para ir até o retículo endoplasmático e iniciar o processo de tradução. Nem todos os organismos têm como material genético, o DNA de filamento duplo. Há vírus que possui moléculas de RNA como material genético. Um exemplo é o TMV, vírus do mosaico do tomate, que é um retrovírus. Antes desse vírus infectar a planta ocorre a produção do DNA a partir do seu RNA usando a enzima chamada transcriptase reserva. A partir daí fixa-se sobre a folha e injeta seu DNA no interior da célula hospedeira, que possui DNA normal de filamento duplo e após a célula passa a trabalhar com as informações genéticas injetadas pelo vírus. 5.3. A Tradução O processo de tradução é o de transformar a informação que o RNAm tem em proteína. Para isso os três elementos, RNAm, RNAt e RNAr se encontram formando um só conjunto (Figura 1.2). (A) (B) 7 Unidade 1 Genética Molecular (C) (D) Figura 1.2 – Representação da síntese de proteínas (tradução), onde (a) é o inicio da síntese com a reunião dos RNA’s e o primeiro códon que é AUG – Metionina; (b) alongamento da cadeia de proteínas; (c) continuação do alongamento e (d) termino da síntese com a entrada do códon de fim. (Fonte: Snustad, D.P.; Simmons, M.J. p.294-299, 2001) O RNAr já fixado no retículo endoplasmático possui dois sítios: o sítio A, também chamado de anterior ou amino-acil e o sítio P, posterior ou peptidil e o sítio E que é o de saída do RNAm. A entrada do RNAm se dá no sítio A, que é reconhecido pelo CAP na extremidade 5’. O primeiro códon do RNAm (conjunto de três nucleotídeos) é exposto no sítio A. Neste instante o RNAt, livre no citoplasma, e que possui o anticódon, é ativado para encontrar o aminoácido correspondente a informação do códon. A ativação do aminoácido específico se dá através da enzima aminoacil RNAt sintetase e demanda uma reação com ATP. O resultado é um aminoácido adenilado com energia para fixar-se ao RNAt. Quando essa reação ocorre há liberação de energia e o RNAt está carregado com o aminoácido. Esse é levado até o ribossomo. Há então o pareamento do códon com o anticódon no sítio A. A partir de agora a fita do RNAm anda dentro do ribossomo. Com esse movimento o par códon-anticódon passa do sítio A para o sítio P e novo códon é exposto no sítio A para que outro RNAt seja ativado e traga outro aminoácido. Quando ambos os sítios, A e P, estão ocupados com as duplas códons-anticódons ocorre ligação entre os resíduos de aminoácidos. Com o deslocamento, mais uma vez, ocorre a liberação do RNAt do sítio P e o do sítio A passa para o P. Dois resíduos de aminoácidos já estão ligados entre si. Com a ativação de proteínas de elongação, chamadas fatores e elongação (EF), a cadeia de proteínas vai se formando, pois os aminoácidos vão sendo colocados conforme a informação ditada pelo códon exposto no sítio A do ribossomo. O processo continua sequencialmente até encontrar o ponto final. O ponto final é caracterizado por três códons. São eles: UAA, UAG e UGA. Esses códons vêm na porção 3’, antes da cauda de Poli A e determina o desligamento do RNAm do RNAr. O que permanece é a proteína formada com sua sequência primária de aminoácidos e já com suas outras estruturas, secundária, terciária e quaternária, definidas. Essa correlação existente entre códons no RNAm e aminoácidos na proteína, permitiram o estabelecimento de um código genético. 6. O Código Genético Depois das descobertas que: (1) o DNA é o material genético; (2) que o RNAm é uma cópia do DNA e o intermediário entre a informação genética e a proteína e (3) que a estrutura primária da proteína está em 8 Unidade 1 Genética Molecular acordo com a informação constante no DNA, ficou estabelecido um código, com pequenas variações entre os organismos e que resume todo o processo de tradução. Os itens a seguir demonstram o código genético: 1) Há colinearidade entre genes e proteínas – O RNAm entra nos ribossomos na forma de sequência de códons – 3 bases ribonucleotídicas juntas – que determinam a ativação enzimática do RNAt respectivo e do aminoácido específico. Se há, por exemplo, 250 códons existirão 250 aminoácidos na cadeia polipeptídica. 2) O código é em trincas – Com a explicação do item anterior percebe-se que cada três bases ribonucleotídicas no RNAm corresponde a um códon e que nenhuma dessas bases será aproveitada para outro códon, anterior ou posterior. 3) O código é degenerado – Ao se verificar a tabela de códons percebe-se que vários aminoácidos são codificados por mais de um códon, por exemplo, glicina é codificada por GGG, GGC, GGA e GGU. Exceção a esta propriedade tem a metionina que é codificada apenas por AUG e triptofano por UGG, somente. 4) O código é dito “não ambíguo” – Em condições naturais cada códon sintetiza sempre o mesmo resíduo de aminoácido seja qual for à proteína. A ambiguidade pode ser encontrada em sistemas de cultivo de células. Por exemplo, uma linhagem de E. coli sensível ao antibiótico estreptomicina, irá codificar isoleucina, leucina ou serina diante do antibiótico para a sequência UUU. Normalmente ela codifica para fenilalanina (BURNS e BOTTINO, 1989). 5) O código tem ponto inicial – O códon de início das cadeias é o AUG que codifica metionina e está sempre na porção 5’ do RNAm. Parece que a metionina está presente em todas as sínteses, sempre após um ponto final ou no início da cadeia. Se esse aminoácido não tiver função fisiológica na cadeia polipeptídica é retirado enzimaticamente. 6) O código tem ponto final – Na posição 3’ o RNAm traz códons que permitem o desligamento do RNAm do ribossomo e da proteína formada, tudo isso enzimaticamente. Esses códons não possuem transportadores específicos e são constituídos pelas seguintes sequências de ribonucleotídeos: UAA, UAG e UGA. O final da cadeia não tem apenas um desses códons e sim vários, para fornecer ao ribossomo a informação para que as cadeias possam se desligar. 7. A Tabela de códons A tabela de códons abaixo demonstrada é o resultado final do experimento de Marshall Nirenberg e Heinrich Matthaei que se utilizaram da bactéria Escherichia coli em meio de cultura (GRIFFITHIS et al, 2006). Segunda Base Primeira Base G A 1 Início da cadeia G A C U Terceira Base GLICINA ÁC. GLUTÂMICO ALANINA VALINA G GLICINA ÁC. GLUTÂMICO ALANINA VALINA A GLICINA ÁC. ASPÁRTICO ALANINA VALINA C GLICINA ÁC. ASPÁRTICO ALANINA VALINA U ARGININA LISINA TREONINA METIONINA1 G ARGININA LISINA TREONINA ISOLEUCINA A SERINA ASPARAGINA TREONINA ISOLEUCINA C 9 Unidade 1 Genética Molecular C U SERINA ASPARAGINA TREONINA ISOLEUCINA U ARGININA GLUTAMINA PROLINA LEUCINA G ARGININA GLUTAMINA PROLINA LEUCINA A ARGININA HISTIDINA PROLINA LEUCINA C ARGININA HISTIDINA PROLINA LEUCINA U TRIPTOFANO FIM DA CADEIA SERINA LEUCINA G FIM DA CADEIA FIM DA CADEIA SERINA LEUCINA A CISTEÍNA TIROSINA SERINA FENILALANINA C CISTEÍNA TIROSINA SERINA FENILALANINA U 8. A Proteína Depois de formada via processos de transcrição e tradução, derivadas de um ou mais genes, a proteína tem a função de: catálise enzimática, sustentação mecânica, controle do crescimento e diferenciação celular. A catálise enzimática é a expressão fenotípica indireta do gene na qual a proteína formada possui o destino de transformar substratos, aumentar a velocidade das reações quando necessário. Pode-se neste caso citar como exemplo a enzima fosfofrutocinase que catalisa a transformação da frutose 6-fosfato em frutose 1,6-bifosfato, na rota metabólica da glicólise. A ação de sustentação mecânica, devido às proteínas está na presença do colágeno, uma proteína fibrosa presente na pele e ossos dos animais. No controle de crescimento e diferenciação celular, a expressão do gene se dá pelo controle da informação genética que permite a multiplicação das células no processo de mitose e na diferenciação dessas células, para que elas assumam o papel destinado no local onde se encontram. Exemplos dessas proteínas são os hormônios vegetais, tais como, giberelina, auxina, citocinina. A expressão fenotípica direta tem-se como exemplo, os genes Z1; Z2 e Z3 que controlam a produção de isozimas lipoxigenases, responsáveis pela associação de compostos carbonílicos de cadeia curta às proteínas. Os compostos carbonílicos são responsáveis pelo sabor desagradável no grão de soja e seus derivados. A síntese de proteínas de reserva das sementes tem sido estudada extensivamente em muitas plantas cultivadas, com o objetivo de melhorar o valor nutricional através de técnicas de manipulação genética. Nas leguminosas, as proteínas estão nos cotilédones e nas gramíneas no endosperma. Entre as proteínas de reserva das sementes as prolaminas e glutelinas estão nos cereais e em gramíneas selvagens, enquanto que as globulinas e as albuminas são encontradas em dicotiledôneas. Quando as sementes estão em formação, as proteínas de reserva são produzidas ao nível de retículo endoplasmático, sendo posteriormente transportadas para os locais de reserva que são os vacúolos, chamados como corpos proteicos. 9. Regulação da produção de enzimas Viu-se no início deste capítulo que a produção de determinado fenótipo, cor púrpura das flores de feijão, é dependente exclusivo de dois genes de interação epistática. A cor branca evidencia a falta de um alelo dominante. Os fenótipos finais, como resultantes de todo processo molecular, são dependentes dos genes, das interações entre si e deles com o meio ambiente de forma que a cor púrpura só será produzida pela presença dos dois alelos dominantes. Essa situação mostra que as enzimas são reguladas pelo alelo presente no genótipo das plantas. 10 Unidade 1 Genética Molecular Sob esse aspecto e do ponto de vista dos cromossomos, pode-se dizer que todas as células do vegetal possuem todos os genes, porém surge uma questão, como é que ocorre a regulação metabólica desses genes? Quando se analisa uma cenoura, por exemplo, pode-se perceber que no colo a cor verde aparece quando ela fica a descoberta do solo. A luz, portanto é a indutora para que genes responsáveis pela produção de clorofila fiquem ligados e o fenótipo verde apareça. No ápice da cenoura a cor é sempre constante. Neste ponto os genes responsáveis pela produção de clorofila estão desligados ou bloqueados e a cor verde não se manifesta. Outro processo indutivo de regulação gênica pode ser observado quando sementes colocadas no solo começam o processo de germinação. Nesse caso é necessário que moléculas de água penetrem pelo tegumento atingindo o embrião. Entretanto para que o embrião seja nutrido, a giberelina, um hormônio vegetal é ativado e, a partir dele, enzimas são produzidas para a degradação do endosperma. A primeira enzima produzida pela indução da giberelina é a alfa-amilase na camada de aleurona, tornando-se, portanto, a principal hidrolase na germinação das sementes. É uma endoenzima que hidroliza das ligações -(1,4) ao longo dos polímeros de amilose e amilopectina, transformando o amido em açúcares que irão migrar para os pontos de crescimento do embrião. A giberelina, que é produzida nas células do eixo embrionário, difunde-se até o escutelo e a camada de aleurona, onde atua como um ativador primário na cascata de sinais, que culmina com a indução de um fator de transcrição (o GAMyb) e a expressão gênica das enzimas hidrolíticas (UEGUCHI-TANAKA et al., 2000). Em ervilhas altas foi identificado o gene Le(le) que promove o alongamento do caule. O alelo Le codifica uma enzima que hidrolisa a giberelina GA20 para produzir GA1. O alelo recessivo le codifica uma enzima defectiva que tem função diminuída na proporção de 1/20 da normal, deixando as plantas anãs por possuírem menos GA1. A giberelina também atua sobre as proteínas que regulam a divisão celular (CDK’s) – proteínas quinases dependentes de ciclina – em plantas de arroz submersas. Nessas plantas a giberelina ativa o ciclo celular primeiro na transição da fase G1 para a fase S, provocando aumento da atividade mitótica. Os genes CDK’s são então ativados nas fases anteriores da mitose e quando atingem essa fase disparam a divisão celular no meristema intercalar do caule, aumentando o número de células e também possibilitando seu alongamento. Seja pela presença de alelos, seja pela de agentes indutores, o processo de regulação da produção de proteínas ocorre quando os genes controladores permitem. Na falta do agente indutor os genes controladores produzem uma proteína que se une aos genes operadores, impedindo a ação da RNA polimerase. Quando agente indutor estiver presente, esse induz que os genes controladores produzam uma proteína que não mais se liga ao gene operador, liberando a transcrição dos genes estruturais. O mecanismo de liga ou desliga provocado pela presença ou ausência do indutor, segue o modelo bioquímico “chave-fechadura”. Na ausência do indutor, o sítio ativo da enzima liga-se ao gene promotor e bloqueia a transcrição. Esse mesmo sítio se altera pela presença do agente indutor e agora a enzima não mais se liga ao promotor e a transcrição ocorre (Ver apresentação em Power point – Genética Molecular). Esse sistema de regulação gênica segue o modelo “operon” descrito por Griffithis (2006) em procariontes. Nos organismos eucariontes, entre eles as plantas, o mecanismo de regulação gênica é mais complexo com o “silenciamento” ou não dos genes a serem transcritos, de acordo com os estágios de desenvolvimento da planta (GRIFFITHIS et al., 2006). 10. Referências bibliográficas BURNS, G.W.; BOTTINO, P.J. Genética. 6.ed. Rio de Janeiro: Guanabara-Koogan. 1991. p.381. DE ROBERTIS, E.D.P.; DE ROBERTIS Jr., E.M.F. Bases da Biologia Celular e Molecular. 2.ed. Rio de Janeiro: Guanabara-Koogan. 1993. p.307. 11 Unidade 1 Genética Molecular GARDNER, E.J. Genética. 5.ed. Rio de Janeiro: Interamericana. 1977. p.47-79. GARDNER, E.J.; SNUSTAD, D.P. Genética. 7.ed. Rio de Janeiro: Interamericana. 1986. p.497. MANTEL, S.H.; MATHEWS, J.A.; McKEE, R.A. Princípios de Biotecnologia em Plantas. Ribeirão Preto: Sociedade Brasileira de Genética. 1994. p.344. RAMALHO, M.; SANTOS, J.B.; PINTO, C.B. Genética na agropecuária. 2.ed. São Paulo: Editora Globo. 1989. p.19-59. SNUSTAD, D.P.; SIMMONS, M.J. Fundamentos de Genética. 2.ed. Rio de Janeiro: Guanabara-Koogan. 2001. p.756. SUZUKI, D.T.; GRIFFITHS, A.J.F.; MILLER, J.H.; LEWONTIN, R.C. Introdução à Genética. 4.ed. Rio de Janeiro: Guanabara-Koogan. 1992. p.633. UEGUCHI-TANAKA, M.; FUJISAWA, Y.; KOBAYASHI, M. et al. Rice dwarf mutante dl which is defective ini the alpha subunit of the heteromeric G protein, affects gibberellin signal transduction. Procedings Natural Academic Science. USA. v.97. p.11638-11643, 2000. Exercícios 1. Uma célula produz cerca de 4.000 proteínas cada uma com 250 aminoácidos, em média. Calcule o comprimento mínimo que deve ter o DNA desta célula, em número de nucleotídeos. R: 3.000.000 de nucleotídeos. 2. Um filamento simples com as seguintes bases nitrogenadas: ...AAAGTTCC... . Pode-se saber se pertence à classe dos RNA’s ou do DNA? Se for o DNA, qual é o seu filamento complementar? Se formasse um RNAm destes filamentos de que bases seria constituído? R: DNA. Fio complementar – ..TTTCAAGG.. RNAm ..UUUCAAGG.. e ..AAAGUUCC.. 3. Usando a informação da tabela de códons (na página 9), determine quais são os seguintes polipeptídicos formados a partir do RNAm dados: R: MET – PRO – GLU – PRO – AS – GLI – GLI – PF | ..MET – FEN – PRO – SER – TRE – ALA – PF..| ..LIS – TRE – TRI – ARG – TRE – HIS – PF a. Considerando o primeiro filamento apenas, é possível se determinar a polaridade deste RNAm e do DNA? Se sim, quais serão? R: Sim. RNAm 5’ – 3’ | DNA 3’ – 5’ 4. Se uma proteína tiver a seguinte sequência de aminoácidos: a. Quais as sequências de nucleotídeos no DNA, no RNAm e RNAt que correspondem em cada caso (Cite apenas uma possível). R: 1ª sequência: DNA – GCCGTGACTTACTATCACACT / RNAm – CGG CAC UGA b. AUG AUA GUG UGA / RNAt – GCC GUG UAC UAU CAC | 2ª sequência: DNA – TACTAGTTGCATAAGGACATT / RNAm – AUG AUC AAC GUA UUC CUG UAA / RNAt – UAC UAG UUG CAU AAG GAC | 3ª sequência: DNA – TCCAGCAGTACCCCTACCAGG / RNAm – AGG UCG UCA UGG GGA UGG UCC / RNAt – UCC AGC AGU ACC CCU ACC AGG | 4ª sequência: DNA – TACGCGTAAACCATCATCATCTACGTAAGGATC / RNAm – AUG CGC AUU UGG UAG UAG UAG AUG CAU UCC UAG / RNAt – UAC GCG UAA ACC UAC GUA ACC. Determine a polaridade de cada um dos filamentos do DNA que possui a informação genética. R: 1ª sequência: 5’ – 3’ / 2ª sequência: 5’ – 3’ / 3ª sequência: indeterminada / 4ª sequência: 5’ – 3’. 12 Unidade 1 Genética Molecular 5. A distância entre pares de bases no DNA é de 3,4 angstrons. Qual o tamanho do DNA do milho se ele possui 1,36 x 1010pb? E o fumo, já que ele tem 2,18 x 109pb? Dados: 1 angstrons = 10-10m. R: Milho – 4,624 m. Fumo – 0,74m. 6. Em feijão (Phaseolus vulgaris L.) o gene da enzima málica foi codificado e partes dele se encontram abaixo especificado. O fio a ser usado nessa questão é o que tem TIMINA na extremidade 3’. A partir dessa informação: a. Qual o pré-RNAm e o RNAm? R: Pré-RNAm – 5’ AUG AAC UCG CAU GUC AAAU AAGUUG GUACCU UGGAUGCAGUUUUAG 3’ / RNAm – 5’ AUG AAC UCG CAU AAG UUG UGG AUG CAG UUU UAG 3’ b. Quais os aminoácidos que farão parte dessa enzima? R: MET – AS – SER – HIS – LIS – LEU – TRI – MET – GLU – FEN – PF c. Quantos RNAt diferentes serão necessários para essa síntese? R: 9 RNAt 7. Ativações metabólicas são necessárias para que ocorra a síntese de proteínas nos ribossomos. A porção do gene abaixo descrito servirá de molde para a produção de uma proteína especifica que a célula utilizará para a quebra de cadeias de amido no endosperma das sementes. Baseado nisso diga: a. Quais transportadores são ativados. R: RNAt – UAC CCA AUA CCG AUG AAA GCU; b. Quais rotas metabólicas são ativadas para produção dos aminoácidos. R: Ciclo de Krebs (oxalacetato); Glicólise (3-fosfoglicerato); Glicólise (fosfoenolpiruvato); Glicólise (3-fosfoglicerato); c. Qual o destino dessa proteína produzida. R: Glicólise (3-fosfoglicerato); Glicólise (fosfoenolpiruvato); Ciclo de Krebs (-cetoglutarato); germinação das sementes. 8. O gene FLORICAULA (FLO) selvagem controla a formação de flores na espécie Anthirrinum, enquanto que o seu floricaula (flo) impede a formação de flores. De acordo com essa informação dê uma explicação, pelo ponto de vista da transcrição gênica que esclareça a diferença entre os alelos de um mesmo gene. 9. Descreva o processo enzimático da síntese do DNA e da produção de moléculas de RNA relacionando-os com os períodos do ciclo celular. 10. Descreva as funções dos pontos iniciais e do final na síntese de proteínas relacionando-os com as propriedades do código genético. 11. Uma molécula de RNAm possui 236 nucleotídeos de purinas e 325 de pirimidinas, para a formação de um polipeptídio. Quantos aminoácidos poderão ser formados a partir dessas quantidades de nucleotídeos? R: 79 aminoácidos a partir das purinas e 108 a partir das pirimidinas; 12. Para a formação das cadeias de proteínas são necessários aminoácidos correspondente aos códons do RNAm. Foi detectada na célula uma cadeia polipeptídica com a seguinte sequência de aminoácidos: TIROSINA – VALINA – ASPARTATO – HISTIDINA – LISINA. Baseado nessa cadeia de a origem metabólica desses aminoácidos. R: As origens metabólicas de cada um dos aminoácidos são: Glicólise (3-fosfoglicerato); Glicólise (piruvato); Ciclo de Krebs (oxalacetato); Ribose-5-fosfato; Ciclo de Krebs (oxalacetato). 13
Download