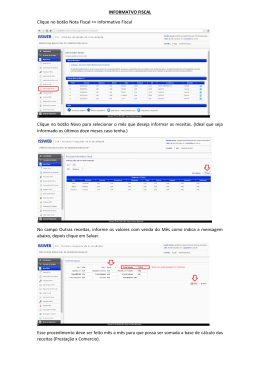
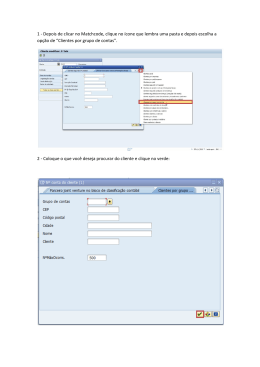
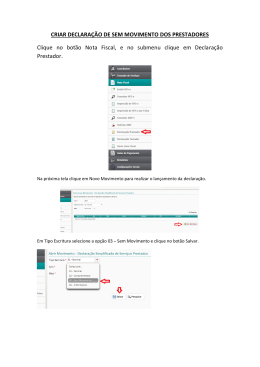
Instruções: Siga estas instruções para analisar os seus dados. Esta pasta contém a documentação e dados originais do seu projeto. Mantenha uma cópia para referências futuras, em caso de dúvida entre em contato. [email protected] 1. Os arquivos *.ab1 ou *.scf são os cromatogramas gerados no ABI 3730 e podem ser vistos no Chromas ou outro software similar. Uma cópia gratuita do Chromas pode ser baixada no seguinte endereço: http://www.technelysium.com.au/chromas_lite.html 2. O arquivo *.cm5 pode ser visto no software Clone Manager 5 ou versão mais nova. Este arquivo tem o mapa completo do seu plasmídeo. 3. Um arquivo de texto tipo gene bank com a sequência do seu plasmídeo pode ser utilizada para reconstruir o mapa do seu plasmídeo com um software da sua preferência se o Clone Manager não for acessível. Nós recomendamos o software gratuito pDRAW32: http://www.acaclone.com/ 4. Para o alinhamento das sequências pode se usar as seguintes ferramentas online: http://www.genone.com.br/sms2/ http://www.ch.embnet.org/software/LALIGN_form.html http://pbil.univ-lyon1.fr/lfasta.php http://www2.igh.cnrs.fr/bin/lalign-guess.cgi 5. O arquivo *.SPF é o contig montado e criado pelo software Sequencher da Gene Codes Corporation. Este arquivo é muito útil para genes maiores que 1kb. Ele pode ser visualizado no Sequencher 4.1.4 ou mais atual. Uma versão demo completamente funcional, sem as funções salvar e exportar, pode ser baixada dos endereços abaixo: • Para Windows: Version 4.7 http://www.genecodes.com/downloads/PC/Sequencher4.7Demo.zip • Para Macintosh OSX: Version 4.7 http://www.genecodes.com/downloads/MAC/Sequencher4.7Demo.dmg 6. Para Visualizar os dados do alinhamento usando o Sequencher siga os passos: 1) Faça o download e instale o Sequencher 2) Abra o arquivo GS*.SPF com um clique duplo nele. 3) Dê um duplo clique no icone Contig[0001] 4) Agora você verá o diagrama do esquema de alinhamento que foi realizado. A orientação de cada sequência é indicada pelas setas. 5) Clique no botão Bases, mova o cursor pelas sequências e selecione as bases clicando nelas. Então clique no botão Show Chromatograms 6) Após a janela Chromatograms se abrir, marque uma posição na Consensus line ao final da janela do Contig (mostrado anteriormente) e todos os cromatogramas correspondentes a regioão serão mostrados. As bases ambiguas estarão marcadas por um ponto abaixo das sequências consenso e as bases correspondentes estarão sublinhadas. 7) Examine as regões de ambiguidade cuidadosamente. Pelo menos uma das sequências do cromatograma deverá ser convincente. www.GenOne.com.br
Baixar