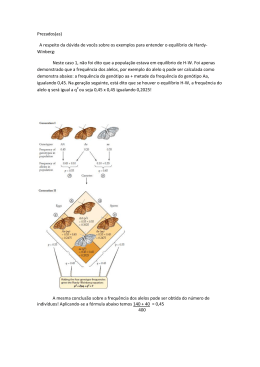
ALINE ANDRADE FREUND DIAGNÓSTICO MOLECULAR DAS ATAXIAS ESPINOCEREBELARES TIPO 1, 2, 3, 6 E 7: ESTUDO POPULACIONAL E EM INDIVÍDUOS COM SUSPEITA CLÍNICA Tese apresentada como requisito à obtenção do título de Doutor em Ciências da Saúde, do Curso de Pós-Graduação em Medicina Interna e Ciências da Saúde, do Setor de Ciências da Saúde da Universidade Federal do Paraná. Orientador: Prof. Lineu César Werneck Curitiba 2007 Livros Grátis http://www.livrosgratis.com.br Milhares de livros grátis para download. ALINE ANDRADE FREUND DIAGNÓSTICO MOLECULAR DAS ATAXIAS ESPINOCEREBELARES TIPO 1, 2, 3, 6 E 7: ESTUDO POPULACIONAL E EM INDIVÍDUOS COM SUSPEITA CLÍNICA Tese apresentada como requisito à obtenção do título de Doutor em Ciências da Saúde, do Curso de Pós-Graduação em Medicina Interna e Ciências da Saúde, do Setor de Ciências da Saúde da Universidade Federal do Paraná. Orientador: Prof. Lineu César Werneck Curitiba 2007 AGRADECIMENTOS O dicionário Michaelis da língua portuguesa explica que gratidão é a qualidade de quem é grato, é agradecimento e reconhecimento. Segundo o “Pequeno Tratado das Grandes Virtudes” de André Comte-Sponville, “A gratidão é a mais agradável das virtudes; não é, no entanto, a mais fácil. A gratidão é um mistério, não pelo prazer que temos com ela, mas pelo obstáculo que com ela vencemos. É a mais agradável das virtudes, e o mais virtuoso dos prazeres.” Assim sendo é com imensa alegria e com profundo reconhecimento que agradeço ao orientador Dr. Lineu César Werneck pela confiança. Foi com quem aprendi que a ciência vai além da curiosidade. Agradeço à CAPES, CNPq e Fundação Araucária, instituições que permitiram a viabilidade prática tão bem como a possibilidade de dedicar-me a este trabalho. Agradeço ao Dr. Hélio Teive e a sua equipe de residentes que contribuíram com a amostra de pacientes. Agradeço à Dra. Rosana Hermínia Scola e ao Dr. Paulo Lorenzoni que clarearam algumas idéias auxiliando em alguns momentos difíceis. Agradeço o apoio de minhas queridas amigas do laboratório Raquel, Eunice e Nyvia pelos chás acompanhados de divertidas conversas indispensáveis para preencher o coração! Agradeço à Dra. Elvira que além da amizade, alegria e do norte profissional, foi alguém que nunca me deixou esmorecer e que me mostrou, com seu exemplo, um caminho nobre para ser humano! Agradeço às minhas grandes amigas pessoais Lupe Furtado e Magda Clara pelo carinho de sempre e pelas horas infindáveis filosofando sobre a vida e a ciência. Agradeço aos meus pais pelo amor e pela vida! Ao meu marido pela compreensão, companhia, amor mas principalmente pelo bom-humor com o qual sempre soube conduzir as nossas vidas trazendo leveza para os momentos mais impossíveis! Muito obrigada! Enfim, a Deus por tudo! SUMÁRIO LISTA DE FIGURAS ................................................................................................................................. II LISTA DE TABELAS ...............................................................................................................................III LISTA DE ABREVIATURAS...................................................................................................................IV RESUMO ...................................................................................................................................................VI ABSTRACT ............................................................................................................................................. VII I. INTRODUÇÃO ........................................................................................................................................8 1.1. Ataxias hereditárias..............................................................................................................9 1.2. Ataxias cerebelares autossômicas dominantes (ACAD) ............................................................10 1.2.2. Sinais e sintomas clínicos presentes nas AEC subtipos 1, 2, 3, 6 e 7 ................................................. 15 1.2.3. Gene e proteína envolvidos nas AEC subtipos 1, 2, 3, 6 e 7 .............................................................. 17 1.2.4. Tratamento atual e perspectivas sobre patogênese e tratamento......................................................... 21 1.3. Importância do estudo em população assintomática .................................................................25 1.4. Princípio de Hardy-Weinberg....................................................................................................26 II. OBJETIVOS .....................................................................................................................................30 2.1. Objetivo principal ......................................................................................................................31 2.2. Objetivos secundários:...............................................................................................................31 III. MATERIAIS E MÉTODOS ........................................................................................................32 3.1 População e amostra ..................................................................................................................33 3.2. Protocolos..................................................................................................................................35 3.2.1. Extração de DNA ............................................................................................................................... 35 3.2.2. Amplificação dos genes de interesse .................................................................................................. 36 3.2.3 Detecção do produto de amplificação.................................................................................................. 39 3.3. Análise de resultados .................................................................................................................40 IV. RESULTADOS...........................................................................................................................42 4.1. Produtos de amplificação ..........................................................................................................43 4.2. Definição dos alelos...................................................................................................................44 4.3. Freqüência alélica .....................................................................................................................49 4.4. Equilíbrio de Hardy-Weinberg e heterozigosidade ...................................................................52 4.5. Análise Clínica...........................................................................................................................56 V. DISCUSSÃO ....................................................................................................................................62 5.1. Considerações sobre a metodologia de análise de fragmento em eletroforese de capilar ........63 5.2. Importância do estudo populacional .........................................................................................67 5.3. Metodologia para diagnóstico molecular ..................................................................................73 VI. CONCLUSÕES....................................................................................................................................77 VII. REFERÊNCIAS BIBLIOGRÁFICAS................................................................................................79 VIII. APÊNDICES .....................................................................................................................................97 Apêndice 1: Instrumento de coleta de dados para grupo controle ...................................................98 Apêndice 2: Instrumento de coleta de dados para pacientes............................................................99 Apêndice 3: Termo de consentimento informado livre e esclarecido - Pacientes...........................100 Apêndice 4: Termo de consentimento informado livre e esclarecido – Grupo Controle................103 Apêndice 5: Comitê de ética e de pesquisa em seres humanos.......................................................106 Apêndice 6: Representação gráfica da amostra de pacientes ........................................................108 Apêndice 7: Lista de reagentes .......................................................................................................109 Apêndice 8: Preparo de soluções....................................................................................................110 Apêndice 9: Características clínicas da amostra de 56 pacientes analisados................................111 I LISTA DE FIGURAS FIGURA 1: DOENÇAS COM REPETIÇÕES EXPANDIDAS ..................................................................................18 FIGURA 2: QUADRO DE PUNNET PARA EQUILÍBRIO DE HARDY-WEINBERG. ................................................28 FIGURA 3: LOCALIZAÇÃO DE ANELAMENTO DOS INICIADORES NO LOCUS ATXN 3. .....................................38 FIGURA 4: LOCALIZAÇÃO DE ANELAMENTO DOS INICIADORES NO LOCUS ATXN 7. .....................................39 FIGURA 5: AMPLIFICAÇÃO DOS ALELOS DE TAMANHO NORMAL PARA OS GENES ATXN1, ATXN2, ATXN3, ATXN6 E ATXN7.............................................................................................................................43 FIGURA 6: AMPLIFICAÇÃO DOS ALELOS ANORMALMENTE EXPANDIDOS OBSERVADOS NA AMOSTRA DE PACIENTES PARA ATAXIA ESPINOCEREBELAR 2, 3, 6 E 7....................................................................44 II LISTA DE TABELAS TABELA 1: GENÉTICA MOLECULAR DAS ATAXIAS CEREBELARES AUTOSSÔMICAS DOMINANTES .................13 TABELA 2: LIMITE DE REPETIÇÕES DAS PRINCIPAIS ATAXIAS CEREBELARES ...............................................15 TABELA 3: CÁLCULO DAS REPETIÇÕES CAG EM CADA LOCUS ....................................................................41 TABELA 4: MÉDIA (± DP) E VARIÂNCIA DOS ALELOS EM PARES DE BASE (PB) ENTRE CONTROLES E PACIENTES ANALISADOS PARA ATAXIA ESPINOCEREBELAR 1............................................................45 TABELA 5: MÉDIA (± DP) E VARIÂNCIA DOS ALELOS EM PARES DE BASE (PB) ENTRE CONTROLES E PACIENTES ANALISADOS PARA ATAXIA ESPINOCEREBELAR 2.............................................................46 TABELA 6: MÉDIA (± DP) E VARIÂNCIA DOS ALELOS EM PARES DE BASE (PB) ENTRE CONTROLES E PACIENTES ANALISADOS PARA ATAXIA ESPINOCEREBELAR 3.............................................................47 TABELA 7: MÉDIA (± DP) E VARIÂNCIA DOS ALELOS EM PARES DE BASE (PB) ENTRE CONTROLES E PACIENTES ANALISADOS PARA ATAXIA ESPINOCEREBELAR 6.............................................................48 TABELA 8: MÉDIA (± DP) E VARIÂNCIA DOS ALELOS EM PARES DE BASE (PB) ENTRE CONTROLES E PACIENTES ANALISADOS PARA ATAXIA ESPINOCEREBELAR 7.............................................................48 TABELA 9: MÉDIA (±DP) EM PB DE CINCO REPETIÇÕES DE CORRIDA ELETROFORÉTICA EM CINCO AMOSTRAS DIFERENTES PARA OS CINCO LOCI ......................................................................................................50 TABELA 10: FREQÜÊNCIA ALÉLICA % (± EP%) NOS CINCO LOCI ANALISADOS PARA ATAXIA ESPINOCEREBELAR 1, 2, 3, 6 E 7 ENTRE CONTROLES E PACIENTES......................................................51 TABELA 11: FREQÜÊNCIA DAS DOENÇAS NOS 115 INDIVÍDUOS COM SUSPEITA CLÍNICA ..............................52 TABELA 12: NÚMEROS DE INDIVÍDUOS OBSERVADOS E ESPERADOS PARA OS POSSÍVEIS GENÓTIPOS DO GENE ATXN 1 NA AMOSTRA DO GRUPO CONTROLE. ....................................................................................53 TABELA 13: NÚMEROS DE INDIVÍDUOS OBSERVADOS E ESPERADOS PARA OS POSSÍVEIS GENÓTIPOS DO GENE ATXN 2 NA AMOSTRA DO GRUPO CONTROLE. ....................................................................................54 TABELA 14: NÚMEROS DE INDIVÍDUOS OBSERVADOS E ESPERADOS PARA OS POSSÍVEIS GENÓTIPOS DO GENE ATXN 3 NA AMOSTRA DO GRUPO CONTROLE. ....................................................................................54 TABELA 15: NÚMEROS DE INDIVÍDUOS OBSERVADOS E ESPERADOS PARA OS POSSÍVEIS GENÓTIPOS DO GENE CACNA 1 NA AMOSTRA DO GRUPO CONTROLE. .................................................................................55 TABELA 16: NÚMEROS DE INDIVÍDUOS OBSERVADOS E ESPERADOS PARA OS POSSÍVEIS GENÓTIPOS DO GENE ATXN 7 NA AMOSTRA DO GRUPO CONTROLE. ....................................................................................55 TABELA 17: PROBABILIDADE OBTIDA APÓS APLICAÇÃO DO TESTE DO QUI QUADRADO (χ2) NO QUAL A HIPÓTESE NULA É QUE NÃO HÁ DIFERENÇA SIGNIFICATIVA ENTRE AS FREQÜÊNCIAS GENOTÍPICAS OBSERVADAS E ESPERADAS NOS CINCO LOCI ANALISADOS (EQUILÍBRIO DE HARDY-WEINBERG)......56 TABELA 18: HETEROZIGOSIDADE OBTIDA NOS CINCO LOCI ANALISADOS. ...................................................56 TABELA 19: DADOS GERAIS DE INDIVÍDUOS COM AS CARACTERÍSTICAS CLÍNICAS DA AMOSTRA DOS 56 PACIENTES ANALISADOS ....................................................................................................................58 TABELA 20: COMPARAÇÃO ENTRE DIAGNÓSTICO MOLECULAR E CLÍNICO DOS 115 PACIENTES PARA AEC 1, AEC 2, AEC 3, AEC 6 E AEC 7 ........................................................................................................60 TABELA 21: NÚMERO DE PACIENTES COM A RESPECTIVA SUSPEITA CLÍNICA E DIAGNÓSTICO MOLECULAR DA AMOSTRA TOTAL DE 115 PACIENTES..................................................................................................61 TABELA 22: TESTE “T” PARA AMOSTRAS INDEPENDENTES COMPARANDO A MÉDIA DOS ALELOS DE TODOS OS 115 PACIENTES E DA POPULAÇÃO ASSINTOMÁTICA (CONTROLES). ...............................................61 TABELA 23: FREQÜÊNCIAS DE ATAXIA ESPINOCEREBELAR DESCRITAS NA POPULAÇÃO BRASILEIRA...........72 III LISTA DE ABREVIATURAS A= Adenina ACAD= Ataxia Cerebelar Autossômica Dominante AEC= Ataxia Espinocerebelar AXH= Alanina- Aminoácido não epecificado-Histidina ATXN= ataxin (ataxina) C= Citosina CAG= Citosina-Adenina-Guanina CCD= Charge-Coupled Device (Dispositivo de Carga Acoplado) CEP= Comitê de Ética e Pesquisa em Seres Humanos DH= Doença de Huntington DM= Distrofia miotônica DMJ= Doença de Machado - Joseph DNA= Desoxiribonucleic acid (Ácido desoxirribonucléico) dNTPs = nucleotídeos (A, T, C, G) DP= Desvio Padrão DRPLA= Dentatorubropallidoluysian Atrophy (Atrofia dentatorubropalidoluisana) EA= Episodic Ataxia (Ataxia Episódica) EDTA =Ácido etilenodiamino tetracético EP= Erro padrão EPM= epilepsia mioclônica progressiva FAM= fluorescência azul G= Guanina H= Heterozigosidade HC-UFPR= Hospital de Clínicas da Universidade Federal do Paraná IV HW= Hardy-Weinberg NED=fluorescência amarela OMIM= Online Mendelian Inheritance of Man (Herança Mendeliana Humana online) PAGE= Polyacrylamide gel electrophoresis (Eletroforese em gel de poliacrilamda) pb= pares de base PCR= Polimerase chain reaction (Reação em cadeia da polimerase) Q= aminoácido glutamina RNA= Ribonucleic acid (Ácido ribonucléico) RNAm= RNA mensageiro RNAi= RNA de interferência ROX= fluorescência vermelha SBMA= Spinal and bulbar muscular atrophy (Atrofia muscular bulbo espinal) SDS= Sodium Dodecyl sulfate (N-Dodecil sulfato de sódio) shRNA= small hairpins RNA (pequenos grampos de RNA) SLE= Solução de Lise de Eritrócitos T= Timina TE= Tris - EDTA UTR= Untranslated region (Região não traduzida) VIC=fluorescência verde VPP= Valor preditivo positivo VPN = Valor preditivo negativo Taq= Termophilus acquaticus TRIS= Hidroximetil-aminometano V RESUMO As ataxias espinocerebelares (AEC) são doenças neurodegenerativas nas quais os pacientes apresentam sinais e sintomas semelhantes decorrente do comprometimento do cerebelo e suas conexões. A análise da repetição (CAG)n nos loci de AEC 1, AEC 2, AEC 3, AEC 6 e AEC 7 foi realizada para definir o tamanho e as freqüências dos alelos. Esta análise é importante para compreensão molecular, determinação da variação normal dos alelos na população do Paraná, utilidade diagnóstica e determinação da frequência das doenças. A amostra consiste em 154 doadores de sangue da população do Paraná e 115 pacientes com suspeita clínica de ataxia. Os produtos de PCR foram submetidos à eletroforese em capilar. Em doadores de sangue o tamanho das repetições e os respectivos alelos mais freqüentes foram: AEC 1 de 19 a 36, sendo o alelo 30 mais freqüente (32,29%); AEC 2, de 2 a 28, com o alelo 20 mais freqüente (86,44%); AEC 3, de 12 a 34, e mais freqüentes os alelos 12 e 20 (26,57% e 27,96% respectivamente); AEC 6, de 2 a 13, sendo mais frequente o alelo 8 (39,59%) e AEC 7, de 2 a 10, sendo mais frequente o alelo 4 (70,61%). Nenhum desvio do equilíbrio de Hardy-Weinberg foi detectado. Nos pacientes com ataxia, o tamanho das repetições foram: AEC 1 de 23 a 36 com nenhum alelo expandido acima do normal; AEC 2 de 14 a 56 repetições com 4 alelos expandidos acima do normal; AEC 3 de 12 a 74 repetições com onze alelos expandidos acima do normal; AEC 6 de 2 a 21 repetições com um único alelo expandido acima do normal e AEC 7 de 2 a 50 repetições com dois alelos expandidos em níveis anormais. A ataxia mais freqüente entre os pacientes foi a AEC 3 (21,74%), seguido AEC 2 (5,22%), AEC 7 (2,61%), AEC 6 (0,87%) e nenhum caso de AEC 1. A sensibilidade da metodologia foi 62,22 %, a especificidade 90,00 %, o valor preditivo positivo (VPP) 80,00 % e o valor preditivo negativo (VPN) foi 78,75 %. Os dados estatísticos auxiliaram na definição dos alelos e freqüências, demonstrando reprodutibilidade e confiabilidade metodológica. A análise genética da população assegurou a qualidade técnica além de demonstrar estabilidade alélica na população não afetada. A metodologia utilizada no estudo atual é útil em discriminar clinicamente indivíduos normais e afetados para o diagnóstico molecular, distinguindo geneticamente os cinco subtipos de ataxia. VI ABSTRACT The spinocerebellar ataxia (SCA) are neurodegenerative illnesses which the patients present similar signals and symptoms due to the compromising of the cerebellum and its connections. The trinucleotide (CAG)n repeats was done at the SCA 1, SCA 2, SCA 3, SCA 6 and SCA 7 to identify allele size ranges and frequencies. This analysis is important for molecular understanding, determination of the normal range of the alelles in normal population of Paraná, for diagnostic purpose and determination of disease frequency. The sample consisted of 154 blood donors from Southern Brazil (Paraná) and 115 patients with a clinical suspicion of ataxia. PCR products were submitted to capillary electrophoresis. In the blood donors, the size ranges and most frequent alleles for the five loci were: SCA 1, 19 to 36 bp and allele 30 is the most frequent (32.29%); SCA 2, 6 to 28 bp and the most frequent is allele 20 (84.44%); SCA 3, 12 to 34 bp and allele 12 and 20 are the most frequent (26.57% e 27.96% respectively); SCA 6, 2 to 13 bp and the most frequent is allele 8 (39.50%) and SCA 7, 2 to 10 bp and the most frequent is allele 4 (70.61%). No deviations from HW equilibrium were detected. In the patients, the size ranges for the five loci were: SCA 1, 23 to 36 bp with no expanded alleles above normal population; SCA 2, 14 to 56 bp with four expanded alleles higher than normal controls; SCA 3, 12 to 74 bp with 11 expanded alleles above normal population; SCA 6, 2 to 21 bp with one expanded allele above normal population and SCA 7, 2 to 50 bp with two expanded alleles at abnormal levels. The most frequent ataxia among patients is SCA 3 (21.74%) followed by SCA 2 (5.22%), SCA 7 (2.61%) e SCA 6 (0.87%) and no case of SCA 1. The sensitivity of the methodology was 62.22 %, specificity 90.00 %, predictive positive value (VPP) 80.00% and the predictive negative value (VPN) was 78.75%. The statistical data was useful for defining alleles and their frequencies, showing reprodutibility and methodological confidence. Population genetic analysis ensured the technical quality of the assay and demonstrated allelic stability in the population not affected. The methodology used in the present study can be usefull to distinguish between clinically normal individuals and affected individuals by molecular diagnosis at the five loci analyzed. VII I. INTRODUÇÃO 8 Ataxia na terminologia de origem grega significa “fora de controle”. O termo médico “ataxia” refere-se a distúrbios no controle da postura do corpo, na coordenação motora, no controle da fala e nos movimentos oculares. O termo ataxia locomotora tem sido empregado desde o século XIX, significando mais comumente incoordenação motora [HARDING, 1983]. As ataxias cerebelares são ocasionadas pelo comprometimento do cerebelo e suas conexões. Este comprometimento pode ser atribuído a causas primárias no caso das ataxias congênitas e hereditárias; ou a causas secundárias em ataxias decorrentes de doenças como a esclerose múltipla, tumores, doença vascular encefálica, drogas, infecções, além de síndrome paraneoplásicas, doenças endócrinas e doenças autoimunes [HARDING, 1983]. Em 1863, Nicolaus Friedreich descreveu o primeiro tipo de ataxia primária hereditária com sintomas clínicos de ataxia, cifoescoliose e pé cavo. A partir de então outros autores passaram a descrever diversos sintomas distintos de ataxia, surgindo então as primeiras classificações das doenças cerebelares [FRIEDREICH, 1863 apud ALBANO, 2000]. 1.1.Ataxias hereditárias As ataxias hereditárias são doenças neurodegenerativas nas quais os pacientes apresentam clinicamente ataxia, sintomas inespecíficos e em muitos casos um padrão de herança específico. Estas ataxias são separadas em ataxias hereditárias de início precoce e de início tardio. São consideradas ataxias hereditárias de início precoce aquelas que se iniciam antes dos 20 anos (ex: ataxia de Friedreich). Entre estas encontram-se também as ataxias associadas a distúrbios metabólicos e a defeitos de reparação do DNA (ex: ataxia telangiectásica) e as ataxias congênitas [HARDING, 1983]. 9 A grande heterogeneidade clínica e também anátomo-patológica dos diferentes tipos de ataxias hereditárias [KLOCKGETHER e cols., 2000 apud TEIVE 2005] diagnóstico baseado somente nos achados clínicos [SCHÖLS e cols., 2004]. torna difícil o Atualmente, o uso das técnicas moleculares permite diagnosticar e detectar vários loci causadores de ataxias hereditárias. Dessa forma, é possível a utilização de uma classificação baseada no padrão de herança (autossômica recessiva, autossômica dominante, ligada ao X e mitocondrial), no defeito genético, no mecanismo de desenvolvimento da doença (patofisiologia e patologia), e nas manifestações clínicas e no prognóstico [PILCH e cols., 2002 apud TEIVE, 2005; PULST, 2003 apud TEIVE, 2005]. 1.2. Ataxias cerebelares autossômicas dominantes (ACAD) As ACAD também são conhecidas como ataxias espinocerebelares (AEC) e tem uma incidência de cerca de 1 a 3 casos para cada 100.000 habitantes [SCHÖLS e cols., 2004]. Os pacientes com AEC apresentam os primeiros sintomas geralmente na fase adulta. Em uma mesma família podem existir afetados nas diferentes gerações. Normalmente indivíduos afetados são heterozigotos para o gene. A mais comum das AEC é a doença de Machado-Joseph (DMJ), também chamada de ataxia autossômica dominante tipo 3, ou ainda ataxia espinocerebelar tipo 3 (AEC 3). A DMJ é encontrada nos principais estudos epidemiológicos mundiais, sendo até o presente momento a forma mais comum de AEC encontrada no Brasil [JARDIM e cols., 2001; LOPES-CENDES e cols., 1997; SILVEIRA e cols. 1996; TEIVE e cols., 2001]. A DMJ recebe esse nome porque originalmente foi descrita na família Machado, descendentes de emigrantes portugueses açorianos radicados em Massachussets, e na família Joseph, descendentes de José Bastiana, originário dos Açores [NAKANO e cols., 1972 apud TEIVE, 2005; ROSENBERG e cols., 1976 apud TEIVE, 2005; WOODS e SHAUMBURG, 1972; apud TEIVE, 2005]. 10 A característica das mutações mais freqüentes que desencadeiam AEC é o aumento do número de repetições de uma determinada seqüência de nucleotídeos acima do encontrado em pessoas sem histórico da doença. O aumento de repetições do trinucletídeo CAG é a causa mais freqüente de AEC (Tabela 1). As expansões dinâmicas, como são conhecidas essas mutações, decorrem da instabilidade do DNA repetido num mesmo locus, ocasionando maior propensão a deslizes da enzima DNA polimerase. Estes deslizes acarretam a inserção de mais nucleotídeos nesse segmento, favorecendo a expansão. Assim, a ocorrência da primeira mutação aumenta a chance de novos deslizes nesses sítios, levando a mais expansões e mais deslizes [RICHARDS e cols., 1992]. Acredita-se que a base biológica das repetições na expansão seja a instabilidade das repetições durante a divisão celular tanto na meiose quanto na mitose [DEKA e cols., 1999; TAKIYAMA e cols., 1995; TAKIYAMA e cols., 1999]. Esta instabilidade gera, pelo processo de contração e conversão gênica, diferentes populações celulares com diferentes números de repetições formando um mosaico de células. A instabilidade na meiose ocasiona o mosaicismo gonadal observado no esperma de pacientes. Durante a transmissão estes pacientes apresentam tendência ao aumento do tamanho da expansão através das gerações. Este mosaicismo quando ocorre durante a mitose, é detectado em células somáticas (mosaicismo somático) incluindo linfócitos, células musculares e sistema nervoso central, porém é menos acentuado que o mosaicismo gonadal [KAMEYA e cols., 1995; MACIEL e cols., 1997; STEVANIN e cols., 2000]. Vários autores descrevem que na maior parte dessas doenças causadas por aumento no número de repetições, há diferenças quanto à gravidade das manifestações clínicas e a idade de início dos sintomas dependendo de qual genitor a alelo expandido é herdado. Entre as AEC, quando a transmissão é paterna, verifica-se uma tendência ao aumento no numero de repetições nos filhos, ao contrário de quando o alelo expandido é 11 transmitido pela mãe, no qual essa tendência não é observada. Portanto, esta transmissão paterna implica em manifestações clínicas mais graves nos filhos que muitas vezes podem apresentar os primeiros sintomas de ataxia anterior aos pais. Normalmente, quanto mais grave e quanto mais cedo se dá o início dos sintomas, maior é o número das repetições. Esta observação é conhecida como antecipação de sintomas e é comum nas doenças de expansão [FRASER, 1998 apud ALBANO, 2000]. 1.2.1. Distribuição geográfica das AEC A prevalência das ataxias hereditárias é variável ao redor do mundo. Em Cuba, a prevalência de AEC 2 é a mais elevada do mundo sendo em média de 43 casos por 100.000 habitantes [VELÁZQUEZ-PÉREZ e cols., 2001] e em Portugal, no arquipélago dos Açores, a DMJ apresenta prevalência de 1 para 2402 (10/01/2008). [LIMA e cols. 1997 em http://www.aaadmj.com/doenca.php] No Brasil, nos estados de Minas Gerais, São Paulo, Goiás e do Espírito Santo, foram encontradas 33 famílias com AEC. Dentro dessas famílias existem 426 pacientes afetados, com prevalência média de 6,55 casos por 100.000 habitantes apud TEIVE, 2005]. [CASSA e cols., 1996 No estado do Rio Grande do Sul foram avaliados 66 casos de AEC sendo a proporção de casos de doença de Machado Joseph (DMJ) mais alta em relação às outras AEC estimando-se como 1,8/100.000 e 0,2/100.000 respectivamente, sugerindo um efeito fundador açoriano [JARDIM e cols., 2001; SILVEIRA e cols., 1996]. Observa-se uma correlação entre origem étnica e a prevalência de determinada mutação que causa AEC. A prevalência das diferentes ataxias variam conforme o país e as características étnicas. Dessa forma, a freqüência alélica das expansões também é característica para cada população [BRUSCO e cols., 2004; DRYER e cols., 2003; LIMA e cols., 2005; MARUYAMA e cols., 2002; MOSELEY e cols., 1998; SALEEM e cols., 2000; SCHÖLS e cols., 1997; SCHÖLS e cols., 2004; SHIMIZU e cols., 2004; SILVEIRA e cols., 2002; STOREY e cols., 2000; TANG e cols., 2000; TAKANO e cols., 1998; VAN DE WARRENBURG e cols., 2002; ZORTEA e cols., 2004]. Dentro das AEC, as mais freqüentes ao redor do 12 mundo são os subtipos de AEC 1 (6%), AEC 2 (15%), AEC 3/DMJ (21%), AEC 6 (15%) e AEC 7 (5%) [BIRD e cols., 2007 em http://www.geneclinics.org/profiles/ataxias/details.html#diffdx] (10/01/08). Tabela 1: Genética molecular das ataxias cerebelares autossômicas dominantes Doença Locus Gene/ Proteína AEC 1 6p23 ATXN1/ataxina -1 AEC 2 12q23-24.1 ATXN2/ataxina -2 MJD/ AEC 3 14q21 ATXN3/ataxina - 3 AEC 4 16q24 Desconhecida AEC 5 11p11-q11 AEC 6 19p13 CACNA1/ Canal de Cálcio AEC 7 3p21.2-12 ATXN7/Ataxina -7 AEC 8 13p21 KLHL1AS/? SPTBN2/ Cadeia β da espectrina do cérebro AEC 10 22q13 ATXN10/Ataxina - 10 AEC 11 15q14-21.3 Desconhecida PPP2R2B/ Proteína Serina/treonina Mutação Ref. Bibliográfica CAG/resíduo poliglutamina RICH e cols., 1987 CAG/resíduo poliglutamina GISPERT e cols., 1993 CAG/resíduo poliglutamina KAWAGUCHI e cols., 1994 Desconhecida FLANIGAN e cols., 1996 Desconhecida IKEDA e cols., 2006 CAG/resíduo poliglutamina ISHIKAWA e cols., 1997 CAG/resíduo poliglutamina CTG/3´UTR BENOMAR e cols., 1994 VINCENT e cols., 2000 Expansão ATTCT / íntron 9 MATSUURA e cols., 2000 Desconhecida WORTH e cols., 1999 CAG/ não traduzida HOLMES e cols., 1999 FUJIGASAKI e cols. 2001 Desconhecida HERMAN-BERT e cols., 2000 WATERS e cols. 2006 AEC 12 5q31-33 AEC 13 19q13.3-13.4 KCNC3/ Canal de potássio AEC 14 19q13.4 PRKCG/ proteína quinase C (γ) AEC 15 3 p24.2 Desconhecida Desconhecida KNIGHT e cols., 2003 AEC 16 3p26.2 CNTN4/ Contactina- 4 Desconhecida MIURA e cols. 2006 fosfatase 2A, (subunidade β) Mutação de ponto no exon 4 YAMASHITA e cols., 2000 CAG/resíduo AEC 17 6q27 TBP/TFIID AEC 19 1p21-q21 SCA19/? Desconhecida VERBEEK e cols. 2002, SCHELHAAS e cols. 2004 AEC 20 11 centromérico SCA20/? Desconhecida KNIGHT e cols. 2004 AEC 21 7p21-15 SCA21/? Desconhecida VUILLAUME e cols. 2002 Desconhecida VAN SWIETEN e cols. 2003 AEC 27 13q34 DRPLA 12p13.31 EA 1 12p13 EA 2 19p13 FGF14/Fator de crescimento de fibroblasto 14 poliglutamina DRPLA/ Proteína relacionada a CAG/resíduo atrofina-1 poliglutamina KCNA1/Canal de potássio subfamília A membro 1 CACNA1A/Canalde cálcio tipo P/Q subunidade α 1A NAKAMURA e cols. 2001 NAGAFUCHI e cols., 1994 Mutação de ponto LITT e cols., 1994 Mutação de ponto DIRIONG e cols. 1995 OPHOFF, e cols. 1996 AEC-Ataxia espinocerebelar SCA - spinocerebellar ataxia; EA - episodic ataxia; DRPLA Dentatorubral-Pallidoluysian Atrophy Retirado do site: [BIRD e cols., 2007 em http://www.geneclinics.org/profiles/ataxias/details.html#diffdx] (10/01/08). 13 A análise das mutações em uma amostra da população brasileira mostra o perfil das mutações mais freqüentes, caracterizando a população, que apresenta um alto índice de miscigenação. Assim sendo, no Brasil, um estudo realizado por LOPES-CENDES em 1997, mostrou a freqüência das mutações que causam AEC 1, AEC 2, MJD/AEC 3 e DRPLA em 328 pacientes brasileiros de diferentes regiões do país (Rio Grande do Sul, Santa Catarina, Paraná, São Paulo, Minas Gerais, Rio de Janeiro e Bahia). A freqüência de AEC 1 foi 3%, AEC 2 em 6%, DMJ/AEC 3 foi 30% e nenhuma mutação foi encontrada para DRPLA. Existe um limite de repetições CAG característico de cada uma das AEC para pessoas normais e para indivíduos que apresentam a doença. Além disso, encontram-se alelos intermediários em estudos com famílias. A presença desses grupos distintos de alelos evidencia a importância da definição da faixa de repetições CAG para indivíduos normais e pacientes. Indivíduos com alelos intermediários podem não manifestar clinicamente a doença enquanto outros indivíduos com o mesmo alelo intermediário podem apresentar sinais e sintomas, podendo ser considerados alelos com penetrância reduzida [ANDREW e cols., 1997]. Esta faixa de repetição CAG também pode variar de população para população. Na doença de Huntington (DH), doença que apresenta o mesmo tipo de mutação, e com maior incidência nas populações mundiais sendo mais bem caracterizada que as ataxias, os alelos intermediários são definidos segundo três critérios: 1- Número de repetições CAG responsáveis em gerar novas mutações (premutação); 2- Número de repetições CAG acima do encontrado em indivíduos normais, porém, com número de repetição menor que o encontrado em pacientes; 14 3- Encontrados em indivíduos assintomáticos [GOLDBERG e cols., 1993 apud ANDREW e cols., 1997] Através de dados encontrados na literatura foi possível definir uma faixa entre alelos de tamanho normal, intermediário e expandido, útil para o diagnóstico molecular (Tabela 2). Tabela 2: Limite de repetições das principais ataxias cerebelares Doença Repetições Normal Intermediário Expandido Ref. Bibliográfica AEC 1 CAG 6-39 36-41 39-81 MATILLA e cols., 1993 ZHULKE e cols., 2002 AEC 2 CAG 14-31 32-34 34-57 CANCEL e col.s.,1997 DMJ CAG 13-41 46-56 62-84 GIUNTI e cols., 1995; TAKIYAMA e cols., 1995 VAN ALFEN e cols., 2001 AEC 6 CAG 4-16 19-20 21-27 ZUCHENKO e cols., 1996 AEC 7 CAG 4-17 28-36 37->300 STEVANIN e cols., 1998 1.2.2. Sinais e sintomas clínicos presentes nas AEC subtipos 1, 2, 3, 6 e 7 Na AEC tipo 1 frequentemente o início é após a idade de 20 anos, manifestando-se com ataxia de marcha, disartria, nistagmo, fraqueza muscular, hiperreflexia e oftalmoparesia. Ocorre piora progressiva da ataxia e lentidão dos movimentos sacádicos oculares. Podem estar associado os sintomas de paralisia bulbar e distonia [BANFI ecols., 1994; ARRUDA e TEIVE, 1997]. Histologicamente há perda neuronal e gliose do núcleo denteado, da ponte, do pedúnculo cerebelar médio e das olivas [KAMEYA e cols., 1995]. A AEC tipo 2 é caracterizada por apresentar ataxia cerebelar associada a disartria, tremores, hipo/arreflexia profunda dos membros superiores e também nos membros inferiores (definindo a presença de neuropatia periférica associada), fasciculações de face e de membros e pela presença de movimentos oculares sacádicos lentos [ARRUDA e TEIVE, 1997]. Neuropatológicamente a AEC do tipo 2 é caracterizada por 15 apresentar atrofia cerebelar, com perda das células de Purkinje e das células granulares, perda de neurônios olivares, da substância negra e das células do corno anterior da medula espinhal [PULST e cols., 2003 apud TEIVE, 2005]. A AEC tipo 3 (DMJ) foi originalmente descrita apresentando sintomas de degeneração nigro-espino-denteada, com oftalmoplegia nuclear e degeneração estriatonigral [NAKANO e cols.., 1972 apud TEIVE, 2005 ; ROSENBERG e cols.,1976 apud TEIVE, 2005; WOODS e SHAUMBURG, 1972 apud TEIVE, 2005]. Os principais sintomas são ataxia cerebelar, oftalmoplegia progressiva externa associada a graus variados de sinais piramidais, sinais extrapiramidais e neuropatia periférica. Também podem ser encontrados ataxia cerebelar, associada ao parkinsonismo, distúrbios da motilidade ocular extrínseca, fasciculações musculares, perda dos reflexos profundos, nistagmo e respostas cutâneoplantares extensoras. Os exames neuro-patológicos demonstraram perda neuronal e gliose na substância negra, núcleos pontinos, núcleo vestibular, colunas de Clarke e cornos anteriores da medula espinhal [COUTINHO e ANDRADE, 1978; COUTINHO e cols., 1982; PAULSON e SUBRAMONY, 2003 apud TEIVE, 2005]. A AEC tipo 6 caracteriza-se por apresentar ataxia cerebelar “pura”, podendo estar associada a disartria, nistagmo e perda de sensibilidade profunda. Muitos pacientes apresentam episódios vertiginosos intensos, precedendo o início da ataxia. Em outros casos, paralelamente ao quadro de ataxia cerebelar lentamente progressivo, existem episódios intermitentes de ataxia (que correspondem a ataxia episódica tipo 2). De uma forma geral, a AEC 6 apresenta evolução lentamente progressiva, com início do quadro clínico ao redor dos 50 anos de idade [PULST, 2003 apud TEIVE, 2005]. Estudos de neuroimagem revelam atrofia cerebelar e o exame anátomo-patológico demonstra perda das células de Purkinje do córtex cerebelar e também gliose do complexo olivar inferior. [ISHIKAWA e cols., 2001 apud TEIVE, 2005]. 16 Na forma de AEC tipo 7 encontra-se a associação de ataxia cerebelar com déficit visual progressivo, decorrente de degeneração da retina (distrofia macular). Podem estar associados sinais piramidais, oftalmoplegia, parkinsonismo e particularmente movimentos sacádicos lentos. Os sintomas podem ter seu início na infância ou até os 60 anos de idade, com uma progressão muito mais rápida quando o início é mais precoce. Do ponto de vista neuropatológico existe degeneração olivopontocerebelar associada a perda de células ganglionares da retina [ANDREW e cols., 1997; STEVANIN e cols., 2000] 1.2.3. Gene e proteína envolvidos nas AEC subtipos 1, 2, 3, 6 e 7 A expansão da repetição no DNA implica em mecanismos diferentes de patogenicidade dependendo do local no gene onde essas expansões se encontram, se esta região será traduzida em proteína ou não, qual a seqüência, qual o gene, qual a função (Figura 1). A determinação da base molecular de doenças de repetição CAG pode permitir delinear a base de eventos moleculares que causam a doença. Estes eventos parecem ser característicos de cada região [PAULSON e SUBRAMONY, 2002] Os subtipos de AEC de maior interesse no presente trabalho são os de maior incidência nas populações. Assim, foram selecionadas as ataxias espinocerebelares (com seus respectivos números no Online Mendelian Inheritance in Man - OMIM) dos subtipos 1 (OMIM 164400), 2 (OMIM 183090), 3 (OMIM 109150), 6 (OMIM 183086) e 7 (OMIM 164500) que apresentam como mutação o aumento do número de repetições CAG em loci distintos. Esta região é traduzida em um resíduo de poliglutamina maior do que o esperado, gerando uma proteína anômala capaz de formar agregados que muitas vezes irão se localizar no núcleo da célula. Estas inclusões têm sido ultimamente correlacionadas com a sobrevivência neuronal [SOONG e PAULSON, 2007] e não com o início da patogênese das doenças neurodegenerativas com expansão CAG como se acreditava 17 anteriormente [ZOGHBI e ORR, 2000]. As inclusões parecem refletir uma resposta protetora no neurônio na qual os agregados protéicos apresentam uma estrutura inerte [SOONG e PAULSON, 2007]. Figura 1: Doenças com repetições expandidas - O mecanismo molecular da doença é determinado pela repetição na seqüência, tamanho e posição, qual gene e qual função. As seqüências repetidas para cada doença estão mostradas nos parênteses. As ataxias estão sublinhadas. [Adaptado de PAULSON e SUBRAMONY, 2002] O gene ATXN1 apresenta 450 kb do DNA genômico e está organizado em 9 exons. Os primeiros sete exons fazem parte da região 5’ não traduzida, enquanto que os dois últimos contém a região codificadora de 7.277 pb e a região 3’não traduzida. Os primeiros quatro exons não codificadores apresentam sítios para processamento alternativo em vários tecidos [BANFI e cols., 1996]. A proteína ataxina 1 está presente no cérebro e em vários outros tecidos não neurais. Nos tecidos não neurais, a proteína encontra-se basicamente no citoplasma enquanto nos neurônios a proteína está presente no núcleo. Nas células de Purkinje do cerebelo, a ataxina 1 encontra-se predominantemente no núcleo [SERVADIO e cols., 1995]. A ataxina 1 apresenta um domínio AXH (Alanina – aminoácido não especificado Histidina) dimérico com estrutura β pregueada semelhante a outras proteínas com sítios 18 de ligação ao RNA. Este domínio AXH parece também ter interações distintas com outras proteínas, apresentando um processo peculiar e ainda desconhecido dentro da célula [CHEN e cols., 2004]. Em 2005, IRWIN e cols. demonstraram que a ataxina 1 normal tem papel no processamento do RNA, provavelmente auxiliando o transporte desta molécula para o exterior do núcleo. Estudos com regulação da transcrição em cultivo celular e em Drosófilas evidenciaram que a proteína mutada inibe a transcrição através da diminuição de função do domínio AXH envolvido na modulação do processo transcricional (repressor transcricional) 2002; TSUDA e cols., 2005]. [DE CHIARA e cols., 2005; LAM e cols., 2006; OKAZAWA e cols., Além disso, o resíduo de poliglutamina aumentado ocasiona um agregado de proteínas dentro da célula diminuindo a degradação pela via normal (via ubiquitina-proteasoma) [VERHOEF e cols., 2002]. Outro achado importante é que o aumento de número de repetições CAG na ataxina 1 diminui a atividade do proteasomo o que estaria diretamente ligado a degeneração celular encontrada na AEC 1 [PARK e cols., 2005]. O gene ATXN 2 contém 25 exons e abrange aproximadamente 130 kb. A repetição CAG encontra-se no exon 1 [SAHBA e cols. 1998]. A ataxina 2 é uma proteína de 1.313 aminoácidos (150 KDa) que normalmente é expressa em todas as células do organismo. Encontra-se em concentração elevada nas células de Purkinje do cerebelo e substância negra. Níveis de ataxina 2 aumentam com a idade e na AEC 2. A localização celular de ataxina 2 parece estar no Complexo de Golgi. Quando há aumento do resíduo de poliglutamina na ataxina 2, o funcionamento do complexo de Golgi fica prejudicado e encontram-se microagregados citoplasmáticos de proteína. Verifica-se perda de células de Purkinje devido à morte celular levando a deficiência funcional e anatômica [HUYNH e cols., 2003]. Estudos mais recentes revelam que a proteína ataxina 2 apresenta interação com a proteína citoplasmática ligadora de poli A e também apresenta domínio para interação com RNA mensageiro estando associada a 19 ribossomos [RALSER e cols., 2005]. Dessa forma a ataxina 2 mutada poderia estar intervindo de maneira indireta ou direta, respectivamente, na tradução de alguns RNAs mensageiros em particular [STTATERFIELD e PALLANK, 2006]. O gene ATXN 3 apresenta 48.240 pb com 11 exons [ICHIKAWA e cols., 2001]. A proteína ataxina 3 encontra-se dispersa no citoplasma. O aumento do resíduo de poliglutamina leva a formação de um agregado [IKEDA e cols., 1996; SISODIA e cols., 1998]. A ataxina 3 mutada adota uma conformação característica capaz de ligar-se a matriz nuclear expondo o resíduo de glutamina [PEREZ e cols., 1999]. Assim como na ataxina 1, a correlação com a via ubiquitina-proteasoma também foi observada por WARRICK e cols. em 2005 sendo uma proteína com repetições CAG normal, supressora da neurodegeneração. A ataxina 3 é uma proteína que apresenta um domínio denominado Josefina caracterizado por uma hélice flexível em forma de grampo onde se localiza o sítio ativo. O domínio Josefina possui atividade proteolítica por interagir com cadeias de poliubiquitinas. Esta descoberta associada à modificação pós traducional pela ubiquitinação da sua própria molécula e conseqüente degradação pela via proteasoma, faz um elo entre as proteínas com resíduo de poliglutaminas e vias dependentes de ubiquitina como possível patogênese da doença [BERKE e cols., 2005; NICASTRO e cols., 2005; NICASTRO e cols., 2006]. O gene da proteína do canal de cálcio (CACNA1) apresenta 300 kb com 47 exons. O seqüenciamento de todos os exons e adjacências mostrou grande variação polimórfica incluindo a repetição CAG na região 3’ não traduzida [OPHOFF e cols., 1996]. Esta variabilidade gênica pode resultar em subunidades distintas que formam o poro do canal devido a possíveis processamentos alternativos do RNA mensageiro afetando a estabilidade do mesmo, bem como a funcionalidade do canal 2004]. [JURKAT-ROTT e LEHMANN, Quando há um resíduo de poliglutamina expandido esta proteína acumula-se no 20 citoplasma celular, principalmente das células de Purkinje. Esta inclusão não está ubiquitinilada ao contrário da maioria dos agregados observados nas outras ataxias [ISHIKAWA e cols., 1999]. O gene ATXN7 apresenta 140 Kb e 13 exons [MICHALIK e cols., 1999]. A ataxina 7 faz parte da família de proteínas acetiltransferase de histonas e apresenta uma localização nuclear [McMAHON e cols., 2005]. A proteína mutante apresenta expansão no resíduo de poliglutamina o que ocasiona mudança na sua conformação. A inclusão nuclear contém a proteína anômala, ubiquitina, proteínas de “heat-shock” e subunidades do proteasoma [LEBRE e cols., 2001; LATOUCHE e cols., 2007]. 1.2.4. Tratamento atual e perspectivas sobre patogênese e tratamento Não há até o momento tratamentos definidos para as diferentes formas de AEC existindo, entretanto, inúmeros relatos de testes terapêuticos, com medicações colinérgicas, serotoninérgicas, gabaminérgicas e também dopaminérgicas. Algumas formas raras de AEC episódica podem responder ao uso de acetazolamida [KLOCKGETHER, 2000 apud TEIVE, 2005]. Alguns pacientes apresentam nível reduzido da coenzima Q10 (CoQ10) nos tecidos musculares. A CoQ10, ou ubiquinona, é uma substância cuja função celular é de receptor de elétrons dos complexos I e II da cadeia respiratória da mitocôndria. É, portanto, um agente antioxidante fundamental nas células que sofrem estresse oxidativo como no caso de doenças neurodegenerativas. Estes pacientes podem se beneficiar com a suplementação de CoQ10 apesar dessa reposta terapêutica ser controversa. Além disso, a CoQ10 não evita a neurodegeneração, porém o declínio funcional torna-se mais lento [BAKER e TARNOPOLSKI, 2003; LITTARRU e TIANO, 2005; MONTERO e cols., 2007; SHULTS, 2003] A grande questão que se faz atualmente é como as proteínas anômalas que apresentam esse resíduo de poliglutamina expandido estão envolvidas na etiologia da doença. A relação exata entre o dobramento protéico, o processo de agregação e a 21 formação de inclusões macroscópicas permanece obscura. Acredita-se que as interações protéicas no início da agregação das proteínas anômalas com resíduo de poliglutamina (poli Q) parecem ser fundamentais para desencadear a toxicidade celular. Existem dois processos de interação inicial: 1- pequenos oligômeros da proteína mutante interagindo com proteínas celulares e também com outras proteínas poli Q promovendo a toxicidade; 2- grandes complexos fibrilares com as proteínas com tratos poli Q expandidos formando agregados relativamente neutros a célula [BEHRENDS e cols.; 2006; BULONE e cols., 2006; NAGAI e cols., 2007]. Outra questão ainda sem resposta é exatamente porque as proteínas com tratos expandidos de poli Q são tóxicas ao neurônio. Vários pesquisadores têm descoberto que as proteínas poli Q desempenham suas funções em importantes vias metabólicas tais como interação com fatores transcricionais, modulação da tradução protéica e controle do dobramento e degradação protéica através da via ubiquitina-proteasomo [BERKE e cols., 2005; EVERT e cols., 2000; LA SPADA e TAYLOR, 2003; LATOUCHE e cols., 2007; LEBRE e cols., 2001; MICHALIK e BROECKHOVEN, 2003; NICASTRO e cols., 2005; NICASTRO e cols., 2006; PARK e cols., 2005. VERHOEF e cols., 2002]. Portanto alterações dessas funções como no caso das proteínas com expansão, leva a um desequilíbrio em vias que mantêm o controle de qualidade da célula. Assim diferentes proteínas celulares passam a ser mal elaboradas desencadeando um efeito crônico e possivelmente deletério para o neurônio. Três vias parecem estar envolvidas nas doenças de expansão: chaperoninas moleculares, ubiquitina-proteasomo e autofagia [SOONG e PAULSON, 2007]. As chaperoninas moleculares parecem suprimir a toxicidade dos resíduos poli Q expandidos participando da síntese de proteínas nascentes e prevenindo a agregação e toxicidade das mesmas [BEHRENDS e cols., 2006] 22 A via normal de ubiquitina-proteasomo é a mais importante via de regulação da atividade protéica. A degradação protéica precisa ocorrer no momento exato e em resposta a eventos celulares. A ubiquitina é uma molécula protéica pequena (76 aminoácidos) e o proteasoma é um complexo cilíndrico capaz de reconhecer e clivar uma proteína ubiquitinilada. Resumidamente, para que uma determinada proteína seja degradada, uma cascata de reações ocorre, levando a poli-ubiquitinilação da mesma. O complexo proteína-ubiquitina é reconhecido pelo proteasoma que libera a ubiquitina para reciclagem e desdobra a proteína para clivagem em pequenos peptídeos e aminoácidos. Além da degradação, a ubiquitinação de uma proteína desencadeia uma série de processos celulares como o reparo do DNA, a formação de endocitos no caso de proteínas de membrana, a tradução e a ativação (ou inibição) da transcrição. Estes processos são respostas bioquímicas da célula para tentar corrigir uma proteína mal elaborada e manter a sua homeostasia, evitando assim a presença de proteínas anômalas e não funcionais. A ineficácia da correção leva a formação de agregados protéicos que evoluem a complexos amilóides [HERSHKO e CIECHANOVER., 1991 apud ALBERTS e cols., 1994; RECHSTEINER e cols., 1992 apud ALBERTS e cols., 1994]. A função do proteasomo diminui com a idade ao mesmo tempo em que há o surgimento dos sintomas de AEC. Dentro do processo da via ubiquitina-proteasomo as proteínas com resíduo de poliglutamina parecem desempenhar alguma função ainda não bem estabelecida [HUNTER e cols 2007; RODRIGUES e cols 2007; RUBINSZTEIN, 2006; TRUANT e cols 2006; VALERA e cols 2007; SOONG e PAULSON, 2007]. A autofagia tem sido estudada em diversas doenças neurodegenerativas relacionadas com a idade e com a presença de acúmulos ubiquitinilados [KOMATSU e cols., 2006]. Achados recentes estão de acordo com o modelo que postula interações aberrantes proteína-proteína. Proteínas poli Q expandidas, fragmentos proteolíticos, componentes transcricionais e proteínas da cromatina que formam complexos 23 oligoméricos depletando certos fatores transcricionais e outras proteínas importantes no núcleo. Este evento acarreta alterações na atividade de promotores, perturba a modificação da cromatina por acetil transferases e leva a um mecanismo patogênico de desregulação transcricional [LAM e clos., 2006; RUBINSZTEIN, 2006; SERRA e cols.2006]. A maior contribuição que todos os estudos podem oferecer é a compreensão da doença objetivando um tratamento preciso. Várias estratégias terapêuticas têm sido buscadas pelos pesquisadores para tratamento das doenças de expansão de poliglutamina. Como descrito previamente, a expressão deste tipo de proteína anômala acarreta desequilíbrio celular através de alterações no metabolismo que provavelmente resultam na neurodegeneração. Cada etapa relacionada com o possível processo de patogênese tem sido alvo para estudos terapêuticos entre eles: supressão da expressão do gene mutado por RNA de interferência; inibição do mau dobramento e agregação da proteína; promoção da degradação protéica; ativação da transcrição; ativação da função mitocondrial; inibição da morte da célula neuronal e neuroproteção por fatores neurotróficos e modelos knockout. A padronização de estudos pré-clinicos e o desenvolvimento de biomarcadores para avaliar a eficácia terapêutica na triagem clínica serão necessárias para o desenvolvimento de drogas úteis nas doenças de expansão. Para tanto, a definição do diagnóstico com auxílio de técnicas moleculares torna-se uma etapa importante para a possível orientação terapêutica [COLOMER GOULD; 2005; LATOUCHE ecols, 2007; SHULMAN e cols. 2003; STEVANIN e cols., 2000; XIA e cols., 2004]. O uso do RNA de interferência (RNAi) surgiu com grande potencial terapêutico nas doenças neurodegenerativas principalmente de transmissão autossômica dominante. Existe uma certa eficácia em usar vetores virais expressando pequenos grampos de RNA (shRNA) contra genes terapêuticamente relevantes em modelos de ratos transgênicos para doenças neurodegenerativas tais como AEC, Esclerose lateral amiotrófica, Doença de Huntington e amiloidose. Muito embora o uso do vetor viral 24 para RNAi apresente limitações principalmente no que diz respeito ao uso em humanos, estudos mais recentes têm desenvolvido um vetor não viral para “entrega” do RNAi como alternativa terapêutica para estas doenças [FARAH, 2007; RODRIGUEZ-LEBRON e GONZALEZ- ALEGRE, 2006]. 1.3. Importância do estudo em população assintomática No caso de doenças de expansão o estudo da variabilidade alélica da repetição de trinucleotideos CAG em população normal mostrou-se bastante controverso. Vários autores verificaram que há heterogeneidade do mecanismo mutacional nos diferentes loci de repetições. Entre eles diferentes genes que apresentam regiões de repetição CAG, com características moleculares e de patogenicidade semelhantes, não necessariamente possuem os mesmos padrões dinâmicos [ANDRES e cols., 2002; BERTRANTPETIT e CALAFELL, 2001 apud LIMA e cols., 2005; COSTA e cols., 2002; DEKA e cols., 1995]. Observa-se que alelos expandidos são mantidos na população humana ao longo das gerações. PRESTES e cols. em 2007 realizaram uma comparação entre o valor adaptativo de pacientes com DMJ com seus parentes não afetados pela doença e com a população normal chegando à conclusão de que pacientes com a DMJ apresentam valor adaptativo maior. A tendência mutacional a favor das expansões sugere que a maioria dos casos de novas mutações decorreria da existência de um número elevado de repetições dentro da normalidade. Estes alelos normais, porém com número maior de repetições, ou ainda a existência de alelos intermediários na população não afetada seriam reservatórios para geração de novos alelos expandidos [RUBINSZTEIN e cols., 1994]. De acordo com isso, vários autores associaram a prevalência de uma doença de expansão de trinucleotídeo em uma população específica, com alelos de tamanhos maiores no respectivo locus ou alelos intermediários na população não afetada como é observado na 25 doença de Huntington (DH) e distrofia miotônica (DM) [COSTA e cols., 2002; DAVIES e cols., 1992; DEKA e cols., 1999; GOLDMAN e cols., 1994; RASKIN e cols. 2000; WATKINS e cols., 1995; ZERYLNICK e cols., 1995]. Entretanto, a capacidade de correlacionar a prevalência de uma determinada doença de expansão com a freqüência de alelos normais de tamanho mais elevado no respectivo gene depende diretamente do perfil epidemiológico dessas doenças. Com o objetivo de compreender melhor a dinâmica no locus da DMJ e trazer maior discernimento sobre o mecanismo molecular de mutação no qual se encontram os alelos expandidos no gene, LIMA e cols. em 2005 estudaram o polimorfismo de alelos normais (selvagens). Os dados encontrados não evidenciaram nenhuma correlação entre a presença dos alelos normais com número de repetições aumentados e a alta incidência da doença além da inexistência de alelos intermediários na população. O estudo da freqüência dos alelos e da distribuição genotípica em uma população pode ser efetuado com o princípio de Hardy-Weinberg. 1.4. Princípio de Hardy-Weinberg O princípio de Hardy-Weinberg foi demonstrado independentemente e simultaneamente, pelo matemático inglês Godfrey Harold Hardy e pelo geneticista alemão Wilhelm Weinberg em 1908. Em genética de população a palavra “população” significa um grupo de indivíduos da mesma espécie vivendo dentro de uma área geográfica restrita o suficiente para que todos os membros possam se acasalar ao acaso (Panmixia). Sempre existirá uma certa “estruturação geográfica” geralmente provocada pelo ambiente (ex.: cidades são separadas por desertos e montanhas). Grandes populações, com características mendelianas e estruturadas geograficamente, são interessantes porque é no interior delas que as freqüências alélicas não variam ao longo das gerações. Independe de um gene ser 26 raro ou freqüente, sua freqüência permanecerá a mesma desde que determinadas condições sejam mantidas. O modelo matemático que define essa hipótese determinando as freqüências genotípicas para crescimento ao acaso da população é o princípio (ou lei ou equilíbrio) de Hardy-Weinberg (HW) [NEEL e SCHULL, 1954 apud BEIGUELMAN, 1990; LI, 1972 apud BEIGUELMAN, 1990; DOBZHANSKY, 1970 apud HARTL, 1998; SPIESS, 1977 apud HARTL, 1998; WRIGHT, 1969 apud HARTL, 1998]. Existem oito premissas que deverão estar presentes em uma determinada população para que o princípio de Hardy-Weinberg possa ser verificado: 1. A população deve ser infinita; 2. Apresentar reprodução sexuada; 3. Os indivíduos acasalam-se ao acaso (panmixia); 4. Os indivíduos devem ser diplóides; 5. Não haver sobreposição de gerações; 6. A taxa de mutações deve ser baixa; 7. As migrações são desprezíveis; 8. A seleção natural não ocorre nos alelos analisados No caso mais simples de um único locus com dois alelos A e a com freqüências de p e q, respectivamente, o princípio de HW prediz que as freqüências genotípicas para o homozigoto AA será p², para o heterozigoto Aa será 2pq e para o homozigoto aa será q² [DOBZHANSKY, 1970 apud HARTL, 1998; SPIESS, 1977 apud HARTL, 1998; WRIGHT, 1969 apud HARTL, 1998] (Figura 2). 27 Fêmeas A (p) a (q) A (p) AA (p²) Aa (pq) a (q) aA (qp) aa (q²) Machos Figura 2: Quadro de Punnet para equilíbrio de Hardy-Weinberg. Adaptado de [HARTL, 1998] A constância nas freqüências alélicas é a conseqüência mais importante do princípio de HW. A constância alélica significa que a ausência de forças evolutivas (deriva genética, fluxo gênico, seleção natural e mutação) manterá as freqüências através das gerações. O processo de transmissão mendeliana mantém as freqüências alélicas constantes e então conserva a distribuição da variabilidade genética da forma como ela é [DOBZHANSKY, 1970 apud HARTL, 1998; SPIESS, 1977 apud HARTL, 1998; WRIGHT, 1969 apud HARTL, 1998]. O estudo da variabilidade genética é útil para reconstituir a evolução de um grupo de organismos. As pesquisas sobre polimorfismos genéticos são úteis na busca de relações genéticas entre sub-populações de uma espécie. A idéia é que sub-populações compartilhem alelos através de migração de modo que alelos comuns podem ser provenientes de um ancestral comum. Além disso, os polimorfismos também são úteis como marcadores genéticos podendo estar ligados a um gene nocivo associado a uma doença ao longo de gerações. Em uma família, com histórico de uma determinada doença presente através de gerações, é possível determinar uma linhagem de marcadores associados que co-segregam com o gene causador da doença [DOBZHANSKY, 1970 apud HARTL, 1998; SPIESS, 1977 apud HARTL, 1998; WRIGHT, 1969 apud HARTL, 1998]. Uma análise de cada gene em população normal é necessária para se ter um conhecimento maior sobre uma possível associação entre a representação biológica da epidemiologia das doenças de expansão e o perfil alélico desta população assintomática. [ABADE e cols., 2000 apud LIMA e cols., 2005; SANTOS e cols., 2000 apud LIMA e cols., 2005] Sendo assim, é de 28 fundamental importância a análise em grupo controle para determinação da freqüência alélica na população, e como uma análise preliminar para fornecer informação sobre possíveis associações moleculares e de origem e manutenção da doença na população. Além disso, também é importante salientar que cada população humana pode apresentar um perfil genético próprio e diferente das demais populações. 29 II. OBJETIVOS 30 2.1. Objetivo principal O presente estudo visa determinar o genótipo das repetições CAG nos genes ATXN 1, 2, 3, 6 e 7 em uma amostra da população normal e de pacientes para utilização no diagnóstico molecular de pacientes com suspeita de ataxia. 2.2. Objetivos secundários: 1. Definir tamanho em pares de base (pb) e desvio padrão de cada alelo; 2. Definir a freqüência alélica nos genes ATXN 1, ATXN 2, ATXN 3, ATXN 6 e ATXN 7 na amostra da população não afetada; 3. Verificar se as distribuições genotípicas estão de acordo com o Equilíbrio de Hardy-Weinberg para interpretações populacionis e metodológicas; 4. Definir a freqüência alélica e detectar alelos expandidos nos genes ATXN 1, ATXN 2, ATXN 3, ATXN 6 e ATXN 7 na amostra de pacientes; 5. Oferecer subsídios para avaliar a diversidade dos alelos na população normal e o processo de evolução molecular para manutenção do gene nesta população comparando com a amostra de pacientes. 31 III. MATERIAIS E MÉTODOS 32 3.1 População e amostra O diagnóstico molecular para AEC 1, AEC 2, AEC 3, AEC 6 e AEC 7 foi realizado em 143 indivíduos com história clínica de ataxia cerebelar do Serviço de Neurologia (Ambulatórios e Internamento) do Hospital de Clínicas da Universidade Federal do Paraná (HC-UFPR). Paralelamente ao estudo dos pacientes foi analisado um grupo controle com 154 indivíduos não aparentados da população normal, obtidos aleatoriamente no Banco de Sangue do HC-UFPR. Os indivíduos que aceitaram participar do grupo controle apresentaram idade acima de 18 anos, não relataram serem portadores ou ter qualquer doença genética, hereditária ou mal formações na família, nem terem sido expostos à radiação ou a agentes tóxicos. Os dados de controles e pacientes foram levantados conforme Instrumento de Coleta de Dados presente no Apêndice (Apêndices 1 e 2, respectivamente). A amostra de doadores de sangue consiste em 90 homens e 64 mulheres não aparentados com idade média de 32,29 ± 10,63 anos. Com esta média de idade, essa amostra pode conter indivíduos que poderão manifestar clinicamente sinais e sintomas de ataxia no futuro. Assim, no momento da coleta foi questionado sobre doenças diagnosticadas na família para posterior análise caso houvesse necessidade. Nenhum caso de indivíduos com ataxia diagnosticada foi informado. A naturalidade de 129 indivíduos desta amostra é paranaense. Os 25 indivíduos restantes residem no Estado do Paraná há mais de 10 anos (um do Acre, um da Bahia, um do Ceará, dois de Minas Gerais, dois do Rio de Janeiro, um de Rondônia, dois do Rio Grande do Sul, dez de Santa Catarina, cinco de São Paulo). Além disso, esta amostra apresenta 72,46% de Euro-Brasileiros, 2,17 % de Afro-Brasileiros, 23,19% de miscigenados e 2,17% de Ameríndios. O termo de consentimento assinado foi obtido de cada indivíduo sendo o 33 projeto aprovado pelo Comitê de Ética em Pesquisas em Seres Humanos do HC – CEP 775.005/2004-01 (Apêndices 3 a 5) Para as amostras de sangue que deram entrada no laboratório cujos pacientes apresentavam suspeita clínica de ataxia, foram levantados alguns dados presentes no prontuário. Estes dados estão especificados no Apêndice 2 do Instrumento de Coleta de Dados. Dessa forma foi possível identificar pacientes e respectivos familiares presentes na amostra de indivíduos com suspeita clínica de AEC. Existem 11 famílias nas quais apenas um indivíduo participou da amostra submetida aos cálculos estatísticos. A reconstituição do heredograma não foi possível por não haver dados disponíveis suficientes. Assim, dos 143 indivíduos, somente 115 não aparentados foram analisados evitando algumas das distorções possíveis no cálculo de freqüência alélica e da freqüência dos diferentes subtipos de ataxia no HC-UFPR (Apêndice 6). Apenas 56 indivíduos dos 115 da amostra tiveram os dados clínicos avaliados pelos prontuários analisados. Isto permitiu averiguar a idade de início dos sintomas, os principais sintomas e quando a transmissão é paterna ou materna nesses indivíduos. Estes 56 pacientes com dados clínicos disponíveis indicam a seguinte suspeita clínica: cinco pacientes para ataxia espinocerebelar do tipo 2; 16 pacientes para DMJ; um para ataxia espinocerebelar tipo 4; três para ataxia espinocerebelar do tipo 6; dois para ataxia espinocerebelar do tipo 7; três com suspeita de ataxia espinocerebelar tipo 10; um paciente com síndrome do neurônio motor associado a síndrome cerebelar; um com polineuropatia periférica motora; um com suspeita de epilepsia mioclônica; um para tremor essencial; dois pacientes com ataxia esporádica; dois ataxia de Freidreich; um para ataxia cerebelar recessiva e 17 pacientes com diagnóstico clínico a esclarecer. Além destes 56 pacientes, existem 59 pacientes sem dados clínicos disponíveis. Estes 59 pacientes apresentam nos registro do laboratório a seguinte suspeita clínica: 34 dois pacientes para ataxia espinocerebelar do tipo 2; 14 pacientes para DMJ; dois para ataxia espinocerebelar do tipo 6; um para ataxia espinocerebelar do tipo 7; um para ataxia espinocerebelar tipo 8; quatro com suspeita de ataxia espinocerebelar tipo 10; um para ataxia congênita e 34 com diagnóstico a esclarecer. 3.2. Protocolos A fórmula e o preparo das soluções, bem como a origem do material utilizado podem ser encontrados na “Lista de reagentes” e “Preparo de soluções” (Apêndice 7 e 8). 3.2.1. Extração de DNA A técnica de extração de DNA por Fenol – Clorofórmio descrita por SAMBROOK e cols., 1989, modificada, foi o método de escolha para realização deste trabalho. Esta técnica proporciona um produto limpo, garantindo a amplificação de regiões repetitivas como é o caso das expansões CAG e a detecção por fluorescência. O sangue foi coletado em tubo com EDTA 5% (BD Vacutainer) e centrifugado por 15 minutos a 5000 rpm. Em tubos Eppendorf separou-se a nata de leucócitos e adicionou-se 1 mL de Solução de Lise de Eritrócitos 10X (SLE 10X). Após homogeneização os tubos Eppendorf foram centrifugados a 12000 rpm durante 10 minutos e o sobrenadante foi descartado. Estas duas últimas etapas foram repetidas por mais duas vezes. Ao precipitado adicionou-se proteínase K na concentração final 1mg/mL e Dodecilsulfato de Sódio (SDS) para concentração final 0,5%. Após misturar por inversão, os tubos foram incubados em banho-maria de 550C por 2 horas no mínimo. Decorrido o tempo, 500µl de fenol saturado pH 8,0 foi adicionado. As amostras foram homogeneizadas e centrifugadas em 12000 rpm por 10 minutos. A fase aquosa (superior) foi retirada para tubo Eppendorf limpo e 500µl de fenol saturado pH 35 8,0 e 400µl de clorofórmio foram adicionados. Novamente as amostras foram homogeneizadas e centrifugadas (12000 rpm por 10 minutos) e a fase aquosa (superior) foi transferida para outro tubo Eppendorf limpo. Adicionou-se ao sobrenadante 300µl de Acetato de Sódio 3M na amostra. Após homogeneização a amostra foi centrifugada (12000 rpm por 10 minutos). Despejou-se o sobrenadante sobre etanol absoluto gelado. O tubo foi invertido lentamente por várias vezes até precipitar o DNA. Lavou-se o precipitado com 500µl etanol 70% gelado e desprezou-se o sobrenadante. Após secar o DNA, adicionou-se de 50 a 200 µl de TE (Solução de Tris-EDTA), dependendo do tamanho do precipitado do DNA. 3.2.2. Amplificação dos genes de interesse A amplificação foi realizada pela técnica de Reação em Cadeia da Polimerase (PCR) que foi descrita por Kary Mullis na década de 1980. A PCR possibilita a produção de numerosas cópias de seqüências especificas de DNA. O número de repetições CAG de indivíduos assintomáticos está dentro de um limite que abrange alelos distintos chamados de normais. Devido ao menor tamanho destes alelos em relação aos alelos expandidos, a enzima apresenta uma atividade maior, gerando um aumento na concentração do produto de amplificação de alelos ditos normais. Esta amplificação preferencial de alelos normais em relação aos alelos expandidos pode acarretar em resultados “falso negativo”. Conhecendo este fato, foi imprescindível a padronização prévia dos protocolos de amplificação usando ambos alelos (normais e expandidos) obtidos através de amostras de indivíduos que haviam sido tipados para estes genes por outros laboratórios. Diferentes marcadores fluorescentes foram selecionados com base nos tamanhos dos alelos de indivíduos assintomáticos descritos previamente [DORSCHNER e cols., 2002]. 36 Assim, garantiu-se que loci cujos alelos possam apresentar o mesmo tamanho, recebam fluoróforos distintos (FAM, VIC e NED). Cada indivíduo foi submetido a um total de três reações de PCR. Uma reação triplex para AEC 1, AEC 2 e AEC 6; outra reação para AEC 3 e uma terceira reação para amplificação de AEC 7. Foram utilizados nessas reações 10 a 30 ng de DNA genômico com volume final de 15 µL. As condições de PCR foram realizadas no termociclador MJ research PTC model (MJ research,inc. Watertown, MA, USA). O protocolo de amplificação para as expansões CAG em AEC 1, AEC 2 e AEC 6 foi padronizado como uma reação triplex visando à amplificação concomitante das respectivas três regiões de interesse em um único tubo. Para esta reação foi utilizado o sistema Accuprime DNA polimerase da Invitrogen/Stratagene, La Jolla, CA, USA. A reação de PCR continha 1X tampão BII (2 mM de cada nucleotídeo; 1,5 mM de MgCl2; 200mM Tris-HCl pH 8,4), 1,5U Accuprime Taq DNA polimerase e 1,5 pmol de cada iniciador (universal e reverso). As seqüências utilizadas foram: para AEC 1 (1F_FAM_AACTGGAAATGTGGACGTAC [KAMEYA e cols., 1995], AEC 2R_CGGGCTTGCGGACATTGG) 2 e 1R_CAACATGGGCAGTCTGAG) (2F_VIC_GGGCCCCTCACCATGTCG [PULST e (6F_NED_CACGTGTCCTATTCCCCTGTCATCC cols., 1996] e AEC e 6 e 6R_TGGGTACCTCCGGAGGGCCGCTGGTG) [ZHUCHENKO e cols., 1996]. As condições no termociclador foram de 1 ciclo a 96°C por 10 minutos; 32 ciclos de três temperaturas sendo: 1 minuto a 94°C para desnaturação, rampa de 0,2oC/segundo, 1 minuto e 30 segundos a 61°C para anelamento e 1 minuto e 30 segundos a 68°C; e 1 ciclo de 1 hora a 68°C para extensão final. A reação de amplificação para as expansões CAG para a AEC 3 foi realizada com 200 mM de cada nucleotídeo, 1X tampão de PCR (10mM Tris-HCl; pH9,0), 37 50mM KCl e 0,1% Triton X); 1,5 mM de MgCl2 25mM e 1,5 U Taq DNA polimerase. A seqüência de iniciadores foi desenhada para este estudo (Programa DNAStar) e 12 pmol de cada iniciador para o locus AEC 3 (3F_NED_CTGGCCTTTCACATGG e 3R_CCAGTCACTACTTTGATTCGTG) foi utilizado (Figura 3). No termociclador, as condições utilizadas foram padronizadas pela metodologia de PCR-Touchdown cols., 1991] [DON e e foram de 1 ciclo de 10 minutos a 95°C; 5 ciclos de três temperaturas de 1 minuto a 95°C para desnaturação, 1 minuto a 59°C para anelamento, 1 minuto e 30 segundos a 72°C para extensão; 20 ciclos de três temperaturas de 1 minuto a 95°C para desnaturação, 1 minuto a 60°C para anelamento, 1 minuto e 30 segundos a 72°C para extensão; 7 ciclos de três temperaturas de 1 minuto a 95°C para desnaturação, 1 minuto a 61°C para anelamento, 1 minuto e 30 segundos a 72°C para extensão; e 1 ciclo de 1 hora a 72°C para extensão final. 5’atctgtatcagactaactgctcttgcattcttttaataccagtgactactttgattcgtgaaacaatgtattttccttatgaa tagtttttctcatggtgtatttattcttttaagttttgttttttaaatatacttcacttttgaatgtttcagacagcagcaaaagcagc aacagcagcagcagcagcagcagcagggggacctatcaggacagagttcacatccatgtgaaaggccagccac cagttcaggagcacttgggagtgatctaggtaaggcctgctcaccattcatcat 3’ Figura 3: Localização de anelamento dos iniciadores no locus ATXN 3. Em laranja encontram-se os iniciadores que flanqueiam a região de interesse e em azul encontra-se a região de repetição da trinca CAG. A amplificação para as expansões CAG do locus AEC 7 foi realizada com 200 mM de cada nucleotídeo, 0,7 X tampão de PCR (10mM Tris-HCl; pH9,0), 50mM KCl e 0.1% Triton X); 1,5 mM de MgCl2 e 1,5 U Taq DNA polimerase e 4% de DMSO. A seqüência de iniciadores também foi desenhada para este estudo (Programa DNAStar) e 15 pmol de cada iniciador (7F_VIC_TTGTAGGAGCGGAAAGAATGTC e 7R_CTTCAGGACTGGGCAGAGG) foram utilizados (Figura 4). O programa para amplificação desta região também foi padronizado pela metodologia de PCR- 38 Touchdown [DON e cols., 1991] sendo: 1 ciclo de 10 minuto a 96°C, 5 ciclos de três temperaturas de 1 minuto a 95°C para desnaturação, 1 minuto a 51°C para anelamento e 1 minuto e 30 segundos a 72°C extensão; 21 ciclos de três temperaturas de 1 minuto a 95°C para desnaturação, 1 minuto a 52°C para anelamento, 1 minuto e 30 segundos a 72°C para extensão; e 7 ciclos de 1 minuto a 95°C para desnaturação, 1 minuto a 55°C para anelamento, 1minuto e 30 segundos a 72°C para extensão; e por fim uma extensão final de 72°C por 1 hora. 5’ctggcgcctccttaaaaaacggcccccgcgcgactctttcccccttttttttgttacattgtaggagcggaaagaatg tcggagcgggccgcggatgacgtcaggggggagccgcgccgcgcggcggcggcggcgggcggagcagcggc cgcggccgcccggcagcagcagcagcagcagcagcagcagcagccgccgcctccgcagccccagcggcagc agcacccgccaccgccgccacggcgcacacggccggaggacggcgggcccggcgccgcctccacctcggccgc cgcaatggcgacggtcggggagcgcaggcctctgcccagtcctgaagtgatgctgggacagtcgtggaatctgtgg gtagaggcttccaaacttcatgggaagacggtgagtgtccacgccctcct 3’ Figura 4: Localização de anelamento dos iniciadores no locus ATXN 7. Em laranja encontram-se os iniciadores que flanqueiam a região de interesse e em azul encontra-se a região de repetição da trinca CAG. 3.2.3 Detecção do produto de amplificação Para detecção do produto de amplificação, foi preparado para cada indivíduo uma mistura de 1 µL da reação de PCR na proporção de 1:6 com formamida deionisada e 0,25 µl do marcador de tamanho e peso molecular GS-500 ROX para um volume final de 6µl. Após desnaturação (94o C por 3 minutos) e rápido resfriamento em gelo (3 minutos), as amostras foram injetadas no seqüenciador automático ABI PRISM 3100 Avant Genetic analyzer (Hitachi High Technologies Corporation – Tokio/Japão) com arranjo de capilar de 36 cm x 50 µm contendo Performance Optimized Polymer-4 (POP-4 Applied Biosiystems) por 5 segundos de injeção. Os alelos foram detectados em câmara CCD devido à marcação dos iniciadores com fluoróforos distintos (FAM, VIC e NED). Os dados foram capturados no Software ABI Data Collection. O tamanho dos alelos foi definido comparando-se ao marcador GS-500 ROX pelo Genemapper v3.1. 39 3.3. Análise de resultados A determinação das repetições CAG em cada alelo foi definida a partir do tamanho em pares de base (pb) obtido nas etapas anteriores. O tamanho do produto de amplificação para os alelos de cada locus de cada indivíduo foi capturado automaticamente a partir do programa Genemapper do seqüenciador automático ABI PRISM 3100 Avant. Este resultado de tamanho em pb foi obtido através do cálculo comparativo da eletroforese concomitante da amostra com o marcador de tamanho e peso molecular GS-500 ROX. Para cada alelo, o tamanho em pares de base (pb) fornecido pelo aparelho incluía a repetição CAG mais uma região residual onde ocorre o anelamento dos iniciadores. Assim, verifica-se que para AEC 1, AEC 2, AEC 3, AEC 6 e AEC 7 a região residual, sem as repetições CAG, apresenta 123 pb, 61 pb, 165 pb, 103 pb e 252 pb, respectivamente. Objetivando definir os alelos de forma fidedigna, todos os indivíduos que apresentaram um tamanho em pb aproximado do valor teórico para um determinado alelo foram agrupados em diferentes tabelas. Isto foi realizado para cada alelo dos cinco loci. Usando análise estatística do tamanho em pares de base, foram obtidas as médias, os desvios padrões (DP) e as variâncias de cada alelo para as duas amostras populacionais em análise: indivíduos clinicamente normais e pacientes com suspeita clínica de ataxia. Um segundo cálculo foi realizado para obtenção do número de repetições CAG com auxílio de planilha do Excell. O número de repetições CAG foi definido através da subtração do tamanho fornecido pelo aparelho com a região residual, e dividido por três, em virtude dos três nucleotídeos da unidade de repetição CAG. Cada alelo representa o 40 número de repetição CAG. O cálculo para a obtenção do número das repetições CAG fica definido como explicitado na tabela 3. Desses resultados, foi possível realizar análise estatística com auxílio do programa SSPS v. 13.0 e cálculos do princípio de Hardy-Weinberg (HW) e de heterozigosidade (H) com auxílio do programa GENEPOP (version 3.4) [RAYMOND e ROUSSET 1995]. Tabela 3: Cálculo das repetições CAG em cada locus Doença/Gene Produto de Amplificação (pb) Número de repetições CAG AEC 1/ATXN1 Repetições (CAG)n + 123 (Produto de Amplificação - 123)/3 AEC 2/ATXN2 Repetições (CAG)n + 61 (Produto de Amplificação - 61)/3 DMJ/ATXN3 Repetições (CAG)n + 165 (Produto de Amplificação -165)/3 AEC 6/CACNA1 Repetições (CAG)n + 103 (Produto de Amplificação - 103)/3 AEC 7/ATXN7 Repetições (CAG)n + 252 (Produto de Amplificação - 252)/3 41 IV. RESULTADOS 42 4.1. Produtos de amplificação Os alelos analisados de tamanho normal, nas reações de PCR resultaram em um produto de amplificação consistente, com picos precisos, sem resíduos no fundo do eletroferograma (Figura 5). Figura 5: Amplificação dos alelos de tamanho normal para os genes ATXN1, ATXN2, ATXN3, ATXN6 e ATXN7- picos precisos, sem resíduos no fundo do eletroferograma Nos alelos expandidos anormais, que foram observados na amostra dos pacientes, verificam-se bandas de mosaicismo somático e de “stutter” que também estão presentes em eletroforese em gel de poliacrilamida (PAGE). Entretanto, um pico dominante evidente foi observado em relação aos outros de acordo com a amplitude e a área do mesmo. Este é o pico referente ao alelo mais representativo encontrado em cada paciente (Figura 6). 43 Figura 6: Amplificação dos alelos anormalmente expandidos observados na amostra de pacientes para Ataxia Espinocerebelar 2, 3, 6 e 7. 4.2. Definição dos alelos Indivíduos com o mesmo alelo apresentaram pequenas diferenças no tamanho em pares de base (pb) a partir de valores teóricos, isto é, o número de repetições do trinucleotídeo CAG somado ao produto de amplificação residual específico para cada locus como descrito previamente. A variabilidade inter-ensaio para o mesmo alelo entre os diferentes indivíduos para cada locus AEC, foi entre 0,12 a 1,08 pb para indivíduos clinicamente normais. Entre os alelos mais freqüentes de todos os loci a maior variância foi observada no alelo 5 do locus AEC 7. A amostra de pacientes apresentou variabilidade inter-ensaio entre 0,11 a 1,66 pb (Tabela 4 a 8). 44 Tabela 4: Média (± ± DP) e variância dos alelos em pares de base (pb) entre controles e pacientes analisados para ataxia espinocerebelar 1. AEC 1 Controles Pacientes Alelos Média (pb) ± DP Var N Alelos Média (pb) ± DP Var N 19 180,45 0 1 19 - 0 - 20 183,36 0 1 20 - 0 - 23 193 0 1 23 193 0 1 24 196,04 0 1 24 - 0 - 25 199 0 1 25 198 0 1 26 201,36 ± 0,56 0,31 9 26 201,67±0,58 0,33 5 27 204,31 ± 0,54 0,29 9 27 204,93±0,70 0,49 5 28 206,7 ± 0,75 0,56 22 28 207,03 ± 0,72 0,54 11 29 210,07 ± 0,48 0,23 87 29 210,38±0,60 0,36 63 30 212,86±0,28 0,08 99 30 212,94 ± 0,43 0,2 86 31 215,70±0,34 0,14 33 31 215,76±0,44 0,2 17 32 218,57±0,51 0,26 25 32 218,63 ± 0,57 0,32 27 33 221,18±0,37 0,14 9 33 221,42 ± 0,45 0,2 9 34 223,89 ± 0,31 0,1 4 34 - 0 - 35 227,12 ± 0,5 0,25 3 35 228 0 1 36 230,03±0,07 0,01 3 36 230,39 ± 0,9 0,81 4 N= número de cromossomos. - não detectado. Var = variância. 45 Tabela 5: Média (± ± DP) e variância dos alelos em pares de base (pb) entre controles e pacientes analisados para ataxia espinocerebelar 2. AEC 2 Controles Pacientes Alelos Média (pb) ± DP Var N Alelos Média (pb) ± DP Var N 6 79 0 1 6 - 0 - 14 - 0 - 14 103 0 1 15 106 0 1 15 105 0 1 17 111 0 1 17 110,98±0,04 0,01 5 18 116,21 0 1 18 - 0 - 19 - 0 - 19 116,9±0,06 0,04 2 20 119,98±0,25 0,06 266 20 119,84±0,24 0,06 195 21 122,97 ± 0,36 0,13 23 21 123,52±0,18 0,01 16 22 125,63 ± 0,87 0,76 3 22 125 0 1 24 128 0 2 24 - 0 - 25 135 0 6 25 134,66±0,48 0,23 2 26 136,89 0 1 26 - 0 - 27 140,55 0 1 27 - 0 - 28 143,74 0 1 28 143,63±0,54 0,29 2 37 171 0 1 38 173 0 3 43 190 0 1 56 228 0 1 N= número de cromossomos. - não detectado. Var = variância. Alelos expandidos encontram-se em negrito 46 Tabela 6: Média (± ± DP) e variância dos alelos em pares de base (pb) entre controles e pacientes analisados para ataxia espinocerebelar 3. AEC 3 Controles Pacientes Alelos Média (pb) ± DP Var N Alelos Média (pb) ± DP Var N 12 196,94 ± 0,29 0,08 82 12 197,13 ± 0,4 0,16 47 13 199,55 ± 0,57 0,71 1 13 199,37 ± 0,7 0,49 4 14 - 0 - 14 201,63 0 1 15 206 0 4 15 214,44 0 1 16 207,97 0 2 16 208,52 ± 0,29 0,09 7 17 211,98 ± 0,73 0,53 7 17 212,60 ± 1,14 1,3 10 18 215,41 ± 1,03 1,06 34 18 214,84 ± 0,87 0,76 26 19 217,99 ± 1,07 1,14 12 19 217,08 ± 0,48 0,23 12 20 222,03 ± 0,35 0,12 87 20 222,23 ± 0,2 0,04 38 21 224,23 ± 1,08 1,17 18 21 223,99 ± 0,18 0,01 17 22 227,33 ± 0,92 0,85 4 22 226,83 ± 1,01 1,02 7 23 230,48 ± 0,96 0,92 4 23 230,33 ± 1,00 0,99 8 24 233,3 ± 0,28 0,08 38 24 233,6 ± 0,31 0,96 14 25 235,94 ± 0,51 0,26 7 25 236,46 ± 0,21 0,05 6 26 238,88 ± 0,97 0,94 2 26 238,82 ± 0,05 0,002 5 27 241,48 0,55 1 27 240,95 0 1 28 245 0 2 28 - 0 - 29 246,93 0,17 1 29 246,64 0 1 34 262 0 2 34 - 0 - 62 347,66 ± 0,93 0,87 2 64 354,32 ± 0,28 0,08 2 65 357,79 ± 0,26 0,66 4 66 360,83 ± 0,76 0,56 3 67 363,17 ± 1,66 2,76 2 68 365,68 ± 0,89 0,8 4 69 370 0 3 70 372,23±0,32 0,11 2 71 374,7 0 1 72 377,5 0 1 74 383 0 1 N= número de cromossomos. - não detectado. Var = variância. Alelos expandidos encontram-se em negrito 47 Tabela 7: Média (± ± DP) e variância dos alelos em pares de base (pb) entre controles e pacientes analisados para ataxia espinocerebelar 6. AEC 6 Controles Pacientes Alelos Média (pb) ± DP Var N Alelos Média (pb) ± DP Var N 2 107,93 ± 0,6 0,36 6 2 108,06±0,82 0,67 6 4 115,66 ± 0,31 0,1 25 4 115,48±0,11 0,01 3 5 116,57 0 1 5 116,97±0.09 0,01 12 8 127,88 ± 0,37 0,14 122 8 127,67±0.54 0,29 81 9 130,81 ± 0,38 0,14 57 9 130,77±0.42 0,17 33 10 133,84 ± 0,55 0,3 90 10 133,77±0.40 0,16 77 11 136,49 ± 0,72 0,52 6 11 136,41±0,82 0,68 16 12 - - - 12 140 0 1 13 143 0 1 13 - - - 21 167 0 1 N= número de cromossomos. - não detectado. Var = variância. Alelos expandidos encontram-se em negrito Tabela 8: Média (± ± DP) e variância dos alelos em pares de base (pb) entre controles e pacientes analisados para ataxia espinocerebelar 7. AEC 7 Controles Pacientes Alelos Média (pb) ± DP Var N Alelos Média (pb) ± DP Var N 2 257,17 ± 0,9 0,81 4 2 257 0 2 4 265,19 ± 0,58 0,34 218 4 265,40±0,44 0,19 142 5 267,71 ± 1,05 1,1 26 5 267,55±0,52 0,27 27 6 270,91 ± 0,67 0,45 49 6 271,15±0,34 0,11 46 7 273,66 ± 0,51 0,26 8 7 274,26±0,57 0,01 10 8 276,68 ± 0,12 0,01 2 8 - - - 10 283,3 0 1 10 - - - 39 370,37±0,52 0,27 2 50 419,51 0 1 N= número de cromossomos. - não detectado. Var = variância. Alelos expandidos encontram-se em negrito 48 A variabilidade intra-ensaio foi obtida repetindo cinco vezes a corrida eletroforética de um mesmo produto de amplificação de cinco indivíduos diferentes para os cinco loci. Esta variabilidade também foi calculada com intervalo de confiança de 95% e foi entre 0,02 a 0,69 pb (Tabela 9). A partir desses resultados foi possível definir o limite de variação de tamanho em pares de base para cada alelo de cada locus no programa do aparelho ABI 3100 Avant e proceder com os cálculos de freqüência e de estudo populacional. 4.3. Freqüência alélica Na amostra de indivíduos clinicamente normais o número de repetição CAG variou entre 19 até 36 para AEC 1 (média 29,80 ± 2,02) com 16 alelos diferentes. Na AEC 2, o número de repetições CAG variou entre 6 a 28 (média 20,18 ± 1,35) com 12 alelos distintos. Em AEC 3 o número de repetições CAG variou de 12 a 34 repetições com 18 alelos diferentes e com alta dispersão entre esses alelos (média 18,64 ± 4,57). No grupo controle o tamanho dos alelos em AEC 6 variou entre 2 a 13 repetições (média 8,52 ± 1,68) com 8 alelos diferentes. O tamanho dos alelos para AEC 7 variou de 2 a 10 repetições (média 4,87±1,11) com 7 alelos diferentes (Tabela 10). 49 Tabela 9: Média (±DP) em pb de cinco repetições de corrida eletroforética em cinco amostras diferentes para os cinco loci . Amostra I Loci Amostra II Amostra III Amostra IV Amostra V Alelo I: 29 Alelo II:30 Alelo I: 30 Alelo II: 30 Alelo I: 30 Alelo II: 31 Alelo I: 30 Alelo II: 31 Alelo I: 29 Alelo II:30 209,63 ± 0,2 212,19 ± 0,25 212,29 ± 0,21 212,29 ± 0,21 212,34 ± 0,04 214,65 ± 0,69 212,30 ± 0,13 216,16 ± 0,06 209,41 ± 0,05 212,29 ± 0,06 Alelo I: 20 Alelo II: 20 Alelo I: 20 Alelo II: 20 Alelo I: 25 Alelo II: 28 Alelo I: 20 Alelo II: 21 Alelo I: 20 Alelo II: 20 119,30 ± 0,08 119,30 ± 0,08 119,09 ± 0,09 119,09 ± 0,09 133,91± 0,04 142,54 ± 0,30 119,07± 0,10 122,10 ± 0,06 119,00 ± 0,05 119,00 ± 0,05 Alelo I: 12 Alelo II: 27 Alelo I: 20 Alelo II: 20 Alelo I: 12 Alelo II: 20 Alelo I: 12 Alelo II: 12 Alelo I: 12 Alelo II: 12 197,36 ± 0,02 254,28 ± 0,19 222,35 ± 0,11 222,35 ± 0,11 197,32 ± 0,03 222,19 ± 0,02 197,29 ± 0,02 197,29 ± 0,02 197,36 ± 0,10 197,36 ± 0,10 AEC 6 Alelo I: 8 Alelo II: 10 Alelo I: 10 Alelo II: 10 Alelo I: 5 Alelo II: 10 Alelo I: 8 Alelo II: 8 Alelo I: 9 Alelo II: 9 127,92 ± 0,06 133,68 ± 0,07 133,67 ± 0,04 133,67 ± 0,04 116,48 ± 0,04 133,71 ± 0,04 127,88 ± 0,03 127,88 ± 0,03 130,7 ± 0,02 130,7 ± 0,02 AEC 7 Alelo I: 4 Alelo II: 6 Alelo I: 2 Alelo II: 4 Alelo I: 4 Alelo II: 4 Alelo I: 4 Alelo II: 6 Alelo I: 4 Alelo II: 6 265,42 ± 0,06 271,16 ± 0,05 257,73 ± 0,06 265,36 ± 0,06 265,36 ± 0,08 265,36 ± 0,08 265,47 ± 0,04 271,26 ± 0,1 265,39 ± 0,12 271,3 ± 0,07 AEC 1 AEC 2 AEC 3 50 Tabela 10: Freqüência alélica % (± ± EP%) nos cinco loci analisados para Ataxia espinocerebelar 1, 2, 3, 6 e 7 entre controles e pacientes. AEC 1 AEC 2 AEC 3 Alelos Controles Pacientes Alelos Controles Pacientes Alelos Controles Pacientes 19 0,35 ± 0,34 - 6 0,35 ± 0,34 - 12 26,57 ± 2,53 20,43 ± 2.89 20 0,35 ± 0,34 - 14 0,35 ± 0,34 0,43 ± 0,48 13 0,35 ± 0,34 1,74 ± 0,93 23 0,35 ± 0,34 0,54±0,54 15 0,35 ± 0,34 0,43 ± 0,48 14 - 0,43 ± 0,48 24 0,35 ± 0,34 - 17 0,35 ± 0,34 2,17 ± 1,06 15 1,4 ± 0,67 0,43 ± 0,48 25 0,35 ± 0,34 0,54±0,54 19 0,35 ± 0,34 0,87 ± 0,67 16 0,7 ± 0,48 3,04 ± 1,23 26 2,78 ± 0,94 2,15±1,06 20 84,44±1,95 84,35 ± 2,64 17 2,45 ± 0,88 4,34 ± 1,46 27 2,78 ± 0,94 2,15±1,06 21 7,29 ± 1,48 7,00 ± 1,85 18 10,84 ± 1,78 11,3 ± 2,27 28 7,29 ± 1,48 4,84±1,57 22 1,04 ± 0,57 0,43 ± 0,48 19 3,85 ± 1,10 5,22 ± 1,60 29 28,11±2,56 27,42±3,27 23 0,69 ± 0,47 - 20 27,96 ± 2,57 16,52 ± 2,67 30 32,29±2,66 37,1±3,54 25 1,74 ± 0,74 0,87 ± 0,67 21 5,94 ± 1,35 7,39±1,88 31 10,76±1,77 7,53±1,93 26 0,35 ± 0,34 - 22 1,4 ± 0,67 3,04 ± 1,23 32 7,99 ± 1,54 11,83±2,37 27 0,35 ± 0,34 - 23 1,4 ± 0,67 3,47 ± 1,32 33 2,78 ± 0,94 3,76±1,39 28 0,35 ± 0,34 0,87 ± 0,67 24 12,24 ± 1,87 6,09 ± 1,72 34 1,39 ± 0,67 - 37 - 0,43 ± 0,48 25 2,1 ± 0,82 2,61 ± 1,14 35 1,04 ± 0,58 0,54±0,54 38 - 1,30 ± 0,82 26 0,7 ± 0,48 2,17 ± 1,05 36 1,04 ± 0,58 1,6±0,92 43 - 0,43 ± 0,48 27 0,35 ± 0,34 0,43 ± 0,48 56 - 0,43 ± 0,48 28 0,7 ± 0,48 - 29 0,35 ± 0,34 0,43 ± 0,48 34 0,7 ± 0,48 - AEC 6 AEC 7 Alelos Controles Pacientes Alelos Controls Pacientes 62 - 0,87 ± 0,67 2 2,08 ± 0,81 2,66±1.17 2 1,15 ± 0,67 2,17 ± 1,34 64 - 0,87 ± 0,67 4 7,98 ± 1,54 1,06±0.75 4 70,61 ± 2,88 64,38 ± 4,38 65 - 1,74 ± 0,94 5 0,35 ± 0,34 5,32±1.64 5 8,4 ± 1,75 10,00 ± 2,75 66 - 1,30 ± 0,81 8 39,59±2,79 35,11±3.48 6 16,03 ± 2,32 18,26 ± 3,54 67 - 0,87 ± 0,67 9 18,4 ± 2,21 14,36±2.56 7 2,67 ± 1,02 3,48 ± 1,68 68 - 1,74 ± 0,94 10 29,17±2,59 33,52 ± 3,44 8 0,76 ± 0,55 - 69 - 1,30 ± 0,81 11 2,08 ± 0,81 6,91 ± 1,85 10 0,38 ± 0,39 - 70 - 1,03 ± 0,72 12 - 0,53 ± 0,53 39 - 0,87 ± 0,85 71 - 0,43 ± 0,48 13 0,35 ± 0,34 - 50 - 0,43 ± 0,60 72 - 0,43 ± 0,48 21 - 0,53 ± 0,53 74 - 0,43 ± 0,48 – não detectado. Alelos expandidos encontram-se em negrito. 51 Entre pacientes o número dos alelos variou de 23 a 36 repetições em AEC 1 (media 29,96 ± 1,74) com os 12 alelos diferentes. Nenhum alelo expandido foi encontrado no locus AEC 1. No locus AEC 2 o tamanho dos alelos variou de 14 a 56 repetições (média 20,65 ± 3,83). O locus AEC 2 mostrou quatro alelos expandidos em seis pacientes. O locus AEC 3 tem o tamanho dos alelos variando de 12 a 74 repetições com 28 alelos diferentes (média de 24,65 ± 16,93). Há 11 alelos expandidos em 25 pacientes. O tamanho entre os alelos em AEC 6 variou de 2 a 21 repetições (8,75± 2,04) com 9 alelos diferentes. Somente um alelo expandido para o locus AEC 6 foi encontrado em apenas um paciente. Estes pacientes no locus AEC 7 apresentaram o tamanho dos alelos variando de 2 a 50 repetições (média 5,60 ± 6,12) e dois alelos expandidos foram encontrados em três pacientes (Tabela 10). Das AEC pesquisadas, a ataxia mais freqüente entre os pacientes é AEC 3, seguido de AEC 2, AEC 7 e AEC 6. Nenhum caso de AEC 1 foi encontrado (Tabela 11). Tabela 11: Freqüência das doenças nos 115 indivíduos com suspeita clínica Doença Freqüência (%) Número de indivíduos AEC 1 0 0 AEC 2 5,22 6 AEC 3 21,74 25 AEC 6 0,87 1 AEC 7 2,61 3 Todos os indivíduos que apresentaram expansão são heterozigotos apresentando um alelo normal e o outro alelo expandido. 4.4. Equilíbrio de Hardy-Weinberg e heterozigosidade Nas tabelas 12 a 16 encontram-se os principais genótipos na amostra de controles para os genes das ataxias estudadas. Encontram-se também os respectivos 52 números de indivíduos observados e os números de indivíduos esperados. O cálculo para o número de indivíduos esperados foi realizado de acordo com o equilíbrio de Hardy – Weinberg (HW). Tabela 12: Números de indivíduos observados e esperados para os possíveis genótipos do gene ATXN 1 na amostra do grupo controle. Genótipos 28/27 29/27 29/28 29/29 30/26 30/28 30/29 30/30 31/29 31/30 32/29 32/30 32/31 32/32 33/29 33/31 34/30 36/30 Outros TOTAL Observado 2 2 7 9 5 9 27 14 11 11 7 7 2 2 3 3 2 2 29 154 Esperado 0,585 2,258 5,927 11,289 2,592 6,805 26,247 14,906 8,749 10,045 6,491 7,453 2,484 0,882 2,258 0,864 1,296 0,972 41,897 154 53 Tabela 13: Números de indivíduos observados e esperados para os possíveis genótipos do gene ATXN 2 na amostra do grupo controle. Genótipos Observado Esperado 111 108,449 20/20 14 18,293 21/20 2 0,732 21/21 2 2,613 22/20 2 1,742 24/20 4 4,355 25/20 19 17,816 Outros TOTAL 154 154 Tabela 14: Números de indivíduos observados e esperados para os possíveis genótipos do gene ATXN 3 na amostra do grupo controle. Genótipos 12/12 17/12 18/12 18/18 19/12 20/12 20/18 20/19 20/20 21/12 21/20 23/20 24/12 24/18 24/20 24/24 Outros TOTAL Observado 10 3 10 5 3 15 7 4 15 6 8 2 13 4 7 2 80 154 Esperado 10,000 1,867 8,267 1,632 2,933 21,333 8,702 3,088 11,088 4,533 4,772 1,123 9,333 3,807 9,825 2,088 49,609 154 54 Tabela 15: Números de indivíduos observados e esperados para os possíveis genótipos do gene CACNA 1 na amostra do grupo controle. Genótipos Observado Esperado 4 2,383 8/2 10 9,136 8/5 21 22,443 8/8 4 4,247 9/5 23 21,052 9/8 5 4,801 9/9 8 6,732 10/5 30 33,366 10/8 14 15,512 10/9 15 12,146 10/10 3 2,383 11/8 35 19,799 Outros TOTAL 154 154,000 Tabela 16: Números de indivíduos observados e esperados para os possíveis genótipos do gene ATXN 7 na amostra do grupo controle. Genótipos 4/2 4/4 5/4 6/4 6/5 7/4 8/4 Outros TOTAL Observado 2 60 18 36 4 6 2 26 154 Esperado 2,126 65,211 15,594 29,770 3,540 4,962 1,418 31,379 154,000 Calculando-se o Qui-quadrado foi possível determinar se há diferença significativa entre o observado e o esperado. Em nenhuma situação, a probabilidade obtida após cálculo no Qui-quadrado foi significante, indicando não haver nenhum desvio no equilíbrio de HW em nenhuma das distribuições genotípicas encontradas dos cinco loci analisados (Tabela 17). 55 Tabela 17: Probabilidade obtida após aplicação do teste do Qui Quadrado (χ χ2) no qual a hipótese nula é que não há diferença significativa entre as freqüências genotípicas observadas e esperadas nos cinco loci analisados (Equilíbrio de HardyWeinberg). Loci χ2 p Graus de Liberdade AEC 1 12,87 0,80 18 AEC 2 2,10 0,83 5 AEC 3 20,13 0,33 18 AEC 6 4,73 0,97 12 AEC 7 5,04 0,65 7 Os dados do heterozigosidade (H) que mostram a diversidade entre indivíduos dentro desta amostra para os cinco loci encontram-se na Tabela 18. Tabela 18: Heterozigosidade obtida nos cinco loci analisados. Loci Heterozigosidade (H) AEC 1 0,79 AEC 2 0,24 AEC 3 0,82 AEC 6 0,72 AEC 7 0,47 4.5. Análise Clínica Muitos dos 115 pacientes que deram entrada no Hospital de Clínicas a fim de contribuir com a amostra pertencem a outros centros de diagnóstico clínico. Assim sendo, não existe rastreabilidade dentro do Hospital de Clínicas para avaliação dos dados clínicos de todos esses indivíduos. Portanto, a análise clínica foi realizada somente em 56 pacientes cujos prontuários foram encontrados entre os 115 indivíduos da amostra com suspeita clínica. Destes 56 indivíduos, 31 são do sexo masculino (55,36 %) e 25 do sexo feminino (44,64 %) cujos principais sinais neurológicos foram marcha 56 atáxica (49 pacientes) e disartria (45 pacientes) (Tabela 19). Maiores detalhes clínicos dos pacientes estão descritos no Apêndice 9. 57 Tabela 19: Dados gerais de indivíduos com as características clínicas da amostra dos 56 pacientes analisados Subtipos Diagnóstico de AEC Molecular Média de Idade Sexo F Mutações Herdadas M Mãe Pai (±DP) - Principais Sinais Neurológicos Marcha Disartria Atáxica Tremor Nistagmo/ Movimentos Sinais Intensional Oftalmoplegia Sacádicos Piramidais / Lentos Espasticidade 0 0 0 0 Início Sint. AEC 1 0 0 0 0 0 0 0 0 AEC 2 5 (8,93) 31,83±10,32 3 (50) 3 (50) 4 (0,93) 1 (0,29) 4 (3,5) 6 (4,82) 1 (0,29) 1 (0,5) 4 (0,93) 0 AEC 3 14 (25,01) 35,08±9,05 6 (40) 8 (60) 6 (1,82) 8 (2,57) 13 (11,38) 13 (10,45) 2 (0,57) 12 (6) 3 (0,7) 3 (0,48) AEC 6 0 0 0 0 0 0 0 0 0 0 0 0 AEC 7 3 (5,36) 34,67±16,16 0 3 (100) 1 (0,30) 2 (0,64) 3 (2,62) 3 (2,41) 1 (0,29) 1 (0,5) 1 (0,23) 2 (0,32) Normal 34 (60,71) 29,25±13,98 16 (48) 17(52) 5 (1,52) 7 (2,25) 29 (25,38) 23 (18,48) 12 (3,43) 14 (7) 5 (1,16) 4 (0,64) TOTAL 56 (100) 31,26±12,43 25 (44,64) 31(55,35) 17(30,36) 18(32,14) 49 (87,5) 45(80,36) 16(28,57) 28(50,0) 13(23,21) 9(16,07) Os números em parênteses representam as freqüências (%). 58 A idade média foi 31,26 ± 12,43 anos. O diagnóstico molecular foi confirmado pela eletroforese de capilar em 22 pacientes sendo: cinco pacientes para AEC 2, quatorze pacientes AEC 3 e três pacientes em AEC 7. Em onze pacientes cujos alelos expandidos foram encontrados, a doença foi transmitida pela mãe e a idade média de início dos sintomas foi 38,45 ± 7,19. Em outros onze pacientes os alelos expandidos foram transmitidos pelo pai e a média de idade de início dos sintomas foi 27,73 ± 9,14. O teste “t” para amostras independentes comparando a idade média de início dos sintomas entre os sexos dos genitores transmissores da doença mostra diferenças significativas entre o sexo do genitor que transmite o gene (t=3,05; p=0,006; α=95%). A manifestação é mais precoce quando o sexo do genitor é masculino. Comparando-se o diagnóstico molecular com o diagnóstico clínico foi possível estabelecer uma relação entre eles determinando se existe diferença significativa entre as duas formas de diagnóstico (Tabela 20). Além disso, foi possível calcular a sensibilidade, a especificidade, o valor preditivo positivo (VPP) e o valor preditivo negativo (VPN) do teste. Os valores contidos na tabela 20 referem-se a todos os 115 pacientes uma vez que estes foram submetidos ao teste molecular por apresentarem alguma suspeita clínica de ataxia, isto é, o teste foi realizado em indivíduos com sintoma de ataxia. Entretanto, é muito importante salientar que sendo o Hospital de Clínicas um centro de referência, muitos pacientes são encaminhados com suspeita de ataxia, porém sem a precisa definição do diagnóstico clínico em virtude da grande sobreposição de sinais e sintomas, tornando a amostra de pacientes bastante heterogênea. 59 Tabela 20: Comparação entre diagnóstico molecular e clínico dos 115 pacientes para AEC 1, AEC 2, AEC 3, AEC 6 e AEC 7 Diagnóstico Molecular Suspeita clínica para pelo menos um dos Expansão Sem expansão 28 (a) 17 (b) 45 (a+b) 7 (c) 63 (d) 70 (c+d) 80 (b+d) 115 subtipos analisados Outras ataxias e Diagnóstico a esclarecer 35 (a+c) 2 χ = 32,86 p = 0,000 Sensibilidade: a/(a+c) = 80,00 % VPP: a/(a+b) = 62,22 % Especificidade: d/(b+d) = 78,75 % VPN: d/(c+d) = 90,00 % Estatisticamente há diferenças significativas entre os dois diagnósticos (χ2=32,86; p=0,00). Em 45 indivíduos clinicamente positivos, somente 28 foram confirmados pelo diagnóstico molecular e sete indivíduos, que apresentaram expansão, não foram definidos clinicamente com nenhum dos subtipos de ataxia estudado, isto é, apenas o diagnóstico molecular foi capaz de detectá-los (Tabela 21). O teste de “t” para amostras independentes foi usado para comparar as médias dos alelos entre pacientes e o grupo controle. Estes resultados mostraram diferenças significativas para os loci de AEC 2 e AEC 3 (Tabela 22). 60 Tabela 21: Número de pacientes com a respectiva suspeita clínica e diagnóstico molecular da amostra total de 115 pacientes. Suspeita Clínica Diagnóstico Molecular AEC 1 Número de casos Confirmada - Sem alelos AEC 1 AEC 2 AEC 3 AEC 6 AEC 7 expandidos - AEC 2 7 71,4% - 5 - - - 2 AEC 3 30 66,7% - - 20 - - 10 AEC 6 5 20% - - - 1 - 4 AEC 7 3 66,7% - - - - 2 1 Diagnóstico a esclarecer 70 - - 1 5 - 1 63 Total 115 - - 6 25 1 3 80 Outras AEC e – não detectado. Indivíduos confirmados somente pelo diagnóstico molecular encontram-se em negrito. Tabela 22: Teste “t” para amostras independentes comparando a média dos alelos de todos os 115 pacientes e da população assintomática (Controles). MC MP NC NP DPC DPP t p Valor F p AEC 1C vs. AEC 1P 29,81 29,97 308 230 2,02 1,68 -0,94 0,35 1,44 0,004 AEC 2 C vs. AEC 2 P 20,18 20,84 308 230 1,35 4,13 -2,26 0,025 9,36 0,00 AEC 3 C vs. AEC 3 P 18,48 24,75 308 230 4,5 0,00 AEC 6 C vs. AEC 6 P 8,51 8,89 308 230 1,68 2,64 -1,87 0,062 2,46 0,00 0,00 AEC 7 C vs. AEC 7 P 4,5 5,2 308 230 0,99 5,12 -1,79 0,076 26,45 0,00 17,22 -5,23 14,63 C- Controles; P- Pacientes; M- média; N- número de cromossomos; DP- desvio padrão; gl- graus de liberdade; p- probabilidade. Valores de p significantes encontram-se em negrito. 61 V. DISCUSSÃO 62 5.1. Considerações sobre a metodologia de análise de fragmento em eletroforese de capilar O genoma de mamíferos apresenta quantidades enormes de seqüências de DNA repetitivas sendo, em sua maioria, identificadas como trechos de uma única unidade que se repete subseqüentemente. Esta unidade de repetição quando varia de 2 a 6 pb é conhecida como microsatélites. O estudo de microsatélites tem sido útil para comparações genéticas e para mapeamento do genoma devido a características como o alto grau de polimorfismo e por estarem distribuídos em várias regiões ao longo do genoma. Muito embora as regiões de microsatélites estejam distribuídas em maior parte nas regiões espaçadoras de genes, estas podem também estar presentes em genes funcionais encontrando-se tanto na região não codificadora (intron) quanto na região codificadora (exon) [BROOK e cols., 1992; CUMMINGS e ZOGHBI, 2000; DAVID e cols., 1997; KOOB e cols., 1999; LA SPADA e cols., 1991; ORR e cols., 1993; PULST e cols., 1996]. O diagnóstico molecular destas doenças de expansão é muitas vezes bastante controverso por questões éticas e por faltar um tratamento definitivo e específico. Entretanto, a possibilidade de diagnóstico oferece ao clínico e ao paciente e seus familiares, o esclarecimento do possível prognóstico contribuindo também para o aconselhamento genético e planejamento familiar. Além disso, o diagnóstico molecular também oferece uma perspectiva para que possíveis estratégias terapêuticas possam ser adotadas em um futuro, à medida que estudos moleculares entre outros forem desvendando os processos celulares de degeneração e os tratamentos para as mesmas [SCHÖLS e cols., 2004]. A metodologia de análise de fragmento em eletroforese de capilar tem sido utilizada para determinação do número de repetições em algumas doenças de expansão [LE e cols., 1997]. A partir de uma análise cuidadosa é possível diferenciar 63 indivíduos com o número normal de repetições do indivíduo com número expandido e também de indivíduos que apresentam alelos intermediários. Neste estudo, pequenas variações no tamanho em pares de base (pb) foram observadas entre indivíduos que apresentam o mesmo alelo a partir de valores teóricos. Esta variação é provavelmente ocasionada por fatores tais como a temperatura de eletroforese, o número do lote do polímero e o tempo de uso do capilar [LARSEN e cols., 1997; LE e cols., 1997; ROSENBLUM e cols., 1997]. Para definir os alelos de forma fidedigna foi necessário separar indivíduos com alelos de tamanho em pares de base aproximado e calcular a média, o desvio e a variância de cada alelo em cada locus. Verificam-se pequenas diferenças no desvio e na variância para um mesmo alelo, que de modo algum compromete a interpretação dos resultados. Este resultado significa que os protocolos definidos apresentam reprodutibilidade, credibilidade e precisão metodológica. Os métodos de diagnóstico utilizados para definir alelos em regiões de repetição têm sido publicados geralmente comparando a eletroforese de capilar com experimentos em gel de poliacrilamida (PAGE). Há uma discrepância no tamanho do produto de amplificação entre estas metodologias. Observa-se que dados obtidos em eletroforese de capilar apresentam tamanho dos alelos virtualmente menor. Este fato deve-se a migração mais rápida em eletroforese de capilar, acarretando em um ou dois alelos (representados pelos picos no gráfico) a menos do que é obtido na PAGE [LARSEN e cols., 1997; LE e cols., 1997; DORSCHNER e cols., 2002]. Os fatores que contribuem para estas discrepâncias podem ser efeitos “crosslinked” que é maior na PAGE porque há maior resistência na corrida do produto de amplificação quando comparada ao polímero da eletroforese de capilar. Outros fatores podem ser: o efeito eletro–osmótico associado à duração da capacidade 64 desnaturante do polímero e as diferenças na estrutura secundária entre os produtos de amplificação e os marcadores de peso e tamanho molecular [ROSENBLUM 1997]. e cols., As diferenças de estrutura são importantes uma vez que o tamanho dos alelos é definido através de cálculo matemático pelo programa comparando o marcador com o fragmento amplificado através de corrida concomitante. No presente estudo verificou-se a presença do alelo de 36 repetições no locus ATXN 1 em indivíduos assintomáticos e em indivíduos com suspeita clínica da doença. O gene ATXN 1 apresenta na seqüência do DNA de alelos normais uma interrupção da região de repetição CAG com a trinca CAT. Alelos expandidos não apresentam esta interrupção. Esta trinca CAT no interior das repetições CAG parece ser a responsável em manter a estabilidade dos alelos normais durante a replicação do DNA. Alelos normais para este gene apresentam a faixa de 6 a 39 repetições CAG com interrupções de 1 a 4 trincas CAT na região. Alelos expandidos apresentam 39 a 81 repetições sem interrupções CAT 1993]. [MATILLA e cols., Analisando indivíduos com o alelo de 39 repetições CAG, ZHULKE e cols. em 2002 verificaram que quando não há a interrupção CAT existe manifestação clínica da doença. Entretanto, quando a trinca CAT está presente, indivíduos com 39 repetições são fenotipicamente normais. Alelos intermediários variam de 36 a 41 repetições CAG. Indivíduos tipados para esta alelo de 36 repetições não necessariamente apresentarão sinais e sintomas de AEC 1, possivelmente sendo um alelo de penetrância reduzida. A presença deste alelo em indivíduos assintomáticos pode ser um indício que pacientes apresentando 36 repetições CAG são normais para AEC 1. Evidentemente, o sequenciamento deste gene nesses indivíduos e o acompanhamento clínico nos casos com suspeita clínica para AEC 1 é indispensável. 65 Além disso, em AEC 2 também é observado que indivíduos com alelos intermediários de 32 e 33 repetições CAG, a presença da trinca CAA no interior das repetições, confere um fenótipo normal [IMBERT e cols., 1996]. Toda esta especulação pode levar a compreensão exata de como alelos intermediários são estáveis evitando o surgimento de alelos expandidos. Outro fato interessante é que alelos intermediários apresentam uma ambigüidade entre a possibilidade do fenótipo normal e doente sendo importante elucidar possíveis elementos cis envolvidos com mecanismos moleculares que podem levar à um ou ao outro fenótipo. Em AEC 2 o alelo mais freqüente é o de 20 repetições CAG (86,44%). Em outras populações o alelo descrito como mais freqüente, sendo 94 % do total de alelos, apresenta 22 repetições [IMBERT e cols., 1996; SANPEI e cols., 1996; PULST e cols., 1996]. Para o locus ATXN 3 o número de repetições CAG variou de 12 a 34 repetições sendo os alelos com 12 e 20 repetições CAG (54,53% somados) os mais freqüentes. A análise deste locus em famílias brasileiras com história clínica de Doença de Machado Joseph (DMJ) realizado por LOPES-CENDES e cols., 1997 mostrou que o tamanho dos alelos variou de 12 a 33 repetições CAG. Este mesmo locus em outras populações apresentou os alelos de 14 e 23 repetições CAG como mais freqüentes, sendo de 48,4% a freqüência dos dois alelos somados [LIMPRASERT e cols., 1996]. As comparações de freqüências alélicas devem ser feitas com estudos populacionais que utilizem a metodologia de eletroforese de capilar. Provavelmente as diferenças observadas entre o tamanho dos alelos deste estudo e os de outros autores como os citados são ocasionadas por diferenças nas metodologias. Devido à freqüência similar, pode tratar-se do mesmo alelo. 66 A melhor forma de definir o número exato de repetições é o isolamento do gene por clonagem molecular, com subseqüente seqüênciamento. Isto deve ser feito com pelo menos alguns indivíduos da amostra. Assim, é possível realizar o cálculo de correção do número de repetições CAG através da correlação dos resultados das amostras de controles e de indivíduos com suspeita clínica analisados por EC e por sequenciamento. 5.2. Importância do estudo populacional Além das diferenças metodológicas, existe ainda a variabilidade populacional das repetições uma vez que cada população humana pode apresentar um perfil genético próprio e diferente das demais como é verificado no presente trabalho. Na população brasileira, no locus ATXN 1 a faixa de alelos normais foi descrita entre 29 a 37 repetições CAG [JARDIM e cols., 2001; SILVEIRA e cols., 2002]. Neste estudo foram encontrados alelos na população do Paraná com menos de 29 repetições em ambas as amostras (Tabela 4). Em ATXN 2 não foi descrito ainda em outras populações [CANCEL e cols., 1997] o alelo de 6 repetições CAG que foi observado na população assintomática deste estudo (Tabela 5). Na população brasileira a faixa de alelos varia para este locus de 19 a 24 repetições 2002] enquanto [JARDIM e cols., 2001; SILVEIRA e cols., que este estudo apresentou a variação dos alelos normais de 6 a 28. O locus da DMJ apresentou alelos expandidos variando de 62 a 74 repetições CAG. Alelos expandidos de 62-65 repetições não foram encontrados nem por JARDIM e cols. em 2001 em populações brasileiras do Rio Grande do Sul e nem por SILVEIRA e cols. em 2002. Estes alelos expandidos podem se tratar de alelos com penetrância reduzida porque não são encontrados em todas as populações de pacientes embora já tenham sido descritos em pacientes com a doença [GIUNTI e cols., 67 1995]. A variação alélica encontrada no locus CACNA 1 assim como em ATXN 7 revela a presença de alelos de 2 repetições CAG que não foram descritos em literatura ainda. Além disso, neste trabalho, o numero máximo de repetições nos alelos normais em ATXN 7 foi até 10 repetições CAG. O gene ATXN 7 apresenta menor variabilidade do que descrito em literatura onde podemos encontrar até 17 repetições dentro da normalidade [DAVID e cols., 1997]. Neste estudo nenhum alelo intermediário foi observado na amostra da população não afetada para AEC 2, AEC 3, AEC 6 e AEC 7. Entretanto, em estudos com famílias apresentando sintomas das ataxias espinocerebelares 1, 2, 3, 6 e 7 observam-se a presença de alelos de tamanho intermediário [PULST e cols., 1996; RANUM e cols., 1995; TAKIYAMA e cols., 1994; VAN ALFEN e cols., 2001; ZUHLKE e cols., 2002]. Provavelmente, alelos intermediários são tão raros quanto os alelos expandidos, sendo encontrados somente em famílias de afetados com a doença. Na AEC 3, estudos realizados na população assintomática Portuguesa mostraram que não houve nenhuma correlação significativa entre a freqüência da DMJ e as freqüências dos alelos normais maiores. Esta análise foi realizada levando-se em consideração o número de repetições de tamanhos pequenos, médios ou grandes (>27 repetições CAG) entre diferentes subgrupos da população [ANDRES e cols., 2002; LIMA e cols., 2005]. A maioria dos cromossomos mutados em pacientes com AEC 3 encontrados em diferentes populações do mundo apresenta um haplótipo comum e um segundo haplótipo associado a um pequeno grupo de pacientes. Dois outros haplótipos foram encontrados em poucas famílias com a doença. Todos estes haplótipos podem ter se derivado de um único haplótipo original que foi alterado em decorrência das mutações em microssatélites [GASPAR e cols., 2001]. Os estudos que associam haplótipos intragênicos com a instabilidade do tamanho das repetições 68 CAG no cromossomo normal no locus AEC 3 não sustentam a hipótese de que alelos intermediários dariam origem aos alelos expandidos [MACIEL e cols., 1999]. Os resultados do estudo atual, no qual nenhum alelo expandido ou intermediário foi encontrado na população normal para AEC 3, concorda com os estudos haplotípicos citados acima. Porém, é importante levar em consideração que o tamanho da amostra que é pequeno. A distribuição da DMJ é atribuída a uma elevada taxa de alelos mutados originalmente e não ao processo de contração gênica e conseqüente expansão. Isso poderia explicar as freqüências alélicas observadas neste trabalho. Este dado está de acordo com o modelo que postula a presença de poucos eventos mutacionais do quais a maioria dos pacientes da DMJ ao redor do mundo teria se originado [ANDRES e cols., 2002]. Além disso, acredita-se que a origem da doença de Machado Joseph no Brasil é provavelmente devida a um efeito fundador quando o gene responsável foi trazido durante a época das navegações Portuguesas. Neste tempo, por causa das perseguições religiosas, o alelo mutado causador da DMJ poderia ter se dispersado, sendo a causa da incidência elevada da doença no Brasil, nos Estados Unidos e na maior parte do mundo ocidental cols., 1996]. [HARDING, 1993; LIMPRASERT e cols., 1996; LOPES CENDES e cols., 1997; SILVEIRA e Tal fator corrobora a freqüência elevada da doença encontrada no estudo atual. Curiosamente, associada ao estudo populacional está a análise de estrutura secundária do transcrito de RNA mensageiro (RNAm) de doenças de expansão com o trinucleotídeo CAG. Em 2004, MICHLEWSKI e KRZYOSIAK, publicaram um artigo comparando a estrutura secundária de diferentes transcritos de RNAm do locus ATXN 3. Estes autores concluíram que a variante genética apresentando o nucleotídeo C (Citosina) logo após as repetições CAG confere maior instabilidade 69 destas regiões. Esta instabilidade facilita o aumento da variabilidade no locus ATXN 3. Esta rica fonte de variabilidade para evolução aumenta o risco de gerar alelos patogênicos. Esta variante C, como é chamada, está presente em humanos associada principalmente, mas não exclusivamente, a alelos mutados (isto é, expandidos). É rara nos chimpanzés e ausente em outros primatas, espécies onde não são observados alelos expandidos [LIMPRASERT e cols., 1996]. Este resultado corrobora com o pressuposto de que evolutivamente a espécie humana apresentaria taxa de alelos mutados originalmente elevada e um único haplótipo original teria sido alterado em decorrência das mutações em microssatélite [ANDRES e cols., 2002; LIMA e cols., 2005]. Entretanto, existem outros loci gênicos com características moleculares semelhantes (repetições CAG em regiões codificadoras) que apresentam mecanismos diferentes na dinâmica das repetições como o que ocorre na doença de Huntington (DH) em que alelos intermediários e expandidos são observados em população normal [COSTA e cols., 2002; RASKIN e cols., 2000]. Estes valores intermediários tenderiam a desencadear o aumento de repetições levando à expansão. Assim como na DH, existem estudos com a amostra de indivíduos clinicamente normais para DMJ em diferentes populações, que sustentam a idéia de que a presença de alelos de tamanho igual ou maior que 27 repetições CAG está correlacionada com maior prevalência da DMJ nos pacientes [TAKANO e cols., 1998]. De acordo com o presente estudo foram encontrados alelos com tamanho igual ou maior que 27 repetições de AEC 3. Há um interesse epidemiológico na freqüência destes alelos uma vez que estes parecem estar em haplótipos com alto potencial para desencadear as expansões, como se fossem reservatórios através dos quais ocorreriam mais expansões por conversão gênica [BAUER e cols., 2005; CHATTOPADHYAY e cols., 2003; DAVID e cols., 70 1997; LIMPRASERT e cols., 1996; MACIEL e cols., 1997; MITTAL e cols., 2005]. Alelos com tamanho maior que 27 repetições CAG também são observados em populações como Portugueses, Americanos, Japoneses e Africanos; populações que apresentam incidência elevada de pacientes com DMJ [KAWAGUCHI e cols., 1994; LIMPRASERT e cols., 1996; TEIVE e ARRUDA, 2004]. Nenhuma correlação poderá ser feita no locus ATXN 3 apenas com os dados encontrados neste trabalho pois: nos pacientes a DMJ é forma mais comum de ataxia e na amostra controle não existem alelos intermediários (46 a 56 repetições CAG) e foram observados alelos normais de tamanhos elevados (2,1 % dos alelos variam de 27 a 34 repetições CAG). Um estudo com uma amostra populacional maior, na qual possa haver uma estratificação por regiões com incidências distintas de DMJ, por exemplo, poderia levar a uma correlação positiva ou negativa entre a maior incidência da doença com alelos normais cujo número de repetições CAG são maiores que 27. O presente estudo fornece subsídio para compreender a origem das mutações nos loci ATXN 1, ATXN 2, ATXN 3, ATXN 6 e ATXN 7. Entretanto, um estudo haplotípico incluindo outros sítios desses loci deveria ser realizado em ambas as amostras. A freqüência dos subtipos de AEC 1, 2, 3, 6 e 7 encontrada por este estudo é semelhante às encontradas por LOPES-CENDES e cols. em 1997 (AEC 1χ2=2,38/p=0,12; AEC 2-χ2=0,24/p=0,62 e AEC 3-χ2=0,05/p=0,83); JARDIM e cols. em 2001 (AEC 3 -χ2=0,17/p=0,68 e AEC 7-χ2=3,59/p=0,06). Em SILVEIRA e cols. em 2002 as freqüências são semelhantes em AEC 3 (χ2=1,43/p=0,23) e AEC 6 (χ2=0,31/p=0,58) porém diferentes em AEC 2 (χ2=6,51/p=0,01)e AEC 7 (χ2=4,85/p=0,03) (Tabela 23). As diferenças entre esses resultados podem ser 71 devidas a distinção dos loci analisados e provavelmente por se tratar de amostra de pacientes brasileiros de diferentes estados do país. Estes resultados estão de acordo com o que se postula por diversos autores quanto à variabilidade de alelos e de freqüência dos subtipos de ataxia nas diferentes populações. Além disso, esta diferença evidencia ainda mais a importância do estudo populacional em diferentes regiões brasileiras devido à diversificação na constituição étnica do país. Tabela 23: Freqüências de ataxia espinocerebelar descritas na população brasileira Frequencias (%) Doenças Resultado Lopes – atual Cendes e cols., População Silveira e Jardim e cols., Bird, 2007 cols., 2002 2001 População 1997 Portugueses População Mundial Brasileira População e Brasileiros Brasileira (Rio (Paraná) Brasileira Grande do Sul) AEC 1 0 3 - - 6 AEC 2 5,22 6 3 - 15 AEC 3 21,74 30 63 92 21 AEC 6 0,87 - 1 - 15 AEC 7 2,61 - 1 2 5 Outros 69,56 61 32 6 38 - não analisado Através dessa análise populacional minuciosa foi possível identificar uma faixa normal de repetições CAG para indivíduos assintomáticos. Além disso, foi possível definir freqüências populacionais para os cinco genes, caracterizando melhor a população na qual o teste será empregado na rotina do laboratório. O fato de nenhum alelo intermediário ou expandido ter sido encontrado nesta população normal é relevante para o diagnóstico molecular. Tendo em vista a importância do estudo populacional, considera-se a informação da variação de alelos dentro da normalidade fornecida por este trabalho um início para compreender a dinâmica de todos os cinco loci na população do sul do Brasil. 72 A população de indivíduos controles está em equilíbrio de HW para todos os loci analisados, o que assegura a qualidade técnica deste experimento uma vez que uma amostra populacional em ambientes constantes, sem migrações intensas e com baixo índice de consangüinidade deve estar em equilíbrio de Hardy-Weinberg (HW) ou aproximar-se dele BEIGUELMAN]. [NEEL e SCHULL, 1954 apud BEIGUELMAN, 1990; LI, 1972 apud Além do equilíbrio HW os dados de heterozigosidade (H) também caracterizaram a população quanto à quantidade de heterozigotos para cada gene. Este índice é mais um dado que assegura a qualidade do experimento. A heterozigosidade (H) encontrada neste estudo para os loci AEC 1, AEC 2, AEC 3, AEC 6 e AEC 7 foi H=0,79; H=0,24; H=0,82; H=0,72; H=0,47 respectivamente. Heterozigosidade semelhante foi encontrada por SILVEIRA e cols. em 2002 para os mesmos loci (AEC 1-H=0,78; AEC 2-H=0,15; AEC 3-H=0,73; AEC 6-H=0,68; AEC 7-H=0,37). Heterozigosidade similar para o locus ATXN 3 é descrita em outras populações do mundo (H=0,84 e H=0,86 [LIMPRASERT e cols., 1996]. Este resultado associado a ausência de desvio do equilíbrio de HW indica que a região de repetições (CAG)n no gene da DMJ pode ser usada como um marcador nos estudos de genética de população [LIMA e cols., 2005]. 5.3. Metodologia para diagnóstico molecular A análise de uma amostra de indivíduos com suspeita clínica da doença permitiu definir a faixa de alelos que podem caracterizar indivíduos afetados, distinguindo esses dois grupos para utilidades diagnósticas. A diferença na média dos alelos entre as amostras de doadores de sangue e pacientes com suspeita clínica de ataxia mostrada pelo cálculo do teste “t” evidencia este fato. Encontrou-se diferença significativa nos subtipos AEC 2 e DMJ cujas médias de repetições CAG de pacientes é mais elevada (AEC 2 e DMJ). 73 Como descrito anteriormente, o início da doença nos filhos ocorre em idade mais jovem quando o sexo do genitor transmissor do alelo expandido é masculino [KLOCKGETHER e cols 2000 apud TEIVE, 2005]. à instabilidade do DNA Isto ocorre pela dinâmica das expansões devido [DEKA e cols., 1999; RICHARDS e cols.., 1992; TAKIYAMA e cols.,1995]. O teste “t” para amostras independentes comparando a idade média de início dos sintomas entre os sexos dos genitores transmissores da doença mostra diferenças significativas entre o sexo do genitor que transmite o gene. O resultado mostra que o início da doença nos filhos ocorre em idade mais jovem quando o sexo do genitor transmissor do alelo expandido é masculino. Este dado está de acordo com o que se observa na literatura. Os valores de especificidade, sensibilidade, valor preditivo positivo (VPP) e valor preditivo negativo (VPN) foram obtidos comparando o resultado molecular com o diagnóstico clínico. Devido à sobreposição dos fenótipos entre os diferentes subtipos de ataxia o diagnóstico baseado somente no exame clínico torna-se muito difícil. Este painel para os 5 genes responsáveis em causar ataxia espinocerebelar 1, 2, 3, 6 e 7 descrito neste estudo, pôde distinguir os verdadeiros resultados negativos (especificidade) em 78,75 % e os verdadeiros resultados positivos (sensibilidade) em 80,00 % dos pacientes analisados clinicamente. Este resultado de sensibilidade significa que, embora 20,00 % dos pacientes tenham ataxia clinicamente, provavelmente a causa não se deva a nenhum dos 5 genes analisados. Nestes casos, outros testes genéticos, assim como o suporte do clínico para a confirmação do diagnóstico serão substanciais para conclusão diagnóstica. O VPP indica que 62,22% dos casos com diagnóstico clínico deverão ser confirmados pelo diagnóstico molecular para um dos cinco subtipos analisados. O VPN indica que 90,00 % dos pacientes com suspeita clínica de outras ataxias ou com diagnóstico a 74 esclarecer apresentarão resultado molecular negativo para os subtipos analisados. A relevância deste resultado é que 10,00 % dos pacientes foram detectados e diferenciados somente pelo diagnóstico molecular. Qualquer resultado molecular positivo evidencia que a causa da ataxia é de fato o subtipo no qual o alelo expandido foi detectado (alta especificidade e o VPP) [GUEDES e GUEDES, 1988; SOUNIS, 1975; VIEIRA, 2003]. Outro fator importante é que tecnicamente pode haver amplificação preferencial do alelo normal [LARSEN ROSENBLUM e cols., 1997]. e cols., 1997; LE e cols., 1997; DORSCHNER e cols., 2002; Este fato pode não evidenciar a expansão em alguns pacientes principalmente nos casos de AEC 2 e AEC 7 juvenil cujo número de repetições pode ser bastante elevado [SCHÖLS e cols., 2004] fugindo a linearidade e a sensibilidade do método. Entretanto, a metodologia utilizada no presente estudo definiu os protocolos com alelos normais e alelos expandidos. Este fato associado aos resultados populacionais indicam que provavelmente a maior parte dos alelos responsáveis em causar o fenótipo de ataxia espinocerebelar 1, 2, 3, 6 ou 7 pôde ser detectado. No total dos 115 pacientes estudados, o diagnóstico molecular foi confirmado em 35 pacientes sendo seis para AEC 2, 25 para AEC 3, um para AEC 6 e três para AEC 7. Os demais 80 pacientes não apresentaram alelos expandidos nos loci analisados. Dos 115 pacientes, 56 foram analisados clinicamente e apresentaram sobreposição de sintomas clínicos, como se espera em doenças neurodegenerativas, devido à heterogeneidade clínica. O diagnóstico molecular foi confirmado pela eletroforese de capilar em 22 pacientes dos 56 (cinco pacientes para AEC 2, quatorze pacientes para DMJ e três pacientes para AEC 7) representando 62,86% 75 do total de alelos expandidos encontrados na amostra de 115 indivíduos com suspeita clínica. Apesar do número pequenos de indivíduos com expansão detectada em AEC 2 (cinco) a mutação foi transmitida em 80,00% (quatro) dos casos pela mãe. Portanto, para diferenciar indivíduos afetados pelos subtipos estudados e indivíduos normais, podemos recorrer ao resultado do presente trabalho que definiu a faixa de normalidade para cada um dos subtipos e encontrou alelos expandidos em pacientes. Pacientes que apresentarem clinicamente sintomas gerais de ataxia, em alguns casos no início da doença onde há grande dificuldade para o clínico distinguir os subtipos, este diagnóstico será importante principalmente quando terapêuticamente se tornar viável o tratamento específico para cada um dos subtipos. O diagnóstico baseado somente nos achados clínicos é bastante controverso e a metodologia mostrou-se útil tanto na distinção entre os subtipos quanto na confirmação ou exclusão de outros casos com sintomas semelhantes. Além do que foi descrito em literatura, este trabalho fornece dados próprios de investigação úteis para a população Paranaense não só em âmbito de diagnóstico molecular e diferencial, mas também para serem usados em estudos de evolução molecular dos genes em questão. 76 VI. CONCLUSÕES 77 6.1. Principal O genótipo das repetições CAG nos genes ATXN 1, 2, 3, 6 e 7 em indivíduos da população normal e de pacientes foi determinado sendo possível discriminar clinicamente indivíduos normais e afetados para o diagnóstico molecular, distinguindo geneticamente os cinco subtipos de ataxia. 6.2. Secundárias: Originais: a. Na população normal alelos ainda não descritos em literatura foram encontrados e a freqüência populacional dos alelos tão bem como a faixa de normalidade foi definida para população paranaense b. As distribuições genotípicas estão de acordo com Equilíbrio de HardyWeinberg evidenciando a qualidade técnica, além de demonstrar estabilidade alélica na população não afetada. c. Foi possível averiguar a freqüência dos alelos de ataxias no Paraná em indivíduos não aparentados com suspeita clínica. d. A ausência de alelos intermediários na população normal pode ser mais um indício que, evolutivamente, os alelos mutados não decorrem do processo de conversão e contração gênica nestes loci. Confirmatórias: a. Foi possível definir os alelos de cada um dos cinco loci a partir do tamanho em pares de base (pb). b. Alelos expandidos foram detectados em AEC 2, AEC 3, AEC 6 e AEC 7 na amostra de pacientes. A freqüência alélica e da incidência da doença pôde ser calculada. 78 VII. REFERÊNCIAS BIBLIOGRÁFICAS 79 ALBANO LMJ. Genética clínica e molecular das doenças neurológicas com mutações dinâmicas. 1a ed. 2000;97-140 ANDRES A, LAO O, SOLDEVILA M, CALAFELL F, BERTRANPETIT J. Dynamics of CAG repeat loci revealed by the analysis of their variability. Hum Mutat 2002;21:61-70. ANDREW SE, GOLDBERG YP, HAYDEN MR. Rethinking Genotype and phenotype correlations in polyglutamine expansion disorders. Hum Molec Genet 1997;6:2005-2010. ARRUDA WO, TEIVE HA. Hereditary cerebellar ataxias: from hammer to genetics. Arq Neuropsiquiatr 1997;55:666-676 BAKER SK, TARNOPOLSKY MA. Targeting cellular energy production in neurological disorders. Expert Opin Investig Drugs 2003;12:1655-1679. BANFI S, SERVADIO A, CHUNG MY, KWIATKOWSKI TJ JR, MCCALL AE, DUVICK LA, SHEN Y, ROTH EJ, ORR HT, ZOGHBI HY. Identification and characterization of the gene causing type 1 spinocerebellar ataxia. Nat Genet 1994;7:513-520. BANFI S, SERVADIO A, CHUNG MY, CAPOZZOLI F, DUVICK LA, ELDE R, ZOGHBI HY, ORR HT. Cloning and developmental expression analysis of the murine homolog of the spinocerebellar ataxia type 1 gene (SCA1). Hum. Molec Genet 1996;5:33-40. BAUER PO, ZUMROVA A, MATOSKA V, MARIKOVA S, KRILOVA A, BODAY A, SINGH B, GOTEZ P. Absence of spinocerebellar ataxia type3/Machado-Joseph disease within ataxia patients in the Czech population. Europ J Neur 2005;12:851-857. BEHRENDS C, LANGER CA, BOTEVA R, BÖTTCHER UM, STEMP MJ, SCHAFFAR G, RAO BV, GIESE A, KRETZSCHMAR H, SIEGERS K, HARTL FU. Chaperonin TRiC promotes the assembly of polyQ expansion proteins into nontoxic oligomers. Mol Cell. 2006;15;23:887-97 BEIGUELMAN B. Dinâmica dos genes nas famílias e nas populações. 2 ed, 1990, p.203-207; 219-226. BENOMAR A, LE GUERN E, DURR A, OUHABI H, STEVANIN G, YAHYAOUI M, CHKILI T, AGID Y, BRICE A. Autosomal-dominant cerebellar ataxia with retinal degeneration (ADCA type II) is genetically different from ADCA type I. Ann. Neurol 1994;35:439-444. BERKE SJ, CHAI Y, MARRS GL, WEN H, PAULSON HL. Defining the role of ubiquitin-interacting motifs in the polyglutamine disease protein, ataxin-3. J Biol Chem 2005;36:32026-32034. 80 BROOK JD, MCCURRACH ME, HARLEY HG, BUCKLER AJ, CHURCH D, ABURATANI H, HUNTER K, STANTON VP, TIRION JP, HUDSON T, SOHN R, ZEMELMEN B, SNELL RG, RUNDLE SA, CROW S, DAVIES J, SHELBOURNE P, BUXTON J, JONES C, JUVONEN V, JOHNSON K, HARPER PS, SHAW DJ, HOUSMAN DE. Molecular basis of myotonic dystrophy: expansion of a trinucleotide (CTG) repeat 3’ end of a transcript encoding a protein kinase family member. Cell 1992;68:799-808. BRUSCO A, GELLERA C, CAGNOLI C, SALUTO A, CASTUCCI A, MICHIELOTTO C, FETONI V, MARIOTTI C, MIGONE N, DI DONATO S, TARONI F. Molecular genetics of hereditary spinocerebellar ataxia: mutation analysis of spinocerebellar ataxia genes and CAG/CTG repeat expansion detection in 225 Italian families. Arch Neurol 2004;61:727-733. BULONE D, MASINO L, THOMAS DJ, SAN BIAGIO PL, PASTORE A. The interplay between PolyQ and protein context delays aggregation by forming a reservoir of protofibrils. PLoS ONE 2006; 27:111 CANCEL G, DURR A, DIDIERJEAN O, IMBERT G, BURK K, LEZIN A, BELAL S, BENOMAR A, ABADA-BENDIB M, VIAL C, GUIMARAES J, CHNEIWEISS H, STEVANIN G, YVERT G, ABBAS N, SAUDOU F, LEBRE AS, YAHYAOUI M, HENTATI F, VERNANT JC, KLOCKGETHER T, MANDEL JL, AGID Y, BRICE A. Molecular and clinical correlations in spinocerebellar ataxia 2: a study of 32 families. Hum Molec Genet 1997;6:709715. CHATTOPADHYAY B, BASU P, GANGOPADHYAY PK, MUKHERJEE SC, SINHA KK, CHAKRABORTY A, ROY T, ROYCHOUDHURY S, MAJUMDER PP, BHATTACHARYYA NP. Variation of CAG repeats and two intragenic polymorphisms at SCA3 locus among Machado–Joseph disease/SCA3 patients and diverse normal populations from eastern India. Acta Neurol Scand 2003;108:407–414. CHEN YW, ALLEN MD, VEPRINTSEV DB, LOWE J, BYCROFT M. The structure of the AXH domain of spinocerebellar ataxin-1. J Biol Chem 2004;279:3758-3765. COLOMER GOULD VF. Mouse models of Machado-Joseph disease and other polyglutamine spinocerebellar ataxias. NeuroRx 2005;2:480-483. COSTA MC, GUIMARÃES L, FERREIRINHA F, SOUSA A, MACIEL P, SEQUEIROS J. Study of the normal CAG tract at the Huntington disease locus in the Portuguese population. Eur J Hum Genet 2002;10:263-264. COUTINHO P E ANDRADE C. Autosomal dominant system degeneration in Portuguese families of the Azorean islands: a new genetic disorder involving cerebellar, pyramidal, extrapyramidal and spinal cord motor functions. Neurology 1978;28:703-709. 81 COUTINHO P, GUIMARÃES A, SCARAVILLI F. The pathology of MachadoJoseph disease: report of a possible homozigous case. Acta Neuropath (Berlim) 1982;58:48-54. CUMMINGS CJ E ZOGHBI HY. Fourteen and counting: unraveling trinucleotide repeat diseases. Hum Mol Genet 2000;9:909-916. DAVID G, ABBAS N, STEVANIN G, DURR A, YVERT G, CANCEL G, WEBER C, IMBERT G, SAUDOU F, ANTONIOU E, DRABKIN H, GEMMILL R, GIUNTI P, BENOMAR A, WOOD N, RUBERG M, AGID Y, MANDEL JL, BRICE A. Cloning of the SCA7 gene reveals a highly unstable CAG repeat expansion. Nature Genet 1997;17:65-70. DAVIES J, YAMAGATA H, SHELBOURNE P, BUXTON J, OGIHARA T, NOKELAINEN P, NAKAGAWA M, WILLIAMSON R, JOHNSON K, MIKIT. Comparison of the myotonic dystrophy associated CTG repeat in European and Japanese populations. J Med Genet 1992;29:766-769. DE CHIARA C, MENON RP, DAL PIAZ F, CALDER L, PASTORE A. Polyglutamine is not all: the functional role of the AXH domain in the ataxin-1 protein. J Mol Biol 2005;354:883-93. DEKA R, MIKI T, YIN SJ, MCGARVEY ST, SHRIVER MD, BUNKER CH, RASKIN S, HUNDRIESER J, FERRELL RE, CHAKRABORTY R. Normal CAG repeat variation at the DRPLA locus in world populations. Am J Hum Genet 1995;57:508-511. DEKA R, GUANGYUN S, SMELSER D, ZHONG Y, KIMMEL M, CHAKRABORTY R. Rate and directionality of mutations and effects of allele size constraints at anonymous, gene-associated, and disease-causing trinucleotide loci. Mol Biol Evol 1999;9:1166-1177. DIRIONG S, LORY P, WILLIAMS ME, ELLIS SB, HARPOLD MM, TAVIAUX S. Chromosomal localization of the human genes for alpha-1A, alpha-1B, and alpha-1E voltage-dependent Ca(2+) channel subunits. Genomics 1995;30:605609. DON RH, COX PT, WAINWRIGHT BJ, BAKER K, MATTICK JS. ‘Touchdown’ PCR to circumvent spurious priming during gene amplification. Nucleic Acids Res 1991;19:4008. DORSCHNER MO, BARDEN D, STEPHENS K. Diagnosis of five spinocerebellar ataxia disorders by multiplex amplification and capillary electrophoresis. J Mol Diagn 2002;4:108-113. DRYER SE, LHUILLIER L, CAMERON JS, MARTIN-CARABALLO M. Expression of K(Ca) channels in identified populations of developing vertebrate neurons: role of neurotrophic factors and activity. J Physiol 2003;97:49-58. 82 EVERT BO, WULLNER U, KLOCKGETHER T. Cell death in polyglutamine disease. Cell Tissue Res 2000;301:189-204. FARAH MH. RNAi silencing in mouse models of neurodegenerative diseases. Curr Drug Deliv 2007;2:161-167. FLANIGAN K, GARDNER K, ALDERSON K, GALSTER B, OTTERUD B, LEPPERT MF, KAPLAN C, PTACEK LJ. Autosomal dominant spinocerebellar ataxia with sensory axonal neuropathy (SCA4): clinical description and genetic localization to chromosome 16q22.1. Am J Hum Genet 1996;392-399. FUJIGASAKI H, MARTIN JJ, DE DEYN PP, CAMUZAT A, DEFFOND D, STEVANIN G, DERMAUT B, VAN BROECKHOVEN C, DÜRR A, BRICE A. CAG repeat exapansion in the TATA box-binding protein gene causes autosomal dominant cerebellar ataxia. Brain 2001;124:1939-1947. GASPAR C, LOPES-CENDES I, HAYES S, GOTO J, ARVIDSSON K, DIAS A, SILVEIRA I, MACIEL P, COUTINHO P, LIMA M, ZHOU YX, SOONG BW, WATANABE M, GIUNTI P, STEVANIN G, RIESS O, SASAKI H, HSIEH M, NICHOLSON GA, BRUNT E, HIGGINS JJ, LAURITZAN M, TRAMBJAERG L, VOLPINI V, WOOD N, RANUM L, TSUJI S, BRICE A, SEQUEIROS J, ROULEAU GA. Ancestral origins of the Machado-Joseph disease mutation: A worldwide haplotype study. Am J Hum Genet 2001;68:523-528. GISPERT S, TWELLS R, OROZCO G, BRICE A, WEBER J, HEREDERO L, SCHEUFLER K, RILEY B, ALLOTEY R, NOTHERS C, HILLERMANN R, LUNKES A, KHATI C, STEVANIN G, HERNANDEZ A, MAGARINO C, KLOCKGETHER T, DURR A, CHNEIWEISS H, ENCZMANN J, FARRALL M, BECKMANN J, MULLAN M, WERNET P, AGID Y, FREUND HJ, WILLIAMSON R, AUBURGER G, CHAMBERLAIN S. Chromosomal assignment of the second locus for autosomal dominant cerebellar ataxia (SCA2) to chromosome 12q23-24.1. Nature Genet 1993;4:295-299. GIUNTI P, SWEENEY MG, HARDING AE. Detection of the Machado-Joseph disease/spinocerebellar ataxia three trinucleotide repeat expansion in families with autosomal dominant motor disorders, including the Drew family of Walworth. Brain 1995;118:1077-1085. GOLDMAN A, RAMSAY M, JENKINS T. Absence of myotonic dystrophy in southern African Negroids is associated with significantly lower number of CTG trinucleotide repeats. J Med Genet 1994;31:37-40. GUEDES MLS, GUEDES JS. Bioestatística - Para profissionais de saúde. 1ª Ed. Ao livro técnico S.A. RJ, 1988. HARDING AE. Classification of the hereditary ataxias and paraplegias. Lancet 1983;1151-1155. 83 HARDING AE. Clinical features and classification of inherited ataxia. Adv Neurology 1993;61:1-14. HARTL D. Population genetics. Editon Française 1998;5-34 HERMAN-BERT A, STEVANIN G, NETTER JC, RASCOL O, BRASSAT D, CALVAS P, CAMUZAT A, YUAN Q, SCHALLING M, DURR A, BRICE A. Mapping of spinocerebellar ataxia 13 to chromosome 19q13.3-q13.4 in a family with autosomal dominant cerebellar ataxia and mental retardation. Am J Hum Genet 2000;67:229-235. HOLMES SE, O'HEARN EE, MCINNIS MG, GORELICK-FELDMAN DA, KLEIDERLEIN JJ, CALLAHAN C, KWAK NG, INGERSOLL-ASHWORTH RG, SHERR M, SUMNER AJ, SHARP AH, ANANTH U, SELTZER WK, BOSS MA, VIERIA-SAECKER AM, EPPLEN JT, RIESS O, ROSS CA, MARGOLIS RL. Expansion of a novel CAG trinucleotide repeat in the 5-prime region of PPP2R2B is associated with SCA12. Nature Genet 1999;23:391-392. HUNTER JM, LESORT M, JOHNSON GV. Ubiquitin-proteasome system alterations in a striatal cell model of Huntington's disease. J Neurosci Res 2007;85(8):1774-1788. HUYNH DP, YANG HT, VAKHARIA H, NGUYEN D, PULST SM. Expansion of the poly Q repeat in ataxin-2 alters its Golgi localization, disrupts the Golgi complex and causes cell death. Hum Molec Genet 2003;12:1485-1496. ICHIKAWA Y, GOTO J, HATTORI M, TOYODA A, ISHII K, JEONG SY, HASHIDA H, MASUDA N, OGATA K, KASAI F, HIRAI M, MACIEL P, ROULEAU GA, SAKAKI Y, KANAZAWA I. The genomic structure and expression of MJD, the Machado-Joseph disease gene. J Hum Genet. 2001;46:413-22. IKEDA H, YAMAGUCHI M, SUGAI S, AZE Y, NARUMIYA S, KAKIZUKA A. Expanded polyglutamine in the Machado-Joseph disease protein induces cell death in vitro and in vivo. Nature Genet 1996;13:196-202. IKEDA Y, DICK KA, WEATHERSPOON MR, GINCEL D, ARMBRUST KR, DALTON JC, STEVANIN G, DURR A, ZUHLKE C, BURK K, CLARK HB, BRICE A, ROTHSTEIN JD, SCHUT LJ, DAY JW, RANUM LP. Spectrin mutations cause spinocerebellar ataxia type 5. Nat Genet 2006;38:184-190. IMBERT G, SAUDOU F, YVERT G, DEVYS D, TROTTIER Y, GARNIER JM, WEBER C, MANDEL JL, CANCEL G, ABBAS N, DURR A, DIDIERJEAN O, STEVANIN G, AGID Y, BRICE A. Cloning of the gene for spinocerebellar ataxia 2 reveals a locus with high sensitivity to expanded CAG/glutamine repeats. Nature Genet 1996;14:285-291. IRWIN S, VANDELFT M, PINCHEV D, HOWELL JL, GRACZYK J, ORR HT, TRUANT. RNA association and nucleocytoplasmic shuttling by ataxin-1. J Cell Sci 2005;118:233-242. 84 ISHIKAWA K, TANAKA H, SAITO M, OHKOSHI N, FUJITA T, YOSHIZAWA K, IKEUCHI T, WATANABE M, HAYASHI A, TAKIYAMA Y, NISHIZAWA M, NAKANO I, MATSUBAYASHI K, MIWA M, SHOJI S, KANAZAWA I, TSUJI S, MIZUSAWA H. Japanese families with autosomal dominant pure cerebellar ataxia map to chromosome 19p13.1-p13.2 and are strongly associated with mild CAG expansions in the spinocerebellar ataxia type 6 gene in chromosome 19p13.1. Am J Hum Genet 1997;61:336-346. ISHIKAWA K, FUJIGASAKI H, SAEGUSA H, OHWADA K, FUJITA T, IWAMOTO H, KOMATSUZAKI Y, TORU S, TORIYAMA H, WATANABE M, OHKOSHI N, SHOJI S, KANAZAWA I, TANABE T, MIZUSAWA H. Abundant expression and cytoplasmic aggregations of alpha-1A voltagedependent calcium channel protein associated with neurodegeneration in spinocerebellar ataxia type 6. Hum Molec Genet 1999;8:1185-1193. JARDIM LB, SILVEIRA I, PEREIRA ML. A survey of spinocerebellar ataxia in south Brazil - 66 new cases with Machado-Joseph disease, SCA 7, SCA 8, or unidentified disease-causing mutations. J Neurol 2001;248:870-876. JURKAT-ROTT K, LEHMANN-HORN F. The impact of splice isoforms on voltage-gated calcium channel alpha1 subunits. J Physiol 2004;1,554:609-619. KAMEYA T, ABE K, AOKI M, SAHARA M, TOBITA M, KONNO H, ITOYAMA Y. Analysis of spinocerebelar ataxia type 1 (SCA1) - related CAG trinucleotide expansion in Japan. Neurology 1995;45:1587-1594. KAWAGUCHI Y, OKAMOTO T, TANIWAKI M, AIZAWA M, INOUE M, KATAYAMA S, KAWAKAMI H, NAKAMURA S, NISHIMURA M, AKIGUCHI I, KIMURA J, NARUMIYA S, KAKIZUKA A. CAG expansions in a novel gene for Machado-Joseph disease at chromosome 14q32.1. Nature Genet 1994;8:221-228. KNIGHT MA, KENNERSON ML, ANNEY RJ, MATSUURA T, NICHOLSON GA, SALIMI-TARI P, GARDNER RJM, STOREY E, FORREST SM. Spinocerebellar ataxia type 15 (SCA15) maps to 3p24.2-3pter: exclusion of the ITPR1 gene, the human orthologue of an ataxic mouse mutant. Neurobiol Dis 2003;13:147-157. KNIGHT MA, GARDNER RJ, BAHLO M, MATSUURA T, DIXON JA, FORREST SM, STOREY E. Dominantly inherited ataxia and dysphonia with dentate calcification: spinocerebellar ataxia type 20. Brain 2004;127:11721181. KOMATSU M, WAGURI S, CHIBA T, MURATA S, IWATA J, TANIDA I, UENO T, KOIKE M, UCHIYAMA Y, KOMINAMI E, TANAKA K. Loss of autophagy in the central nervous system causes neurodegeneration in mice. Nature. 2006;15;441:819-820. 85 KOOB MD, MOSELEY ML, SCHUT LJ, BENZOW KA, BIRD TD, DAY JW, RANUM LP. An untranslated CTG expansion causes a novel form of spinocerebellar ataxia (SCA 8). Nat Genet 1999;21:379-384. LA SPADA AR, WILSON EM, LUBAHN DB, HARDING AE, FISCHBECK KH. Androgen receptor gene mutations in X-linked spinal and bulbar muscular atrophy. Nature 1991;52:77-79 LA SPADA AR, TAYLOR JP. Polyglutamines placed into context. Neuron 2003;38:681-684. LAM YC, BOWMAN AB, JAFAR-NEJAD P, LIM J, RICHMAN R, FRYER JD, HYUN ED, DUVICK LA, ORR HT, BOTAS J, ZOGHBI HY. ATAXIN-1 interacts with the repressor capicua in its native complex to cause SCA1 neuropathology. Cell 2006;29:1299-1300. LARSEN LA, GRONSKOV K, NORGAARD-PEDERSEN B, BRONDUMNIELSEN K, HASHOLT L, VUUST J. High-throughput analysis of fragile X (CGG)n alleles in the normal and premutation range by PCR amplification and automated capillary electrophoresis. Hum Genet 1997;100:564-568. LATOUCHE M, LASBLEIZ C, MARTIN E, MONNIER V, DEBEIR T, MOUATT-PRIGENT A, MURIEL MP, MOREL L, RUBERG M, BRICE A, STEVANIN G, TRICOIRE H. A conditional pan-neuronal Drosophila model of spinocerebellar ataxia 7 with a reversible adult phenotype suitable for identifying modifier genes. J Neurosci 2007;27:2483-2492. LE H, FUNG D, TRENT RJ. Applications of capillary electrophoresis in DNA mutation analysis of genetic disorders. Mol Pathol 1997;50:261-265. LEBRE AS, JAMOT L, TAKAHASHI J, SPASSKEY N, LEPRINCE C, RAVISE N, ZANDER C, FUJIGASAKI H, KUSSEL-ANDERMANN P, DUYCKAERTS C, CAMONIS JH, BRICE A. Ataxin-7 interacts with a Cblassociated protein that it recruits into neuronal intranuclear inclusions. Hum Molec Genet 2001;10:1201-1213. LIMA M, COSTA MC, MONTIEL R, FERRO A, SANTOS C, SILVA C, BETTENCOURT C, SOUSA A, SEQUEIROS J, COUTINHO P, MACIEL P. Population genetics of wild-type CAG repeats in the Machado-Joseph disease gene in Portugal. Hum Hered 2005;60:156-163. LIMPRASERT P, NOURI N, HEYMAN RA, NOPPARATANA C, KAMONSILP M, DEININGER PL, KEATS BJ. Analysis of CAG repeat of the Machado– Joseph gene in human, chimpanzee and monkey populations: a variant nucleotide is associated with the number of CAG repeats. Hum Mol Genet 1996;5:207–213. LITTARRU GP, TIANO L. Clinical aspects of coenzyme Q10: an update. Curr Opin Clin Nutr Metab Care. 2005;8:641-646. 86 LITT M, KRAMER P, BROWNE D, GANCHER S, BRUNT ER, ROOT D, PHROMCHOTIKUL T, DUBAY CJ, NUTT J. A gene for episodic ataxia/myokymia maps to chromosome 12p13. Am J Hum Genet 1994;55:702709. LOPES-CENDES I, TEIVE H, CALCAGNOTTO ME, DA COSTA JC, CARDOSO F, VIANA E, MACIEL JA, RADVANY J, ARRUDA WO, TREVISOL-BITTENCOURT PC, ROSA NETO P, SILVEIRA I, STEINER CE, PINTO JUNIOR W, SANTOS AS, CORREA NETO Y, WERNECK LC, ARAUJO AQ, CARAKUSHANSKY G, MELLO LR, JARDIM LB, ROULEAU GA. Frequency of the different mutations causing spinocerebellar ataxia (SCA1, SCA2, MJD/SCA3 and DRPLA) in a large group of Brazilian patients. Arq Neuropsiquiatr 1997;55:519-529. MACIEL P, LOPES-CENDES I, KISH S, SEQUEIROS J, ROULEAU GA. Mosaicism of the CAG repeat in cns tissue in relation to age at death in spinocerebellar ataxia type I and Machado-Joseph disease patients. Am J Hum Genet 1997;60:993-996 MACIEL P, GASPAR C, GUIMARAES L, GOTO J, LOPES-CENDES I, HAYES S, ARVIDSSON K, DIAS A, SEQUEIROS J, SOUSA A, ROULEAU GA. Study of three intragenic polymorphisms in the Machado–Joseph disease gene (MJD1) in relation to genetic instability of the (CAG)n tract. Eur J Hum Genet 1999;7:147–156. MARUYAMA H, IZUMI Y, MORINO H, ODA M, TOJI H, NAKAMURA S, KAWAKAMI H. Difference in disease-free survival curve and regional distribution according to subtype of spinocerebellar ataxia: a study of 1,286 Japanese patients. Am J Med Genet 2002;114:578-583. MATILLA T, VOLPINI V, GENIS D, ROSELL J, CORRAL J, DAVALOS A, MOLINS A, ESTIVILL X. Presymptomatic analysis of spinocerebellar ataxia type 1 (SCA1) via the expansion of the SCA1 CAG-repeat in a large pedigree displaying anticipation and parental male bias. Hum Molec Genet 1993;2:21232128. MATSUURA T, YAMAGATA T, BURGESS DL, RASMUSSEN A, GREWAL RP, WATASE K, KHAJAVI M, MCCALL AE, DAVIS CF, ZU L, ACHARI M, PULST SM, ALONSO E, NOEBELS JL, NELSON DL, ZOGHBI HY, ASHIZAWA T. Large expansion of the ATTCT pentanucleotide repeat in spinocerebellar ataxia type 10. Nature Genet 2000;26:191-194. MCMAHON SJ, PRAY-GRANT MG, SCHIELTZ D, YATES JR 3RD, GRANT PA. Polyglutamine-expanded spinocerebellar ataxia-7 protein disrupts normal SAGA and SLIK histone acetyltransferase activity. Proc Natl Acad Sci 2005;14,102:8478-8482. MICHALIK A, DEL-FAVERO J, MAUGER C, LOFGREN A, VAN BROECKHOVEN C. Genomic organization of the spinocerebellar ataxia type 87 7 (SCA7) gene responsible for autosomal dominant cerebellar ataxia with retinal degeneration. Hum Genet 1999;105:410-417. MICHALIK A E BROECKHOVEN C. Pathogenesis of polyglutamine disorders: aggregation revisited. Hum Mol Genet 2003;12:173-186. MICHLEWSKI G E KRZYOSIAK WJ. Molecular architecture of CAG repeats in human disease related transcripts. J Mol Biol 2004;340:665-679. MITTAL U, SRIVASTAVA AK, JAIN S, MUKERJI M. Founder haplotype for Machado-Joseph disease in the Indian population. Arch Neurol 2005;62:637640. MIURA S, SHIBATA H, FURUYA H, OHYAGI Y, OSOEGAWA M, MIYOSHI Y, MATSUNAGA H, SHIBATA A, MATSUMOTO N, IWAKI A, TANIWAKI T, KIKUCHI H, KIRA J, FUKUMAKI Y. The contactin 4 gene locus at 3p26 is a candidate gene of SCA16. Neurology 2006;67:1236-1241. MONTERO R, PINEDA M, ARACIL A, VILASECA MA, BRIONES P, SÁNCHEZ-ALCÁZAR JA, NAVAS P, ARTUCH R. Clinical, biochemical and molecular aspects of cerebellar ataxia and Coenzyme Q10 deficiency. Cerebellum 2007;6:118-122. MOSELEY ML, BENZOW KA, SCHUT LJ, BIRD TD, GOMEZ CM, BARKHAUS PE, BLINDAUER KA, LABUDA M, PANDOLFO M, KOOB MD, RANUM LP. Incidence of dominant spinocerebellar ataxias and Friedreich triplet repeats among 361 ataxia families. Neurology 1998;51:16661671. NAGAFUCHI S, YANAGISAWA H, SATO K, SHIRAYAMA T, OHSAKI E, BUNDO M, TAKEDA T, TADOKORO K, KONDO I, MURAYAMA N, TANAKA Y, KIKUSHIMA H, UMINO K, KUROSAWA H, FURUKAWA T, NIHEI K, INOUE T, SANO A, KOMURE O, TAKAHASHI M, YOSHIZAWA T, KANAZAWA I, YAMADA M. Dentatorubral and pallidoluysian atrophy expansion of an unstable CAG trinucleotide on chromosome 12p. Nature Genet 1994;6:14-18. NAGAI Y, INUI T, POPIEL HA, FUJIKAKE N, HASEGAWA K, URADE Y, GOTO Y, NAIKI H, TODA T. A toxic monomeric conformer of the polyglutamine protein. Nat Struct Mol Biol 2007;14:332-340. NAKAMURA K, JEONG SY, UCHIHARA T, ANNO M, NAGASHIMA K, NAGASHIMA T, IKEDA S, TSUJI S, KANAZAWA I. SCA17, a novel autosomal dominant cerebellar ataxia caused by an expanded polyglutamine in TATA-binding protein. Hum Mol Genet 2001;10:1441-1448. NICASTRO G, MENON RP, MASINO L, KNOWLES PP, MCDONALD NQ, PASTORE A. The solution structure of the Josephin domain of ataxin-3: Structural determinants for molecular recognition Proc Natl Acad Sci 2005; 102:10493-10498. 88 NICASTRO G, HABECK M, MASINO L, SVERGUN DI, PASTORE A. Structure validation of the Josephin domain of ataxin-3: conclusive evidence for an open conformation. J Biomol 2006;36:267-277. OKAZAWA H, RICH T, CHANG A, LIN X, WARAGAI M, KAJIKAWA M, ENOKIDO Y, KOMURO A, KATO S, SHIBATA M, HATANAKA H, MOURADIAN MM, SUDOL M, KANAZAWA I. Interaction between mutant ataxin-1 and PQBP-1 affects transcription and cell death. Neuron 2002;34:701713. OPHOFF RA, TERWINDT GM, VERGOUWE MN, VAN EIJK R, OEFNER PJ, HOFFMAN SM, LAMERDIN JE, MOHRENWEISER HW, BULMAN DE, FERRARI M, HAAN J, LINDHOUT D, VAN OMMEN GJ, HOFKER MH, FERRARI MD, FRANTS RR. Familial hemiplegic migraine and episodic ataxia type-2 are caused by mutations in the Ca+2 channel gene CACNL1A4. Cell 1996;87:543-552. ORR HT, CHUNG MY, BANFI S, KWIATKWSKI TJ, SERVADIO AL, BEAUDET AL, MCCALL AE, DUVICK LA, RANUM LP, ZOGHBI HY. Expansion of an unstable trinucleotide CAG repeat in spinocerebellar ataxia type I. Nat Genet 1993;4:221-226. PARK Y, HONG S, KIM SJ, KANG S. Proteasome function is inhibited by polyglutamine-expanded ataxin-1, the SCA1 gene product. Mol Cells. 2005;28,19:23-30. PAULSON HL, SUBRAMONY SH. Ataxia: The whats and hows of acquired and genetic disease. American Academy of Neurology, Education Program Syllabus, Denver, 2002. PEREZ MK, PAULSON HL, PITTMAN RN. Ataxin-3 with an altered conformation that exposes the polyglutamine domain is associated with the nuclear matrix. Hum Molec Genet 1999;8:2377-2385. PRESTES PR, SARAIVA-PEREIRA ML, SILVEIRA I, SEQUEIROS J, JARDIM LB. Machado-Joseph Disease enhances genetic fitness: A comparison between affected and unaffected women and between MJD and the general population. Ann Hum Genet 2007; 7. PULST SM, NECHIPORUK A, NECHIPORUK T, GISPERT S, CHEN XN, LOPES-CENDES I, PEARLMAN S, STARKMAN S, OROZCO-DIAZ G, LUNKES A, DEJONG P, ROULEAU GA, AUBURGER G, KORENBERG JR, FIGUEROA C, SAHBA S. Moderate expansion of a normally biallelic trinucleotide repeat in spinocerebellar ataxia type 2. Nature Genet 1996;14:269276. 89 RALSER M, ALBRECHT M, NONHOFF U, LENGAUER T, LEHRACH H, KROBITSCH S. An integrative approach to gain insights into the cellular function of human ataxin-2. J Mol Biol 2005;11,346:203-14. RANUM LP, LUNDGREN JK, SCHUT LJ. Spinocerebellar ataxia type1 and Machado-Joseph disease:incidence of CAG expansions among adult-onset ataxia patients from 311 families with dominant, recessive, or sporadic ataxia. Am J Hum Genet 1995;57:603-608. RASKIN S, ALLAN N, TEIVE HA, CARDOSO F, HADDAD MS, LEVI G, BOY R, LERENA JUNIOR J, SOTOMAIOR VS, JANZEN-DÜCK M, JARDIM LB, FELLANDER FR, ANDRADE LA. Huntington disease: DNA analysis in Brazilian population. Arq Neuropsiquiatr 2000;58:977-985 RAYMOND M E ROUSSET F. GENEPOP (version 1.2): Population genetics software for exact tests and ecumenicism. J Hered 1995;86:248-249. RICH SS, WILKIE P, SCHUT L, VANCE G, ORR HT. Spinocerebellar ataxia: localization of an autosomal dominant locus between 2 markers on human chromosome 6. Am J Hum Genet 1987;41:524-531. RICHARDS RJ, SUTHERLAND GR. Dynamic Mutations: A new class of Mutations Causing Human Disease. Cell 1992; 70:709-712. RODRIGUES AJ, COPPOLA G, SANTOS C, COSTA C, AILION M, SEQUEIROS J, GESCHWIND DH, MACIEL P. Functional genomics and biochemical characterization of the C. elegans orthologue of the MachadoJoseph disease protein ataxin-3. FASEB J 2007;21:1126-1136. RODRIGUEZ-LEBRON, GONZALEZ-ALEGRE P. Silencing neurodegenerative disease: bringing RNA interference to the clinic. Expert Rev Neurother 2006;6:223-233. ROSENBLUM BB, OAKS F, MENCHEN S, JOHNSON B. Improved singlestrand DNA sizing accuracy in capillary electrophoresis. Nucleic Acids Res 1997;25:3925-3929. RUBINSZTEIN DC, AMOS W, LEGGO J, GOODBURN S, RAMESAR RS, OLD J, BONTROP R, MCMAHON R, BARTON DE, FERGUSON-SMITH MA. Mutational bias provides a model for the evolution of Huntington's disease and predicts a general increase in disease prevalence. Nat Genet 1994;4:525530. RUBINSZTEIN DC. Protein-protein interaction networks in the spinocerebellar ataxias. Genome Biol. 2006;7:229 SAHBA S, NECHIPORUK A, FIGUEROA KP, NECHIPORUK T, PULST SM. Genomic structure of the human gene for spinocerebellar ataxia type 2 (SCA2) on chromosome 12q24.1. Genomics 1998;47:359-364. 90 SALEEM Q, CHOUDHRY S, MUKERJI M, BASHYAM L, PADMA MV, CHAKRAVARTHY A, MAHESHWARI MC, JAIN S, BRAHMACHARI SK. Molecular analysis of autosomal dominant hereditary ataxias in the Indian population : high frequency of SCA 2 and evidence for a common founder mutation. Hum Genet 2000;106:179-187. SAMBROOK J, FRITSCH EF, MANIATS T. Molecular cloning. 2a Ed. USA: Cold Spring Harbor Laboratory 1989;2:9.16-9.19. SANPEI K, TAKANO H, IGARASHI S, SATO T, OYAKE M, SASAKI H, WAKISAKA A, TASHIRO K, ISHIDA Y, IKEUCHI T, KOIDE R, SAITO M, SATO A, TANAKA T, HANYU S, TAKIYAMA Y, NISHIZAWA M, SHIMIZU N, NOMURA Y, SEGAWA M, IWABUCHI K, EGUCHI I, TANAKA H, TAKAHASHI H, TSUJI S. Identification of the spinocerebellar ataxia type 2 gene using a direct identification of repeat expansion and cloning technique. Nature Genet 1996;14:277-284. SCHELHAAS HJ, VERBEEK DS, VAN DE WARRENBURG BP, SINKE RJ. SCA19 and SCA22: evidence for one locus with a worldwide distribution. Brain 2004;127. SCHÖLS L, AMOIRIDIS G, BÜTTNER T, PRZUNTEK H, EPPLEN JT, RIESS O. Autosomal dominant cerebellar ataxia: Phenotype differences in genetically defined sybtypes? Ann Neurol 1997;42:924-932. SCHÖLS L, BAUER P, SCHMIDT T, SCHULTE T, RIESS O. Autosomal dominant cerebellar ataxias: clinical features, genetics, and pathogenesis. Lancet Neurol 2004;3:291–304. SERRA HG, DUVICK L, ZU T, CARLSON K, STEVENS S, JORGENSEN N, LYSHOLM A, BURRIGHT E, ZOGHBI HY, CLARK HB, ANDRESEN JM, ORR HT. RORalpha-mediated Purkinje cell development determines disease severity in adult SCA1 mice. Cell 2006;127:669-671. SERVADIO A, KOSHY B, ARMSTRONG D, ANTALFFY B, ORR HT, ZOGHBI HY. Expression analysis of the ataxin-1 protein in tissues from normal and spinocerebellar ataxia type 1 individuals. Nature Genet 1995;10:9498. SHIMIZU Y, YOSHIDA K, OKANO T, OHARA S, HASHIMOTO T, FUKUSHIMA Y, IKEDA S. Regional features of autosomal-dominant cerebellar ataxia in Nagano: clinical and molecular genetic analysis of 86 families. J Hum Genet 2004;49:610-616. SHULMAN JM, SHULMAN LM, WEINER WJ, FEANY MB. From fruit fly to bedside: translating lessons from Drosophila models of neurodegenerative disease. Curr Opin Neurol 2003;16:443-449. SHULTS CW. Coenzyme Q10 in neurodegenerative diseases. Curr Med Chem 2003;10:1917-1921. 91 SILVEIRA I, LOPES-CENDES I, KISH S, MACIEL P, GASPAR C, COUTINHO P, BOTEZ MI, TEIVE H, ARRUDA W, STEINER CE, PINTO-JÚNIOR W, MACIEL JA, JERIN S, SACK G, ANDERMANN E, SUDARSKY L, ROSENBERG R, MACLEOD P, CHITAYAT D, BABUL R, SEQUEIROS J, ROULEAU GA. Frequency of spinocerebellar ataxia type 1, dentatorubropallidoluysian atrophy, and Machado-Joseph disease mutations in a large group of spinocerebellar ataxia patients. Neurology 1996;46:214-218. SILVEIRA I, MIRANDA C, GUIMARÃES L, MOREIRA MC, ALONSO I, MENDONÇA P, FERRO A, PINTO-BASTO J, COELHO J, FERREIRINHA F, POIRIER J, PARREIRA E, VALE J, JANUÁRIO C, BARBOT C, TUNA A, BARROS J, KOIDE R, TSUJI S, HOLMES SE, MARGOLIS RL, JARDIM L, PANDOLFO M, COUTINHO P, SEQUEIROS J. Trinucleotide repeats in 202 families with ataxia: a small expanded (CAG)n allele at the SCA 17 locus. Arch Neurol 2002;59:623-629. SISODIA SS. Nuclear inclusions in glutamine repeat disorders: are they pernicious, coincidental, or beneficial? Cell 1998;95:1-4. SOONG BW, PAULSON HL. Spinocerebellar ataxias: an update.Curr Opin Neurol. 2007;20:438-446 SOUNIS E. Bioestatística. Princípios fundamentais, metodologia, estatísticas, aplicação à ciências biológicas. 2ª Ed. Revisada, McGraw-Hill, 1975. STEVANIN G, GIUNTI P, DAVID G, BELAL S, DURR A, RUBERG M, WOOD N, BRICE A. De novo expansion of intermediate alleles in spinocerebellar ataxia 7. Hum Molec Genet 1998;7:1809-1813. STEVANIN G, DURR A, BRICE A. Clinical and molecular advances in autosomal dominant cerebellar ataxias : from genotype to phenotype and physiopathology. Eur J Hum Genet 2000;8:4-18. STOREY E, DU SART D, SHAW JH, LORENTZOS P, KELLY L, MCKINLEY GARDNER RJ, FORREST SM, BIROS I, NICHOLSON GA. Frequency of spinocerebellar ataxia types 1, 2, 3, 6, and 7 in Australian patients with spinocerebellar ataxia. Am J Med Genet 2000;95:351-357. STTATERFIELD TF, PALLANCK LJ. Ataxin-2 and its Drosophila homolog, ATX2, physically assemble with polyribosomes Hum Mol Genet 2006;15:2523-2532. TAKANO H, CANCEL G, IKEUCHI T. Close associations between prevalences of dominantly inherited spinocerebellar ataxias with CAG-repeat expansions and frequencies of large normal CAG alleles in Japanese and Caucasian populations. Am J Hum Genet 1998;63:1060–1066 TAKIYAMA Y, OYANAGI S, KAWASHIMA S, SAKAMOTO H, SAITO K, YOSHIDA M, TSUJI S, MIZUNO Y, NISHIZAWA M. A clinical and 92 pathologic study of a large Japanese family with Machado-Joseph disease tightly linked to the DNA markers on chromosome 14q. Neurology 1994;44:1302-1308. TAKIYAMA Y, IGARASHI S, ROGAEVA EA, ENDO K, ROGAEV EI, TANAKA H, SHERRINGTON R, SANPEI K, LIANG Y, SAITO M, TSUDA T, TAKANO H, IKEDA M, LIN C, CHI H, KENNEDY JL, LANG AE, WHERRETT JR, SEGAWA M, NOMURA Y, YUASA T, WEISSENBACH J, YOSHIDA M, NISHIZAWA M, KIDD KK, TSUJI S, GEORGE-HYSLOP PH. Evidence for inter-generational instability in the CAG repeat in the MJD1 gene and for conserved haplotypes at flanking markers amongst Japanese and Caucasian subjects with Machado-Joseph disease. Hum. Molec. Genet, 1995;4:1137-1146. TAKIYAMA Y, SAKOE K, AMAIKE M, SOUTOME M, OGAWA T, NAKANO I, NISHIZAWA M. Single sperm analysis of the CAG repeats in the gene for dentatorubral - pallidoluysian atrophy (DRPLA): the instability of the CAG repeats in the DRPLA gene is prominent among the CAG repeat diseases. Hum Mol Gen, 1999;8:453-457. TANG B, LIU C, SHEN L, DAI H, PAN Q, JING L, OUYANG S, XIA J. Frequency of SCA 1, SCA 2, SCA 3/MJD, SCA 6, SCA 7, and DRPLA CAG trinucleotide repeat expansion in patients with hereditary spinocerebellar ataxia from Chinese kindreds. Arch Neurol 2000;57:540-544. TEIVE HA, IWAMOTO FM, CAMARGO CH, LOPES-CENDES I, WERNECK LC. Machado-Joseph disease versus hereditary spastic paraplegia: case report. Arq. Neuro Psiquiatria 2001;59:809-811. TEIVE HAG E ARRUDA WO. A família Drew de Walworth – um século após a avaliação inicial finalmente o diagnóstico. Arq Neuropsiquiatr 2004;62:177180. TEIVE HA. Avaliação clínica e genética de oito famílias brasileiras com ataxia espinocerebelar tipo 10, Tese de doutoramento 2005, Curitiba, BR. TRUANT R, RAYMOND LA, XIA J, PINCHEV D, BURTNIK A, ATWAL RS. Canadian Association of Neurosciences Review: polyglutamine expansion neurodegenerative diseases. Can J Neurol Sci 2006;33:278-291 TSUDA H, JAFAR-NEJAD H, PATEL AJ, SUN Y, CHEN HK, ROSE MF, VENKEN KJ, BOTAS J, ORR HT, BELLEN HJ, ZOGHBI HY. The AXH domain of Ataxin-1 mediates neurodegeneration through its interaction with Gfi-1/Senseless proteins. Cell 2005;26-122:633-644 VALERA AG, DÍAZ-HERNÁNDEZ M, HERNÁNDEZ F, LUCAS JJ. Testing the possible inhibition of proteasome by direct interaction with ubiquitylated and aggregated huntingtin. Brain Res Bull 2007;30-72:121-123. 93 VAN ALFEN N, SINKE RJ, ZWARTS MJ, GABREËLS-FESTEN A, PRAAMSTRA P, KREMER BP, HORSTINK MW. Intermediate CAG repeat lenghts (53, 54) for MJD/SCA 3 are associated with an abnormal phenotype. Ann Neurol 2001;49:805-807 VAN DE WARRENBURG BP, SINKE RJ, VERSCHUUREN-BEMELMANS CC. Spinocerebelllar ataxias in the Netherlands : prevalence and age at onset variance analysis. Neurology 2002;58:702-708. VAN SWIETEN JC, BRUSSE E, DE GRAAF BM, KRIEGER E, VAN DE GRAAF R, DE KONING I, MAAT-KIEVIT A, LEEGWATER P, DOOIJES D, OOSTRA BA, HEUTINK P. A mutation in the fibroblast growth factor 14 gene is associated with autosomal dominant cerebellar ataxia. Am J Hum Genet 2003;72:191-199. VELÁZQUEZ-PÉREZ L, SANTOS FN, GARCÍA R, PANEQUE HM, HECHAVARRÍA PR. Epidemiology of Cuban hereditary ataxia. Rev Neurol 2001;32:606-611. VERBEEK DS, SCHELHAAS JH, IPPEL EF, BEEMER FA, PEARSON PL, SINKE RJ. Identification of a novel SCA locus (SCA 19) in a Dutch autosomal dominant cerebellar ataxia family on chromosome region 1p21-q21. Hum Genet 2002;111:388-393. VERHOEF L, LINDSTEN K, MASUCCI MG, DANTUMA NP. Aggregate formation inhibits proteasomal degradation of polyglutamine proteins. Hum Molec Genet 2002;11:2689-2700. VIEIRA S. Bioestatística-Tópicos Avançados. Ed. Campus. RJ, 2003. VINCENT JB, NEVES-PEREIRA ML, PATERSON AD, YAMAMOTO E, PARIKH SV, MACCIARDI F, GURLING HM, POTKIN SG, PATO CN, MACEDO A, KOVACS M, DAVIES M, LIEBERMAN JA, MELTZER HY, PETRONIS A, KENNEDY JL. An unstable trinucleotide-repeat region on chromosome 13 implicated in spinocerebellar ataxia: a common expansion locus. Am J Hum Genet 2000;66:819-829. VUILLAUME I, DEVOS D, SCHRAEN-MASCHKE S, DINA C, LEMAINQUE A, VASSEUR F, BOCQUILLON G, DEVOS P, KOCINSKI C, MARZYS C, DESTÉE A, SABLONNIÈRE B. A new locus for spinocerebellar ataxia (SCA 21) maps to chromosome 7p21.3-p15.1. Ann Neurol 2002;52:666-670. WARRICK JM, MORABITO LM, BILEN J, GORDESKY-GOLD B, FAUST LZ, PAULSON HL, BONINI NM. Ataxin-3 suppresses polyglutamine neurodegeneration in Drosophila by a ubiquitin-associated mechanism. Mol Cell 2005;8(1):37-48. WATERS MF, MINASSIAN NA, STEVANIN G, FIGUEROA KP, BANNISTER JP, NOLTE D, MOCK AF, EVIDENTE VG, FEE DB, MULLER U, DURR A, BRICE A, PAPAZIAN DM, PULST SM. Mutations in voltage-gated 94 potassium channel KCNC3 cause degenerative and developmental central nervous system phenotypes. Nat Genet 2006;38:447-451 WATKINS WS, BAMSHAD M, JORDE LB. Population Genetics of trinucleotide repeat polymorphism. Hum Mol Genet 1995;4:1485-1491 WORTH PF, GIUNTI P, GARDNER-THORPE C, DIXON PH, DAVIS MB, WOOD NW. Autosomal dominant cerebellar ataxia type III: linkage in a large British family to a 7.6-cM region on chromosome 15q14-21.3. Am. J. Hum. Genet 1999;65:420-426. XIA H, MAO Q, ELIASON SL, HARPER SQ, MARTINS IH, ORR HT, PAULSON HL, YANG L, KOTIN RM, DAVIDSON BL. RNAi suppresses polyglutamine-induced neurodegeneration in a model of spinocerebellar ataxia. Nature Med 2004;10: 816-820. YAMASHITA I, SASAKI H, YABE I, FUKAZAWA T, NOGOSHI S, KOMEICHI K, TAKADA A, SHIRAISHI K, TAKIYAMA Y, NISHIZAWA M, KANEKO J, TANAKA H, TSUJI S, TASHIRO K. A novel locus for dominant cerebellar ataxia (SCA14) maps to a 10.2-cM interval flanked by D19S206 and D19S605 on chromosome 19q13.4-qter. Ann Neurol 2000;48:156-163. ZERYLNICK C, TORRONI A, SHERMAN SL, WARREN ST. Normal variation at the myotonic dystrophy loci in global human populations. Am J Hum Genet 1995;56:123-130. ZOGHBI HY E ORR HT. Glutamaine repeats and neurodegeneration. Ann Ver Neurosc 2000;23:217-247. ZORTEA M, ARMANI M, PASTORELLO E, NUNEZ GF, LOMBARDI S, TONELLO S, RIGONI MT, ZULIANI L, MOSTACCIUOLO ML, GELLERA C, DI DONATO S, TREVISAN CP. Prevalence of inherited ataxias in the province of Padua, Italy. Neuroepidemiology 2004;23:275-280. ZUCHENKO O, BAILEY J, BONNEN P, ASHIZAWA T, STOCKTON DW, AMOS C, DOBYNS WB, SUBRAMONY SH, ZOGHIBI HY. Autosomal Dominant cerebellar ataxia (SCA 6) associated with small polyglutamine expansions in the α 1 A-voltage-dependent calcium channel. Nature Genetics 1996;15:62-69. ZUHLKE C, DALSKI A, HELLENBROICH Y, BUBEL S, SCHWINGER E, BURK K. Spinocerebellar ataxia type 1 (SCA1): Phenotype-genotype correlation studies in intermediate alleles. 2002;Europ J Hum Genet 10:204209. 95 Endereços eletrônicos: BIRD TD. Hereditary ataxia Overview. 2007; Gene reviews. http://www.geneclinics.org/profiles/ataxias/details.html#diffdx (10/01/2008) http://www.aaadmj.com/doenca.php (10/01/2008) 96 VIII. APÊNDICES 97 Apêndice 1: Instrumento de coleta de dados para grupo controle GRUPO CONTROLE Nome: Sexo: feminino masculino Naturalidade: Data de Nascimento: / / / / Estado Civil: Grupo Étnico: Escolaridade: Profissão: Endereço: Bairro: Cidade CEP: UF: Telefone: Doença: Doença na família: Observações: Data: 98 Apêndice 2: Instrumento de coleta de dados para pacientes PACIENTES Nome: Sexo: feminino masculino Data de Nascimento: Naturalidade: Estado Civil: Grupo Étnico: Número do Registro: Escolaridade: Profissão: / / / / Endereço: Bairro: Cidade CEP: UF: Telefone: Suspeita Clínica: Casos na Família: Observações: Data: 99 Apêndice 3: Termo de consentimento informado livre e esclarecido - Pacientes MODELO DE TERMO DE CONSENTIMENTO INFORMADO LIVRE E ESCLARECIDO – GRUPO DE ESTUDO Título do Projeto: ESTUDO MOLECULAR DAS ATAXIAS CEREBELARES. Investigador: Lineu César Werneck Local da Pesquisa: Serviço de Neurologia, Hospital de Clínicas da Universidade Federal do Paraná. Endereço e telefone: Rua Gal. Carneiro, 161 – Sala 310 – Fone 360-1800, Ramal 6261. Você está sendo convidado (a) a participar de uma pesquisa, coordenada por um profissional de saúde agora denominado pesquisador. Para poder participar, é necessário que você leia este documento com atenção. Ele pode conter palavras que você não entende. Por favor peça aos responsáveis pelo estudo para explicar qualquer palavra ou procedimento que você não entenda claramente. O propósito deste documento é dar a você as informações sobre a pesquisa e, se assinado, dará a sua permissão para participar no estudo. O documento descreve o objetivo, procedimentos, benefícios e eventuais riscos ou desconfortos caso queira participar. Você só deve participar do estudo se você quiser. Você pode se recusar a participar ou se retirar deste estudo a qualquer momento. INTRODUÇÃO As ataxias cerebelares são doenças hereditárias, que afetam o cerebelo (órgão do sistema nervoso central que coordena os movimentos) ou suas conexões, determinando dificuldades na execução dos movimentos, marcha ou palavra. Não existe um tratamento definitivo para essas doenças e ainda faltam conhecimentos para compreender como os sintomas são produzidos a partir das alterações no DNA. As alterações no DNA são variáveis, existindo repetições de segmentos do DNA, que pode variar em cada família. Essas repetições poderão ter relação com a intensidade dos sintomas e a época do aparecimento da doença. Para que essas repetições de segmento do DNA sejam valorizadas, é necessário que exista uma correlação dos sintomas com tipo de alteração, comparando com uma população normal. PROPÓSITO DO ESTUDO Pretende verificar o número de repetições de um determinado segmento do DNA, a fim de estudar qual é a sua relação com os sintomas, comparando com as repetições de uma população normal. Ao mesmo tempo pretende organizar uma biblioteca de DNA (banco de DNA), que poderá ser utilizado no futuro, caso não seja encontrada no momento uma alteração no seu exame e possibilite novos testes, a medida que a ciência for se desenvolvendo. SELEÇÃO Para que você seja elegível para o estudo, deverá ser portador de uma ataxia cerebelar progressiva comprovada por história clínica e exame neurológico, ter investigação neurológica e por neuroimagem negativa para outras causas de ataxia, devendo ser excluídas as doenças tóxico infecciosas que determinem sintomas semelhantes. PROCEDIMENTOS 1. Inicialmente será realizada uma avaliação clínica com exame neurológico por um dos membros da equipe ou do Serviço de Neurologia, com registro de seus dados. 2. Investigação laboratorial para afastar outras doenças que podem simular ataxia cerebelar degenerativa, como exames de sangue rotineiros, neuroimagem, líquido cefalorraquidiano ou testes eletrofisiológico, dependendo dos dados colhidos na avaliação clínica. 100 3. Em seguida serão colhidos 20 ml (centímetros cúbicos) de sangue, que será propriamente identificado para controle do laboratório. 4. Esse sangue será enviado ao Laboratório de Biologia Molecular da Neurologia, de onde será extraído o seu DNA. 5. O DNA será armazenado em “freezer” com a devida identificação. 6. Posteriormente serão feitas a amplificações e classificação de segmentos do DNA que interessam ao estudo, através de métodos de laboratório específicos para as diversas ataxias cerebelares degenerativas. 7. O restante do DNA será armazenado para futuras análises e pesquisas, pois eventualmente é possível que nenhuma das ataxias testadas no momento seja positiva e você possua uma forma diferente ainda não descrita. Acompanhando a literatura, conforme forem sendo descritas, será feito o teste em seu material. Este procedimento não determinará nenhum dano, além do desconforto da picada da agulha para retirada do sangue, que será realizado com técnicas padrões e com material estéril. Participando desse estudo, você estará ajudando a conhecer a incidência da doença na população Brasileira, bem como eventualmente ajudar na elucidação do mecanismo de produção dos sintomas e talvez no desenvolvimento de um tratamento. Se desejar, poderemos fornecer os dados encontrados, mediante solicitação por escrito. PARTICIPAÇÃO VOLUNTÁRIA: Sua decisão em participar deste estudo é voluntária. Você pode decidir não participar no estudo. Uma vez que você decidiu participar do estudo, você pode retirar seu consentimento e participação a qualquer momento. Para tanto, deverá comunicar por escrito, solicitando o restante da amostra do DNA existente e que não foi utilizado. Se você decidir não continuar no estudo e retirar sua participação, você não será punido ou perderá qualquer benefício ao qual você tem direito. CUSTOS Não haverá nenhum custo a você relacionado aos procedimentos previstos no estudo. PAGAMENTO PELA PARTICIPAÇÃO Sua participação é voluntária, portanto você não será pago por sua participação neste estudo. PERMISSÃO PARA REVISÃO DE REGISTROS, CONFIDENCIALIDADE E ACESSO AOS REGISTROS: O Investigador responsável pelo estudo e equipe irão coletar informações sobre você. Em todos esses registros um código substituirá seu nome. Todos os dados coletados serão mantidos de forma confidencial. Os dados coletados serão usados para a avaliação do estudo, membros das Autoridades de Saúde ou do Comitê de Ética, podem revisar os dados fornecidos. Os dados também podem ser usados em publicações científicas sobre o assunto pesquisado. Porém, sua identidade não será revelada em qualquer circunstância. Você tem direito de acesso aos seus dados. Você pode discutir esta questão mais adiante com seu médico do estudo. CONTATO PARA PERGUNTAS Se você ou seus parentes tiver (em) alguma dúvida com relação ao estudo, direitos do paciente, ou no caso de danos relacionados ao estudo, você deve contatar o Investigador do 101 estudo ou sua equipe. Se você tiver dúvidas sobre seus direitos como um paciente de pesquisa, você pode contatar Comitê de Ética em Pesquisa em Seres Humanos (CEP) do Hospital de Clínicas da Universidade Federal do Paraná, pelo telefone: 360-1800. O CEP trata-se de um grupo de indivíduos com conhecimento científicos e não científicos que realizam a revisão ética inicial e continuada do estudo de pesquisa para o mantê-lo seguro e proteger seus direitos. DECLARAÇÃO DE CONSENTIMENTO DO PACIENTE: Eu li e discuti com o investigador responsável pelo presente estudo os detalhes descritos neste documento. Entendo que eu sou livre para aceitar ou recusar, e que eu posso interromper minha participação a qualquer momento sem dar uma razão. Eu concordo que os dados coletados para o estudo sejam usados para o propósito acima descrito. Eu entendi a informação apresentada neste termo de consentimento. Eu tive a oportunidade para fazer perguntas e todas as minhas perguntas foram respondidas. Eu receberei uma cópia assinada e datada deste Documento de Consentimento Informado. NOME DO PACIENTE ASSINATURA DATA NOME DO RESPONSÁVEL (Se menor ou incapacitado) ASSINATURA DATA NOME DO INVESTIGADOR (Pessoa que tomou o TCLE) ASSINATURA DATA 102 Apêndice 4: Termo de consentimento informado livre e esclarecido – Grupo Controle MODÊLO DE TERMO DE CONSENTIMENTO INFORMADO LIVRE E ESCLARECIDO – GRUPO CONTROLE Título do Projeto: ESTUDO MOLECULAR DAS ATAXIAS CEREBELARES. Investigador: Lineu César Werneck Local da Pesquisa: Serviço de Neurologia, Hospital de Clínicas da Universidade Federal do Paraná. Endereço e telefone: Rua Gal. Carneiro, 161 – Sala 310 – Fone 360-1800, Ramal 6261. Você está sendo convidado (a) a participar de uma pesquisa, coordenada por um profissional de saúde agora denominado pesquisador. Para poder participar, é necessário que você leia este documento com atenção. Ele pode conter palavras que você não entende. Por favor peça aos responsáveis pelo estudo para explicar qualquer palavra ou procedimento que você não entenda claramente. O propósito deste documento é dar a você as informações sobre a pesquisa e, se assinado, dará a sua permissão para participar no estudo. O documento descreve o objetivo, procedimentos, benefícios e eventuais riscos ou desconfortos caso queira participar. Você só deve participar do estudo se você quiser. Você pode se recusar a participar ou se retirar deste estudo a qualquer momento. INTRODUÇÃO As ataxias cerebelares são doenças hereditárias, que afetam o cerebelo (órgão do sistema nervoso central que coordena os movimentos) ou suas conexões, determinando dificuldades na execução dos movimentos, marcha ou palavra. Não existe um tratamento definitivo para essas doenças e ainda faltam conhecimentos para compreender como os sintomas são produzidos a partir das alterações no DNA. As alterações no DNA são variáveis, existindo repetições de segmentos do DNA, que pode variar em cada família. Essas repetições poderão ter relação com a intensidade dos sintomas e a época do aparecimento da doença. Para que essas repetições de segmento do DNA sejam valorizadas, será necessário saber qual o valor normal em uma população normal, isto é, que não seja portadora da doença. Isso vale para esta doença, bem como para outras doenças que poderão incidir na população em geral. PROPÓSITO DO ESTUDO Pretende verificar o número de repetições de um determinado segmento do DNA, a fim de estudar qual é o número normal que existe na população normal, para comparar com pacientes portadores de ataxias. Ao mesmo tempo pretende organizar uma biblioteca de DNA (banco de DNA), que poderá ser utilizado no futuro como controle para outras doenças hereditárias. SELEÇÃO Para que você seja elegível para o estudo, deverá ter idade acima de 18 anos, gozar de boa saúde, não ter nenhuma doença conhecida, não ser portador ou ter conhecimento de qualquer doença genética, hereditária ou mal formações na sua família, não ter sido exposto à radiação ou a agentes tóxicos. PROCEDIMENTOS 8. Inicialmente será preenchido um formulário com seus dados (Nome, idade, sexo, profissão, endereço). 9. Em seguida serão colhidos 20 ml (centímetros cúbicos) de sangue, que será propriamente identificado para controle do laboratório. 103 10. Esse sangue será enviado ao Laboratório de Biologia Molecular da Neurologia, de onde será extraído o seu DNA. 11. O DNA será armazenado em “freezer”. 12. Posteriormente serão feitas a amplificações e identificações de segmentos do DNA que interessa ao estudo, através de métodos de laboratório específicos. 13. O restante do DNA será armazenado para futuras análises e pesquisas, a fim de servir como grupo controle em outras doenças genéticas. Este procedimento não determinará nenhum dano, além do desconforto da picada da agulha para retirada do sangue, que será realizado com técnicas padrões e com material estéril. Participando desse estudo, você estará ajudando a conhecer o padrão normal de DNA na população normal, comparando com as ataxias cerebelares e outras doenças que porventura o seu sangue venha a ser utilizado como controle. Se desejar, poderemos fornecer os dados encontrados, mediante solicitação por escrito. PARTICIPAÇÃO VOLUNTÁRIA: Sua decisão em participar deste estudo é voluntária. Você pode decidir não participar no estudo. Uma vez que você decidiu participar do estudo, você pode retirar seu consentimento e participação a qualquer momento. Para tanto, deverá comunicar por escrito, solicitando o restante da amostra do DNA existente e que não foi utilizado. Se você decidir não continuar no estudo e retirar sua participação, você não será punido ou perderá qualquer benefício ao qual você tem direito. CUSTOS Não haverá nenhum custo a você relacionado aos procedimentos previstos no estudo. PAGAMENTO PELA PARTICIPAÇÃO Sua participação é voluntária, portanto você não será pago por sua participação neste estudo. PERMISSÃO PARA REVISÃO DE REGISTROS, CONFIDENCIALIDADE E ACESSO AOS REGISTROS: O Investigador responsável pelo estudo e equipe irão coletar informações sobre você. Em todos esses registros um código substituirá seu nome. Todos os dados coletados serão mantidos de forma confidencial. Os dados coletados serão usados para a avaliação do estudo, membros das Autoridades de Saúde ou do Comitê de Ética, podem revisar os dados fornecidos. Os dados também podem ser usados em publicações científicas sobre o assunto pesquisado. Porém, sua identidade não será revelada em qualquer circunstância. Você tem direito de acesso aos seus dados. Você pode discutir esta questão mais adiante com seu médico do estudo. CONTATO PARA PERGUNTAS Se você ou seus parentes tiver (em) alguma dúvida com relação ao estudo, direitos do paciente, ou no caso de danos relacionados ao estudo, você deve contatar o Investigador do estudo ou sua equipe. Se você tiver dúvidas sobre seus direitos como um paciente de pesquisa, você pode contatar Comitê de Ética em Pesquisa em Seres Humanos (CEP) do Hospital de Clínicas da Universidade Federal do Paraná, pelo telefone: 360-1800. O CEP trata-se de um grupo de indivíduos com conhecimento científicos e não científicos que 104 realizam a revisão ética inicial e continuada do estudo de pesquisa para o mantê-lo seguro e proteger seus direitos. DECLARAÇÃO DE CONSENTIMENTO DO PACIENTE: Eu li e discuti com o investigador responsável pelo presente estudo os detalhes descritos neste documento. Entendo que eu sou livre para aceitar ou recusar, e que eu posso interromper minha participação a qualquer momento sem dar uma razão. Eu concordo que os dados coletados para o estudo sejam usados para o propósito acima descrito. Eu entendi a informação apresentada neste termo de consentimento. Eu tive a oportunidade para fazer perguntas e todas as minhas perguntas foram respondidas. Eu receberei uma cópia assinada e datada deste Documento de Consentimento Informado. NOME DO PACIENTE ASSINATURA DATA NOME DO RESPONSÁVEL (Se menor ou incapacitado) ASSINATURA DATA NOME DO INVESTIGADOR (Pessoa que tomou o TCLE) ASSINATURA DATA 105 Apêndice 5: Comitê de ética e de pesquisa em seres humanos 106 107 Apêndice 6: Representação gráfica da amostra de pacientes 108 Apêndice 7: Lista de reagentes Amersham-Pharmacia Biotech: dNTPs- Amershan Pharmacia Biotech inc., Piscataway, NJ, USA. Applied Biosystems: Hi-Di Formamide, Performance Optimized Polymer 4 (POP 4), GS – 500 ROX, Arranjo de 4 capilares para ABI PRISM 3100 Avant, Iniciadores marcados com fluorescência FAM, VIC e NED- Foster City, CA, USA. Invitrogen: Proteinase K, Ácido Bórico, SDS, 10mg/mL, TRIS, EDTA, Accuprime DNA polimerase Systems -Invitrogen, Stratagene, La Jolla, CA, USA. Merck: Ácido Clorídrico, Acetato de Sódio, Carbonato de Sódio, Cloreto de Cálcio, Cloreto de Potássio, Cloreto de Sódio, Clorofórmio, Etanol absoluto, Fenol, Hidróxido de Sódio Millipore: Aparelho para obtenção de água ultra pura (água reagente Tipo I) Promega: YT1 DNA polimerase (respectivo Tampão 10X e MgCl2 25mM)Madison, WI, USA. Sigma: Acetato de amônia, Cloreto de Magnésio, DMSO, Glicerol 109 Apêndice 8: Preparo de soluções Solução de Lise de Eritrócitos (SLE): TRIS pH7,6 10 mM; MgCl2 5mM e NaCl 10mM Proteinase K 5mg/mL: Dissolver 10mg de proteinase K em 1 mL de Tampão TE e adicionar 1 mL de glicerol estéril. Armazenar a – 20oC. SDS 10 %: Dissover 10 g de SDS em água destilada e deionizada (q.s.p. 100ml) com auxílio do Banho Maria a 55oC. Armazenar 4 oC. Aquecer a solução no momento de uso Fenol Saturado: Liquefazer o fenol a 65 oC. Depois de liquefeito, adicionar a mesma quantidade de Tris 50mM pH 10,0 e deixar agitando durante a noite. Deixar em repouso por 30 minutos e retirar a fase aquosa (fase superior). Adicionar a mesma quantidade de Tris HCl 50 mM pH 8,0 e deixar agitando por 2 h. Decorrido o tempo esperar 30 minutos e medir o pH do fenol (fase inferior) que deverá ser em torno de 8,0. Caso não esteja em torno de 8,0 repetir as duas etapas acima. Acetato de Sódio 3M: Dissolver 24,1 g de acetato de sódio em água destilada e deionizada q.s.p.100mL Etanol 70%: Misturar 70 mL de etanol absoluto em 30mL de água destilada e deionizada Tampão TE: TRIS pH 7,6 10mM e EDTA pH 8,0 0,1 mM 110 Apêndice 9: Características clínicas da amostra de 56 pacientes analisados Principais Sintomas o N DNA Diag Clin Diag Mol 7 Ataxia DMJ Sexo F Idade Mut herd 47 Mãe Alt Marcha + Disartria Tremor Nistagmo + - + 19 Ataxia normal F 54 - 45 DMJ DMJ F 25 Pai Mov Sac - Espast - + - + - - - + + - + - - 46 AEC 6 normal M 35 Pai + + - + - - 47 AEC 10 normal F 36 Pai + + - + - - 49 AEC 3 normal F 10 - + + - + - - 51 Ataxia normal M 32 - + + + - - - 53 Ataxia DMJ F 37 Pai + + - + - - 54 Ataxia AEC 2 F 14 Pai + + - - + - 79 AEC 2 normal F 12 - + + - - + - 95 PNP mot normal M 38 - + - + - - - 117 DMJ DMJ M 40 Pai + + - + - - 119 DMJ normal F 27 Mãe + - + - - - 120 DMJ DMJ F 49 Mãe + + + + - - 121 AEC 7 AEC 7 M 16 Pai + + + - - - 140 DMJ DMJ F 42 Mãe - + - - + - 141 DMJ DMJ M 26 Pai + + - + - - 151 AEC 6 normal M 50 Mãe + + - + - - 170 Ataxia normal M 23 Pai + + - - + - 171 Friedreich normal M 9 - + - - - - - 193 Freidreich normal F 35 Mãe + + - + - - 217 Hipoβptnmia normal F 11 - + - + - - - 222 Ataxia DMJ M 39 Pai + + - + - - DMJ DMJ M 33 Pai + + - + - + 247 Sind cerebe 248 e motora normal F 46 - + + - + - - 269 Ataxia normal M 46 Mãe + + - + - - 310 DMJ DMJ F 30 Mãe + + + - + + 311 DMJ DMJ M 34 Mãe + + - + + - 325 DMJ DMJ M 19 Pai + + - + - - 368 DMJ DMJ M 32 Pai + + - + - + 379 Ataxia normal M 25 Pai + + - - + - 387 AEC 6 normal F 26 - + + + - - - 392 DMJ normal F 42 - + - - - - 396 Esporádica normal F 20 Pai + + + + - - 400 mioclonica normal M 25 - + - + - - - - Epilepsia 401 Ataxia normal M 39 - - - + - - - 416 DMJ normal F 32 Mãe + + - + - - 429 Ataxia normal F 6 - + + - + - - 430 Essencial normal M 14 - - - + - - - 433 Atx. Espast.. normal M 7 - + + - - - - Tremor 111 Continuação Tabela 11 No DNA Diag Mol Principais Sintomas Sexo Idade Mut herd Alt Marcha Disartria Tremor Nistagmo Mov Sac Espast 453 Ataxia normal M 48 Pai + + + + - + 472 AEC 2 AEC 2 M 33 Mãe + + + - - - 475 AEC 2 AEC 2 M 38 Mãe + + - - + - 479 AEC 2 AEC 2 F 38 Mãe - + - + + - 490 AEC 2 AEC 2 M 26 Mãe + + - - + - 507 Ataxia normal F 20 - + + - - + + 547 AEC 7 AEC 7 M 44 Mãe + + - + - + 565 AEC 10 normal M 40 Pai + + - + - - 574 Ataxia normal M 35 - + + - - - - 580 AEC 10 normal F 48 - + + + + - + 615 AEC 4 normal F 30 - - + - - + + 627 Atx. Reces. normal M 15 - + + - - - - 650 Ataxia normal F 19 - - - - + - - 665 DMJ DMJ M 42 Mãe + - - + - - 706 DMJ normal M 37 - + - - - - - 783 Ataxia AEC 7 M 24 Pai TOTAL + + - - + + 49 45 16 28 13 9 - não informado; + presença; M=masculino; F=feminino; 112 Livros Grátis ( http://www.livrosgratis.com.br ) Milhares de Livros para Download: Baixar livros de Administração Baixar livros de Agronomia Baixar livros de Arquitetura Baixar livros de Artes Baixar livros de Astronomia Baixar livros de Biologia Geral Baixar livros de Ciência da Computação Baixar livros de Ciência da Informação Baixar livros de Ciência Política Baixar livros de Ciências da Saúde Baixar livros de Comunicação Baixar livros do Conselho Nacional de Educação - CNE Baixar livros de Defesa civil Baixar livros de Direito Baixar livros de Direitos humanos Baixar livros de Economia Baixar livros de Economia Doméstica Baixar livros de Educação Baixar livros de Educação - Trânsito Baixar livros de Educação Física Baixar livros de Engenharia Aeroespacial Baixar livros de Farmácia Baixar livros de Filosofia Baixar livros de Física Baixar livros de Geociências Baixar livros de Geografia Baixar livros de História Baixar livros de Línguas Baixar livros de Literatura Baixar livros de Literatura de Cordel Baixar livros de Literatura Infantil Baixar livros de Matemática Baixar livros de Medicina Baixar livros de Medicina Veterinária Baixar livros de Meio Ambiente Baixar livros de Meteorologia Baixar Monografias e TCC Baixar livros Multidisciplinar Baixar livros de Música Baixar livros de Psicologia Baixar livros de Química Baixar livros de Saúde Coletiva Baixar livros de Serviço Social Baixar livros de Sociologia Baixar livros de Teologia Baixar livros de Trabalho Baixar livros de Turismo
Download