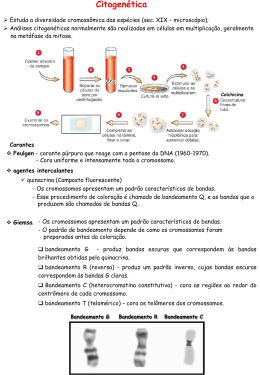
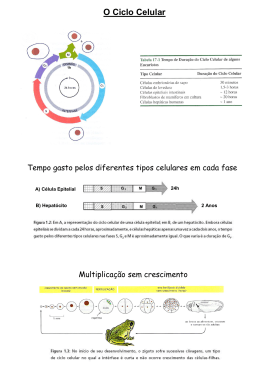
From the Chromosomal Loops and the Scaffold to the Classic Bands of Metaphase Chromosomes Y. Saitoh & U. K. Laemmli Cold Spring Harbor Symposia on Quantititive Biology Vol 83, 1993 Loops da Cromatina O estudo da estrutura dos cromossomos: Microscopia ótica: muito grosseira Microscopia eletrônica: muito detalhada Métodos bioquímicos: interferências destrutivas Em seu estado original, os cromossomos aparentam ser apenas estruturas cilíndricas, preenchidos homogeneamente com fibras nucleoprotéicas. Microscopia Ótica Microscopia Eletrônica Loops da Cromatina A noção de que ocorrem loops na cromatina a cada 100 Kb em média, é bastante sólida. Esta organização foi descoberta em cromossomos metafásicos sem proteínas e histonas, que foram removidas com poliânions de sulfato de dextran e heparina; com intuito de desenovela-los. Loops da Cromatina Os princípios da organização em loops foram comprovados com técnicas de: Seções cromossômicas finas Espalhamento dos cromossomos Imunoflorescência com anticorpos contra Topoisomerase II Loops da Cromatina Em cromossomos distendidos por exposição a baixas concentrações de sal ou pela retirada parcial de H1, os loops são observados como halos periféricos, devido a: Distensão das fibras da cromatina Desespiralização longitudinal das compõe o loop bases que Proteínas formam uma armação ou scaffold Alças de DNA são ligadas pela base SAR = scaffold attachment region DNA rico em AT com sítios para Topo II Loops da Cromatina A base dos loops de cromossomos nativos é uma região longitudinal e hetrocromática denominada de scaffold. Com base no modelo Loop / Scaffold poderiam ser identificadas proteínas específicas do scaffold e de regiões associadas a ele, como as SARs. Topoisomerase II e SARs A SC1 (Scaffold 1) é a principal proteína do scaffold e foi identificada como uma topoisomerase II. Essa proteína se liga seletivamente as SARs e está envolvida com o final da condensação cromossômica. Topoisomerase II e SARs As SRAs são os possíveis constituintes da base dos loops da cromatina e são regiões ricas em AT (cerca de 65 %). Proteínas que se ligam seletivamente as SRAs: Histona H1 HMG I Y - High Mobility Protein ARBD Bandas Cromossômicas São variações na estrutura longitudinal das cromátides que são reveladas por diferentes técnicas de coloração. Classicamente os cromossomos parecem ser construídos por discos empilhados, e cada disco difere de seu vizinho por: Densidade gênica Tempo de replicação Composição de bases Conteúdo de sequências repetitivas Conformação da cromatina Bandas Cromossômicas Os principais padrões cromossômico são: de bandamento Banda C - Heterocromatina centromérica, contendo DNA satélite e praticamente nenhum gene. Banda G - Composta por bandas de hetrocromatina facultativa, de replicação tardia e ricas em AT. Banda Q - Feita com quinacrina, cora também regiões ricas em AT, revelando um padrão semelhante as bandas C. Esquerda: Padrão de bandamento G de alta resolução em prometáfase do cromossomo 1 do Muntjac. As sub-bandas estão coradas em preto. Direita: Coloração com Daunonicina do mesmo cromossomo mostrando a relação 1:1 das regiões ricas em AT para as sub-bandas em preto Bandas Cromossômicas Apenas 20% dos genes humanos mapeados encontram-se em regiões de banda Q e G; e em sua maioria são housekeeping. A heterocromatina facultativa corada pela banda Q é responsável pelo silenciamento de genes tecido-específicos. Bandas Cromossômicas As bandas R possuem um padrão reverso ao encontrado nas bandas Q e G, coram regiões ricas em GC e se replicam na fase S. A maioria dos genes selvagens expressos estão localizados em bandas R. As bandas G / Q são cerca de 3.2 % mais ricas em AT do que as bandas R. Esta pequena diferença não poderia explicar os padrões de fluorescência com Daunomicina. Assim, a única opção seria uma alta organização cromossômica. Tudo é uma questão de como “ver”... Durante a corrida não se vêem as equipes Última volta Sprintador da equipe é conduzido Então, não seria uma questão de corar os cromossomos, mas sim de ver???? ? Modelo de Loops/Scaffold de Cromossomos Nativos Este modelo dita que as fibras das cromátides contêm uma região central, longitudinal denominada corda AT e dois loops diferentes: O Loop Q, que é menor e possui orientação longitudinal O Loop R, que é maior e possui orientação radial Na corda AT, um sinal ótico é emitido por corantes com alta afinidade por AT, pois nesta região estão as SARs. A coloração com Daunonicina cora de amarelo as regiões ricas em AT e a YOYO/MG cora de verde os Loops Q e R. Modelo de Loops/Scaffold de Cromossomos Nativos A construção das bandas R pode ser representada por este modelo como um scaffold central circundado por loops periféricos. O scaffold seria organizado assim: fortemente espiralizado na região de heterocromatina das bandas Q e longitudinalmente desespiralizado nas regiões de banda R. Bandas Q e R As bandas Q se originam da estriação cruzada das cordas AT e podem ser evidenciadas por corantes que se ligam a regiões ricas em AT. As bandas R ocupam uma posição mais central nas cordas AT. A complementaridade entre estas bandas é uma conseqüência direta da estrutura diferenciada das cordas AT. Bandas Q e R A estrutura dos cromossomos metafásicos considerada neste modelo combina com a Citogenética clássica e com os estudos bioquímicos. As bandas Q são muito raras ou ausentes nos invertebrados, nos vertebrados inferiores, como as serpentes e nas plantas. Isto pode ser explicado pela ausência de diferenciação no scaffold, culminando numa coloração homogênea. Detecção da Base dos Loops Foi detectada utilizando tanto anticorpos contra proteínas do scaffold, quanto fluorocromos altamente específicos para AT, como a Daunonicina e o Giemsa. A Dauno cora bandas Q e sabe-se que o DNA precisa ser composto por mais de 65 % de AT para que ela floresça. Assim se as SARs estivessem realmente organizadas no scaffold, um sinal forte seria observado com este tipo de coloração. Detecção do Corpo dos Loops O corante YOYO é um fluorocromo que cora todo o loop da cromatina. Para se corar o corpo de loop, o YOYO é associado a MG, que inibe a emissão das SARs. a) Esquema de protocolos de coloração seletiva para a base ou corpo dos loops da cromatina b) Simulação da coloração utilizando um líquido opaco no qual foi emergida uma mola. Complementaridade entre as Bandas Um cromossomo exposto a YOYO se cora homogeneamente, o que implicaria: Baixa concentração de DNA nas bandas R Cromatina mais aberta Comparando as colorações feitas com YOYO/MG e Dauno, observa-se um padrão complementar nas bandas R a) Comparação entre as colorações YOYO, YOYO/MG e Dauno b) Confocal mostrando bandas Q e R de telômeros. Nas bandas R positivas para YOYO/MG observam-se puffs nas regiões ricas em AT. a) O cromossomo 1 do Muntjac é corado em vermelho pela Dauno (cora cordas AT) e em azul pelo YOYO/MG (cora loops Q e R). b) Em maior aumento são mostrados os braços longos dos mesmos cromossomos. As regiões de overlap se coram de branco. Complementaridade nas sub-bandas A complementaridade perfeita entre as bandas R e Q é conseqüência direta do folding diferencial das cordas AT. Este mesmo padrão pode ser observado nas sub-bandas Tomando como exemplo um cromossomo visto sob microscopia confocal, a banda Q4 possui 4 sub-bandas que podem ser observadas em outras colorações. Topo II e SARs Topo II imunolocaliza o scaffold de cromossomos nativos, pois se liga especificamente a seqüências SARs através de ligações cooperativas. As SARs também possuem sítios para a proteína HMG IY, que se liga preferencialmente a repetições de A. Identificação do scaffold nativo por imunofluorescência. Os cromossomos foram imunocorados com Topo II e Topo II + Dauno. Topo II cora SARs e cordas AT, resultando num padrão de bandas Q e revela um sinal mais central nas bandas R. O Scaffold de Cromossomos Desespiralizados é Giemsa Positivo
Baixar