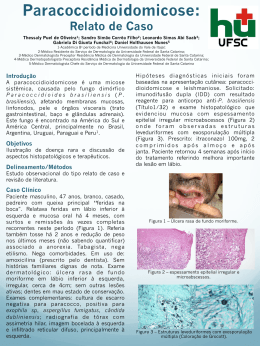
IV Reunião Regional da FeSBE ResumoID: 333-1 Utilização de técnicas subtrativas no estudo da expressão gênica diferencial. Bailao, A. M., Pereira, M., Soares, C. M. A. Universidade Federal de Goiás, UFG Estrada do Campus, UFG, ICB2, Sala 206, Laboratório de Biologia Molecular Palavras-chaves: Representational difference analysis, Gene expression, Paracoccidioides brasiliensis, Infectious process OBJETIVOS: O fungo Paracoccidioides brasiliensis é um patógeno humano com ampla distribuição na América Latina. Este microrganismo causa a paracoccidioidomicose, a micose sistêmica mais prevalente na América Latina. O fungo causa infecção quando o hospedeiro inala propágulos da fase miceliana do organismo. Estas partículas atingem o pulmão e podem disseminar para outros órgãos e tecidos. Embora o perfil de expressão gênica em P.brasiliensis tem sido estudado, pouco se conhece sobre o padrão de expressão de genes desta espécie durante o processo infeccioso. O presente trabalho objetivou a identificação de genes que podem contribuir para adaptação e sobrevivência do P.brasiliensis nos tecidos do hospedeiro durante o processo infeccioso. MÉTODO: Nos últimos anos, têm sido desenvolvidas varias técnicas que fornecem aos pesquisadores a habilidade do estudo de expressão gênica diferencial em um organismo ou célula, identificando gene(s) envolvidos em um determinado processo ou tratamento. A análise de diferença representacional é uma ferramenta poderosa e sensível na identificação de genes diferencialmente expressos. Esta técnica, que em sua forma original foi projetada para identificar diferenças entre populações de DNA genômico, subsequentemente sofreu modificações que permitem comparar diferenças entre populações de RNAm. O RDA é baseado em etapas sucessivas de hibridização subtrativa seguida de PCR que enriquece e isola os transcritos diferencialmente expressos, enquanto simultaneamente remove RNAm com expressão não diferencial. Está tecnologia foi utilizada na identificação de genes deP.brasiliensis induzidos durante infecção de camundongos, incubação com sangue humano, e incubação com plasma. A expressão diferencial dos genes identificados foi confirmada por ensaios de dot blot, northern blot e RT-PCRsq. RESULTADOS: Os dados revelaram que genes relacionados com captação de ferro, síntese de melanina e defesa celular foram superexpressos no modelo de infecção animal. Os transcritos induzidos durante a incubação de células leveduriformes com sangue humano foram aqueles predominantemente relacionados com síntese/remodelamento da parede celular. Genes relacionados com degradação de ácidos graxos, síntese protéica, estresse osmótico, remodelamento da parede celular e defesa celular foram superexpressos no tratamento com plasma humano. CONCLUSÃO: A análise dos dados mostra que o fungo P.brasiliensis apresenta diferentes respostas frente às diferentes condições analisadas, apresentando um favorecimento da captação de micronutrientes e manutenção da fluidez da membrana nos tecidos infectados.Durante a rota de disseminação hematogênica o remodelamento da parede celular e a resposta ao estresse osmótico seriam as estratégias adaptativas preferencialmente utilizadas. Os dados sugerem que o direcionamento do metabolismo para a βoxidação associado com a resposta ao estresse oxidativo é um mecanismo importante para a sobrevivência do fungo no contato com plasma, em sitio de infecção com extravasamento de plasma. As análises de RDA e a comparação entre o repertório de genes induzidos em cada uma das três condições experimentais sugere que P.brasiliensis apresenta uma resposta nicho-específica de adaptação e sobrevivência nos diferentes ambientes encontrados no hospedeiro que compõem as diferentes etapas do processo infeccioso. Apoio Financeiro: FINEP, CNPq
Baixar