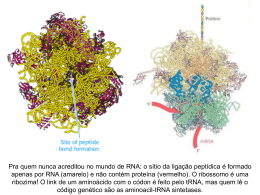
Universidade Federal de Pernambuco Centro de Ciências Biológicas Programa de Pós-Graduação em Genética Billy Manoel dos Santos Identificação molecular de bactérias lácticas presentes no caldo de cana-de-açúcar Recife 2012 Billy Manoel dos Santos Identificação molecular de bactérias lácticas presentes no caldo de cana-de-açúcar Dissertação apresentada ao Programa de Pós-Graduação Universidade em Federal Genética de da Pernambuco como parte dos requisitos exigidos para obtenção do título de Mestre em Genética. Orientador: Marcos Antônio de Morais Júnior Coorientador: Brígida Thays Luckwu de Lucena Recife 2012 Billy Manoel dos Santos Identificação molecular de bactérias lácticas presentes no caldo de cana-de-açúcar Aprovado em ___/___/____ Banca Examinadora: ____________________________________________ Dr. Marcos Antônio de Morais Júnior Universidade Federal de Pernambuco ____________________________________________ Dr. Valdir de Queiroz Balbino Universidade Federal de Pernambuco ____________________________________________ Dra. Brígida Thays Luckwu de Lucena Universidade Estadual da Paraíba ii Dedico este trabalho à minha estimada esposa que sempre esteve do meu lado me estimulando a seguir em frente. iii Agradecimentos Agradeço aos meus orientadores, Dr. Marcos Morais e Dra. Brígida Lucena, por confiarem e me introduzir no fascinante mundo da Ciência, por sempre me ajudar quando mais necessitei e por suas amizades que permanecerão para sempre. À minha mãe Irenice Maria dos Santos que me ofereceu a melhor educação que uma pessoa humilde pode dar. Meus familiares e amigos que também me fortaleceram a me aconselharam sempre. meus superiores hierárquicos da Briosa Polícia Militar de Pernambuco, que me apoiaram e, que quando possível me ajudavam para que pudesse concluir o trabalho que comecei. Sou grato ao meu casal de pastores, Prs. Dawison Alves e Adriana Alves, pelas orações e conselhos. E por fim, e com certeza o mais importante de todos, o meu Pai, que sempre me aconselhou a andar por caminhos planos, segura na minha mão sempre que necessito e que me deu a vida, e hoje eu vivo em função dEle. Pai é por isto e por muito mais eu Te agradeço, pois até aqui o Senhor me ajudou. iv Resumo A indústria alcooleira no Brasil perde anualmente vários milhares de reais devido a episódios de contaminação produzidos por bactérias. Além disso, outros milhares são gastos com o uso de antibióticos industriais para a contenção dessas contaminações. Infelizmente, não há ainda um catálogo completo de informações sobre todas as possíveis espécies bacterianas envolvidas na contaminação industrial, seus efeitos no processo e os fatores que levam a essas contaminações. Com a crescente demanda por fontes de energia renováveis, principalmente o etanol, grandes esforços têm sido feitos para aumentar a sua produção. Este trabalho tem como objetivo principal identificar as bactérias lácticas que entram no processo de fermentação alcoólica através do caldo de cana-de-açúcar com a utilização de ferramentas moleculares para uma rápida e eficiente identificação. Para foram realizadas coletas em quatro destilarias do Nordeste do Brasil durante a safra de 2007/2008. A identificação dos isolados foi realizada através da técnica ARDRA e pela análise do sequenciamento de DNA dos genes pheS e 16S. Os resultados obtidos mostram que os lactobacilos são os principais contaminantes do processo, com o Lactobacillus fermentum se destacando entre as demais. Também foi observado que entram no processo bactérias dos gene Weissella e Leuconostoc, com destaque para a W. confusa e o Leuc. mesenteroides que foram identificadas em três das destilarias estudadas. Além da identificação, os isolados de W. confusa e o Leuc. Mesenteroides foram tipados pelo REP-PCR com o primer (GTG)5. Os resultados mostraram que, além da diversidade de espécies bacterianas detectadas, existe uma diversidade de linhagens dessas duas principais espécies no processo. A espécie Leuc. mesenteroides tem sido apontada como contaminante em processo de fermentação alcoólica industrial, sendo algumas linhagens capazes de causar a formação de dextrana, podendo causar danos ao processo fermentativo. Desta forma se faz necessário trabalhos futuros com o fim de correlacionar os perfis destas espécies com possíveis danos à fermentação, para que com este conhecimento possa ser criadas novas estratégias de controle destes microorganismos. Palavras-chave: Lactobacillus, Leuconostoc, Identificação molecular, processo fermentativo, Weissella v Abstract The alcohol industry in Brazil loses several thousand dollars each year due to episodes of contamination produced by bacteria. In addition, thousands more are spent on the industrial use of antibiotics to contain these contaminants. Unfortunately, there is still no complete catalog of all possible information about bacterial species involved in industrial pollution, its effects on the process and the factors that lead to these contaminants. With the growing demand for renewable energy sources, especially ethanol, great efforts have been made to increase production. This work has as main objective to identify lactic acid bacteria that enter the process of fermentation through the juice sugarcane with the use of molecular tools for rapid and efficient identification. For this, samples were taken at four distilleries in northeastern Brazil during the harvest of 2007/2008. The identification of isolates was performed using the ARDRA technique and analysis of DNA sequencing of the pheS and 16S genes. The results show that lactobacilli are the main contaminants of the process with Lactobacillus fermentum stood out among the others. We also observed that bacteria enter the process of genus Weissella and Leuconostoc, with emphasis on the W. confused and Leuc. mesenteroides that were identified in three distilleries studied. Besides identification, the isolates of W. confusa and Leuc. mesenteroides were typed by rep-PCR with the primer (GTG)5. The results showed that, in addition to the variety of bacterial species found, there are several lines of these two major species in the process. The species Leuc. mesenteroides has been identified as a contaminant in industrial fermentation process, some strains can cause the formation of dextran, causing damage to the process. Thus further work is required in order to correlate the profiles of these species with possible damage to the fermentation, so that this knowledge can be created new control strategies such microorganisms. Key words: Lactobacillus, Leuconostoc, molecular identification, fermentative process, Weissella vi Lista de Figuras Item Página Figura 1. Fotomicrografia eletrônica de três importantes representantes 11 das bactérias lácticas Figura 2. Mapa físico da região 16S-23S ITS do rDNA típico do gênero 21 Lactobacillus Figura 3. Amplificação da região espaçadora 16S-23S do agrupamento 22 de genes de rDNA das amostras de bactérias lácticas da Fermentec Ltda Figura 4. Árvore filogenética feita a partir de sequências do gene 25 16S mostrando a posição do Lactobacillus hammesii sp. nov. strains LP38T e LP39 entre alguns lactobacilli Figura 5. Gêneros de LAB encontrados no caldo de cana das 37 destilarias Japungu, Miriri e Giasa durante a safra 2007/2008. Figura 6. Distribuição percentual das espécies de bactérias lácticas 38 identificadas utilizando as técnicas de ARDRA e sequênciamento do genes 16S e pheS. Figura 7. Bactérias lácticas identificadas a partir da análise do perfil de 39 restrição do rDNA amplificado ARDRA. Figura 8. Bactérias Lácticas identificadas a partir da análise de 40 sequências do gene 16S Figura 9. Árvore filogenética construída a partir do gene pheS utilizando 40 o método Neighbour-Joining dos isolados da destilaria Giasa. Figura 10. Dinâmica da população bacteriana da destilaria Giasa 41 vii durante a safra 2007/2008 Figura 11. Bactérias lácticas identificadas pela análise do perfil de 42 digestão do rDNA amplificado. Figura 12. Dinâmica da população bacteriana da destilaria Japungu 44 durante a safra 2007/2008 Figura 13. Árvore filogenética construída a partir das sequências de 46 nucleotídeos do gene 16S dos isolados encontrados no caldo de cana da destilaria Japungu durante a safra de 2007/2008. Figura 14. Bactérias lácticas identificadas pela análise do perfil de 47 digestão do rDNA amplificado. Figura 15. Diversidade da população bacteriana presente no caldo de 49 cana da destilaria Miriri durante a safra 2007/2008. Figura 16. Árvore filogenética construída a partir das sequências do 50 gene 16S dos isolados da destilaria Miriri Figura 17. Árvore filogenética construída a partir das sequências do 52 gene 16S dos isolados da destilaria Trapiche. Figura 18. Perfil de amplificação da região espaçadora 16S-23S dos 53 isolados identificados como pertencentes ao gênero Weissella. Figura 19. Perfis representativos de rep-PCR dos isolados das 55 destilarias Japungu, Miriri e Trapiche Identificados como W. confusa Figura 20. Perfil de amplificação da região espaçadora 16S-23S dos 56 isolados identificados como pertencentes ao gênero Leuconostoc. Figura 21. Perfis representativos de rep-PCR dos isolados das 56 destilarias Japungu e Trapiche Identificados como Leuc. mesenteroides viii Lista de Tabelas Tabela 1. Contaminantes bacterianos encontrados na Fermentação 8 Etanólica. Tabela 2. Primers utilizados nas reações de PCR e sequenciamentos. 36 Tabela 3. Isolados identificados a partir do sequenciamento dos genes 41 16S e pheS Tabela 4. Isolados identificados através da análise das sequências 43 parciais dos genes 16S e pheS durante a safra 2007/2008. Tabela 5. Isolados identificados a partir do sequenciamento dos genes 48 16S e pheS oriundos da destilaria Miriri durante a safra 2007/2008 Tabela 6. Isolados identificados a partir do sequenciamento dos genes 51 16S e pheS encontrados no caldo de partida da destilaria Trapiche. ix Lista de Abreviaturas Item Definição AAB Bactéria Acido Acético AFLP Polimorfismo de Comprimento de Fragmentos Amplificados ARDRA Análise de Restrição de DNA Ribossomal Amplificado BSA Albumina de Soro Bovino DDH Hibridização DNA-DNA DNA Ácido Desoxirribonucleico ISR Região Espaçadora Intergênica ITS Regiões Espaçadoras Intergênicas LAB Bactéria ácido láctica MLSA Análise de Sequências de Multiplos Loci) PCR Reação em Cadeia da Polimerase PFGE Eletroforese em Campo Pulsátil RAPD Amplificação Aleatória do DNA Polimórfico REA Análise de Enzimas de Restrição rep-PCR Amplificação de elementos de DNA repetitivos bacteriano rRNA Ácido Ribonucleico Ribossomal tRNA Ácido Ribonucleico transportador UFC Unidade Formadora de Colônia x Sumário Item+ Pagina Resumo v Abstract vi Lista de Ilustrações vii Lista de tabelas ix Lista de abreviaturas x 1. Introdução 1 2. Revisão da Literatura 3 2.1 Fermentação Alcoólica 3 2.2 Contaminantes bacterianos do processo de produção do etanol 4 2.3. Bactérias Lácticas (Lactic Acid Bacteria-LAB) 10 2.4. Identificação e Tipagem Genética 15 2.4.1. Perfil de plasmidial 16 2.4.2. Eletroforese em campo pulsátil (PFGE) 17 2.4.3. Amplificação aleatória do DNA polimórfico (RAPD) 17 2.4.4. Análise de enzimas de restrição (REA) 18 2.4.5. Ribotipagem 18 2.4.6. Análise de Restrição do DNA ribossomal amplificado (ARDRA) 19 2.4.7. Sequênciamento de DNA 23 2.4.8. Rep-PCR 27 3. Objetivos 30 3.1. Objetivo Geral 30 xi 3.2. Objetivos específicos 30 4. Material e Métodos 31 4.1. Isolamento, manutenção e identificação bacteriana 31 4.1.1 Isolamento e purificação das Culturas 31 4.2. Tipagem Genética 32 4.2.1. Extração de DNA 32 4.2.2. ARDRA-PCR 33 4.2.3. Rep-PCR 33 4.2.4. Sequênciamento dos genes 16S rRNA e pheS 34 5. Resultados 37 5.1. Análise Molecular das Bactérias isoladas do Caldo de Cana 37 5.1.1. Destilaria Giasa 39 5.1.2. Destilaria Japungu 42 5.1.3. Destilaria Miriri 47 5.1.4. Destilaria Trapiche 51 5.2. Análise Intraespecífica dos isolados 53 5.2.1. Weissella confusa 53 5.2.2. Leuconostoc mesenteroides 55 6. Discussão 57 7. Conclusões 61 8. Referências Bibliográficas 62 9. Anexos 73 10. Curriculum Lattes 84 xii 1. Introdução No Brasil o processo de produção do etanol se dá através da fermentação do caldo e/ou melaço de cana-de-açúcar pela levedura Saccharomyces cerevisiae, a mesma levedura que é utilizada na indústria da panificação. Curiosamente este processo ocorre em ambientes com baixa assepsia, que propiciam a contaminação por vários microrganismos, ocorrendo a entrada de bactérias e/ou a contaminação por outras espécies de leveduras, que são chamados de microrganismos selvagens. Estes podem promover episódios de contaminações que resultam numa queda na taxa de produção do etanol, consequentemente uma redução no rendimento industrial. As contaminações bacterianas, observadas no processo de fermentação do etanol, são apontadas como sendo as principais causas para a queda da viabilidade, do crescimento das leveduras e as que causam maiores prejuízos para a industria, proporcionando uma diminuição na produção do etanol. As bactérias lácticas (Lactic Acid Bacteria – LAB) se destacam como sendo os principais contaminantes bacterianos do processo, fermentando o carboidrato a ácido láctico e a ácido acético em pequenas quantidades, estes metabólitos diminuem o rendimento do etanol pela inibição da fermentação, principalmente pela ação do ácido láctico. Além disso, o estresse celular pelo qual as leveduras são submetidas faz com que excretem nutrientes para o meio, tais como, adenina, guanina, ácido aspártico e nicótico, triptofano, glicina e lisina, que podem estimular o crescimento bacteriano, agindo como um processo de feedback para os episódios de contaminação. Grandes perdas econômicas causadas pela queda do rendimento do etanol proveniente das contaminações por bactérias são observadas. Isto ocorre devido aos gastos com o controle dos episódios de contaminações quanto, quanto a redução da produção efetiva do etanol. A identificação das espécies de LAB que entram no processo de fermentação é de fundamental importância para a criação de uma base de dados para um maior conhecimento da complexa microbiota do processo de produção do etanol. E a partir destas informações buscar maneiras para um controle mais eficaz dos episódios de contaminações para que haja um aumento na produção de etanol. 2 2. REVISÃO DA LITERATURA 2.1. Fermentação Alcoólica O processo de fermentação industrial para a produção de álcool combustível ocorre através de dois processos: a) o processo de batelada alimentada (fed-batch) no qual toda fermentação se processa em dornas individuais com recuperação do fermento e sua reutilização em fermentações posteriores e; b) o processo continuo, que consiste na fermentação através de dornas interligadas nas quais a fermentação ocorre gradualmente em vários estágios, nesse processo também ocorre a reutilização do fermento, pois o mesmo retorna a dorna inicial depois de centrifugado juntamente com o mosto para realimentação do processo (Wheals et al., 1999; SilvaFilho, 2003). As espécie de leveduras comumente utilizadas para fermentar açucares são Saccharomyces cerevisiae, Saccharomyces uvarum, Schizasaccharomyces pombe e Kluyveromyces sp. (Kosaric et al. 1983). Sendo a S. cerevisiae que eram utilizada na industria da panificação. Estas se constituem um bom fermento por serem mais tolerantes a mudanças das condições do meio. Porém estas não são mais utilizadas pois durante o processo pois perdem sua viabilidade rapidamente, permanecendo por poucas gerações, causando instabilidade que oferece condições para organismos oportunistas se estabelecerem no processo, tais como outras leveduras e diversos outros tipos de bactérias (Basso et al., 2008). Para aumentar o desempenho e a longevidade das células de leveduras no processo fermentativo se passou a usar as células de leveduras provenientes do próprio processo de fermentação alcoólica. Esta medida fez reduzir o risco de 3 contaminações por organismos indesejáveis pois estas são mais resistentes e apresentam maior rendimento de etanol (Basso et al. 1993; Basso e Amorim, 1996). Apesar da utilização de células de leveduras obtidas diretamente da fermentação ainda ocorre contaminação do processo. Silva-Filho et al. observou que durante o processo fermentativo linhagens de leveduras introduzidas pelo substrato não estéril substituíram as leveduras introduzidas no começo da fermentação causando instabilidade durante o processo. Esta instabilidade pode ser apontada como uma das causas para o estabelecimento de episódios de contaminação tanto por outras leveduras, tais como a levedura Dekkera bruxelensis foi apontada como uma das maiores contaminantes das destilarias de álcool do Nordeste do Brasil (De Sousa-Liberal et al. 2007). Fato que também foi observado em destilarias de álcool na America do Norte e Europa onde os substratos utilizados são o amido de milho e a beterraba açucareira, respectivamente (Abbott et al. (2005); de Miniac, 1989; Ciani et al., 2003). Atualmente as leveduras utilizadas para a fermentação são obtidas a partir de seleções de S. cerevisiae utilizadas em processos fermentativos anteriores. Estas linhagens podem ser adquiridas comercialmente para este fim. Assim como outras leveduras se estabelecem devido a instabilidade outras bactérias também entram e podem trazer grandes problemas para a fermentação. 2.2 Contaminantes bacterianos do processo de produção do etanol O caldo de cana-de-açúcar se constitui um ótimo substrato para o crescimento de uma flora microbiana diversificada, devido ao sua composição em nutrientes orgânicos e inorgânicos, alta atividade aquosa, pH e temperaturas 4 favoráveis. Assim normalmente é possível se encontrar uma microbiota bacteriana presente no mosto utilizado como matéria-prima para a produção de açúcar e etanol (Yokoya, 1991; Gallo, 1992; de Souza-Liberal et al., 2007). No Brasil o processo fermentativo ocorre em um ambiente séptico e, por conta disso, está propenso a contaminações por bactérias e/ou outros tipos de leveduras. Vários são os problemas que podem surgir durante a fermentação, dentre eles os mais comuns são: Competição por açúcares. As bactérias estranhas ao processo competem e assimilam o açúcar que tinha por finalidade ser convertido em etanol; Toxinas e ácidos. Com o consumo do açúcar pelas bactérias são lançados ao meio toxinas e principalmente ácidos orgânicos que podem inibir o processo por causar a morte da levedura devidos suas propriedades e características importantes com relação aos efeitos inibitórios. Um dos ácidos mais importantes que causam problemas à fermentação é o ácido lático produzido em sua grade parte pelas bactérias lácticas; Floculação. Este é um fenômeno apresentado por leveduras, as quais se unem em agregados denominados flocos, constituídos por centenas e milhares de células. Ela propicia perdas de células de levedura no fundo das dornas ou nas centrifugas. Isto se acentua mais pela utilização do reciclo de células, o qual é utilizado para aumentar a produtividade do processo, reduzindo o tempo e os custos da fermentação (Yokoya & Oliva-Neto, 1991) Muito estudos foram feitos onde ficou claro que as bactérias têm um papel muito importante na fermentação, se destacando desempenhando um mal papel 5 para o processo. Vale salientar que nem todas as bactérias são prejudiciais à fermentação, muitas delas estão relacionadas com a produção de congêneres secundários que contribuem de forma benéfica para a qualidade de bebidas como a cachaça e o wisky uma vez que estes congêneres são responsáveis pela produção de sabor e aroma destas bebidas (Schwan et. al., 2001) Amorim & Oliveira (1982) encontraram como principais contaminantes da fermentação alcoólica as bactérias dos gêneros Acetobacter, Bacillus, Lactobacillus, Leuconostoc e Atreptococcus. Rondini (1985) encontrou no ambiente fermentativo um predomínio de bactérias Gram-positivas (65,1%), dentro das quais 62,1% pertencentes ao gênero Bacillus, 3% ao Lactobacillus e 1,5% pertencendo ao gênero Leuconostoc. Um estudo feito por Silva (1988) da microbiota bacteriana do caldo clarificado, pasteurizado e previamente resfriado encontrou bactérias dos gêneros Bacillus, Lactobacillus e Leuconostoc, com cerca de 3%, 38% e 12%, respectivamente. Gallo (1989) isolou a partir de coletas realizadas em diferentes pontos do processo fermentativo 334 culturas microbianas onde foi contatado a predominância de bactérias Gram-positivas (98,52%), onde 87,76% eram bastonetes, dentre eles 73,95% bastonetes não esporulados. Ainda neste estudo o autor comprovou que o gênero mais frequente foi o Lactobacillus (59,75%), com a identificação das seguintes espécies: L. acidophillus, L. agilis, L. amylophilus, L. animalis, L. buchneri, L. coryniformis subsp. torquens, L. delbruekii subsp. bulgaricus, L. delbruekii subsp.lactis, L. fermentum, L. helveticus, L. murinus, L. plantarum, L. reuteri, L. sake, L. viridescens, L. vitulinus; 26,58% de Bacillus, dos quais foram identificados: B. brevis, B. coagulans, B. lentus, B. megaterium, B.pasteurii, B. stearothermophilus; 6 8,76% de Staphylococcus; 1,56% de Micrococcus; 1,48% de representantes da família Enterobacteriaceae; 1,26% de Pediococcus e 0,70% de Streptococcus. Analises feitas a partir do leite de leveduras sem tratamento acido mostraram que, 62% das bactérias encontradas foram da espécie L. fermentum, 9% de L. murinus, 2% de L. plantarum, 9% de L. vaccinostercus e 2% de Leuconostoc (OlivaNeto, 1990). Em seus estudos de caracterização da microbiota contaminante da produção de álcool combustível, nos Estados Unidos, utilizando o milho como substrato Skiner e Leathers (2004) demonstraram que prevaleceram as bactérias lácticas, em particular Lactobacillus sp., representando cerca de 40% dos contaminantes totais isolados. Alem de outros isolados, menos frequentes, dos gêneros Clostridium, Eubacterium, Lactococcus, Leuconostoc, Pediococcus e Weissella (Tabela 1). A maior parte dos trabalhos realizados tornou evidente a predominância de bactérias, principalmente as Gram-positivas, no processo de fermentação do etanol com um importante destaque aos microorganismos dos gêneros Bacillus e Lactobacillus. As fontes de contaminações são peculiares de cada etapa do processo fermentativo e de cada destilaria. Bem como a seleção de determinadas espécies ou linhagens de microrganismos que favorecidos pelas condições pelas quais ocorre a fermentação em um determinada destilaria (Cherubin, 2003). Além de inúmeras fontes de contaminações que vão desde os microrganismos encontrados no solo onde é plantada a matéria prima até a linha de produção (dornas, fermento, trocadores de calor, etc.) da destilaria. Cherubim (2003) demonstrou que as contaminações bacterianas industriais são prejudiciais quando estão acima de 5,0 x 10 6 UFC/mL e; nestes casos, os 7 prejuízos econômicos são significativos quando atingem valores acima de 1,0 x 10 8 UFC/mL onde acontecem as quedas de rendimentos fermentativos. Tabela 1 Contaminantes bacterianos encontrados na Fermentação Etanólica. Organismo Referência Bacteriodes forsytus Skinner e Leathers (2004) Bifidobacterium sp., Bif. adolescentis, Bif. angulatum, Skinner e Leathers (2004) Clostridium sp., Cl. aerotolerans, Cl. Clostridiiforme Skinner e Leathers (2004) Eubacterium biforme Skinner e Leathers (2004) Fusobacterium nucleatum Skinner e Leathers (2004) Lactobacillus sp., L. acidophilus, L. amylovorus, L. brevis, L. Skinner e Leathers (2004); buchneri, L. casei, L. crispatus, L. delbrueckii subsp delbrueckii, L. Lucena et al. (2010); Chang delbrueckii subsp. lactis, L. diolivorans-like, L. ferintoshensis, L. et al. (1995) fermentum, L. gasseri, L. helveticus, L. hilgardii, L. lindneri, L. manihotivorans, L. mucosae, L. nagelii, L. paracasei subsp. paracasei, L. pentosus, L. plantarum, L. reuteri, L. rhamnosus, L. salivarus, L. vini, Lactobacillus sp. desconhecidas Lactococcus sp., Lac. lactis subsp. lactis, Lac. raffinolactis Skinner e Leathers (2004) Leuconostoc sp., Leuc. carnosum, Leuc. citreum, Leuc. lactis subsp. Skinner e Leathers (2004) lactis, Leuc. mesenteroides subsp. cremoris Oenococcus kitharae-like Lucena et al. (2010) Pediococcus sp., P. acidilactici, P. damnosus, P. parvulos, P. Skinner e Leathers (2004) pentosaceus, Pediococcus sp. desconhecidas Propionibacterium granulosum Skinner e Leathers (2004) Weissella sp. W. confusa, W. paramesenteroides, W. viridescens Skinner e Leathers (2004); Lucena et al. (2010) Modificado a partir de Beckner et al. (2011) 8 Makanjuola et a.l (1992) constatou uma contagem de bactérias de 4,5 x 108 UFC/mL em 30 horas de fermentação resultou em uma queda de 17% no rendimento do etanol. A queda do rendimento fermentativo geralmente deve-se à redução da viabilidade celular das leveduras em função do aumento da acidez no mosto após oito horas de cultivo associado com bactérias dos gêneros Bacillus e Lactobacillus da fermentação alcoólica (Alcarde et al., 2002). Lucena et. al. (2010) em seu estudo realizado em destilarias do Nordeste do Brasil, demonstrou que as LABs são facilmente encontradas nas destilarias. A maior frequência de isolados foram pertencentes ao gênero Lactobacillus tendo a maioria dos isolados identificados como pertencendo as espécies Lactobacillus fermentum e Lactobacillus vini. Estas espécies foram encontradas em meio contendo mais de 10% de etanol, sugerindo uma seleção de bactérias tolerantes a etanol ao longo do tempo em todo processo. Além destas, foram encontrados isolados pertencentes as espécies: L. plantarum, L. nagelii, L. amylovorus, L. brevis, L. diolivorans - like, L. ferintoshensis, L. manihotivorans, L. mucosae e Oenococcus kitaharae - like, todas pertencentes ao gênero Lactobacillus. Além destas também foram encontrados isolados da espécie Weissella parameseteroides. Por estar presentes em processos fermentativos com diferentes substratos e com os problemas que podem surgir no processo pela entrada deste organismos é de suma importância que se conheça quais as espécies de bactérias lácticas estão relacionadas com a contaminação da fermentação alcoólica. 9 2.3. Bactérias Lácticas (Lactic Acid Bacteria-LAB) As bactérias lácticas é como são chamadas as bactérias que são grampositivas, não formadoras de esporos, ácido tolerantes, fastidiosas, estritamente fermentativas, catalase-negativas, desprovidas de citocromos que crescem sob condições microaerofílicas a condições estritamente anaeróbicas. Elas também possuem um baixo conteúdo de guanina e citosina (G+C), ficando entre 33% a 55%. Esta alta heterogeneidade é refletida pelos testes fenotípicos dos isolados de Lactobacillus de diversos ecossistemas, que mostram perfis de fermentação do açúcar intermediários o que dificulta mais a tipagem ao nível de espécie. Dentre o grande número de gêneros que existe de bactérias lácticas os mais importantes são: Bifidobacterium, Carnobacterium, Enterococcus, Lactobacillus, Lactococcus, Leuconostoc, Pediococcus, Streptococcus, Tetraghenococcus e Weissella (Klein et al. 1998; Holzapfel et al. 2001; Moreira et al. 2005). As bactérias lácticas são encontradas em diversos tipos de habitats, desde solo, plantas, adubo, carnes, água, esgotos, restos de alimentos, em fermentações lácticas (na produção de laticínios como: queijos, iogurtes e manteiga) e em fermentações alcoólicas (produção de etanol e bebidas destiladas como: vinho, uísque, cachaça, cerveja). Além desses habitats as LAB também podem, ser encontradas na cavidade oral, trato digestivo e vaginal humano, e podem trazer benefícios para estes ecossistemas (Holzapfel et al. 2001; Morea et al. 1998; Simpson et al. 2001; Du Plessis et al., 2004). Elas também são quimiotróficas e fermentam o carboidrato produzindo como produto final ácido láctico e, em pequenas quantidades ácido acético. Algumas características fisiológicas são de interesse por suas funções como probióticos, uma pré-condição para sobreviver no 10 trato intestinal. Estas características são de menor interesse para a taxonomia das LAB, pois pode ocorrer mudanças morfofisiológicas nas bactérias devido as condições do meio onde elas estão sendo cultivadas, dificultando a identificação precisa dos isolados (Klein et al., 1988). Dentre as inúmeras espécies de bactérias utilizadas pelo homem podem se destacar as pertencentes ao gênero Lactobacillus (Figura 1). Elas estão envolvidas nas fermentações para a produção de diversos produtos tais como: vinho, cerveja, uísque de malte dentre outras bebidas fermentadas. Também estão presentes na fabricação de iogurtes, kefir, queijos. Ainda, outras espécies do gênero Leuconostoc são utilizadas na fabricação de queijos e manteiga. Na produção de vinho e cidra se destacam as bactérias do gênero Oenococcus. Alem de outros gêneros como o Weissella e Pediococcus que são responsáveis pela degradação de carnes e vegetais, fermentação, preparo e conservação de alimentos (Slonczewski e Foster, 2008). Figura 1. Fotomicrografia eletrônica de três importantes representantes das bactérias lácticas. Weissella confusa (A) encontrada em fermentações alcoólica (Lucena et al., 2010); Lactobacillus casei (B) utilizado no preparo (http://www.probioticsnews.creativetesting.co.uk/library); de Oenococcus leites oeni fermentados (C) bactéria frequentemente utilizada na fermentação de vinhos (barra = 3µm) (http://genome.jgipsf.org/oenoe/oenoe.home.html). 11 A identificação das bactérias tem sido, e ainda é feito principalmente através da utilização de marcadores morfofisiológicos e bioquímicos. Entretanto estes marcadores contam com a expressão de fenótipos variados, os quais podem não ser expressos sob certas condições (Nour, 1998). Tradicionalmente, as LAB têm sido classificadas com base nas propriedades fenotípicas, isto é, morfologia, no padrão de fermentação de carboidratos, diferenças entre a resistência a diferentes concentrações de NaCl, crescimento em diferentes tipos de meios e temperaturas e resistência a antibióticos. Por muito tempo estes foram os únicos critérios utilizados para a taxonomia de LAB (Holzapfel et at. 2001; Klein et al. 1998). Já os métodos taxonômicos modernos aplicados a LAB incluem a caracterização fenotípica e genotípica, reveladas em nível molecular. A identificação molecular de LAB é baseada nas análises na homologia DNA-DNA como método de referência. Para tal, sondas de ácidos nucleicos têm sido desenvolvidas para varias espécies de Lactobacillus, frequentemente em combinação com métodos primários (Klein et al. 1998; Tilsala-Timisjärvi e Alatossava 1997). A utilização de técnicas que são baseadas em características fenotípicas e genômicas, conhecida como taxonomia polifásica, têm se mostrado capaz de diferenciar espécies que são fenotipicamente semelhantes, mas diferente ao nível de genotípico. A taxonomia polifásica tem se mostrado confiável por se associar técnicas que revelam diferenças tanto a nível intra como interespecíficos do microrganismos. Isto é de suma importância na industria, uma vez que muitas espécies de bactérias são utilizadas como probiótico, sendo de extrema importância uma classificação mais acuradas desse grupo (Holzapfel et at. 2001; Klein et al. 1998). Por muito tempo as bactérias lácticas foram classificadas por suas características fenotípicas, tais como: morfologia, fermentação de diferentes 12 carboidratos, variação na temperatura de crescimento, fermentação de glicose, entre outras. Mas com os avanços nas técnicas moleculares novas técnicas têm surgido para aumentar e agilizar a identificação de bactérias muito próximas. A definição de espécie biológica não é aceita em espécies procarióticas, sendo necessário uma novo conceito de espécies para as mesmas. Em seu estudo Vandamme et al. (1996) definiu uma espécie procariótica como "uma categoria que engloba (preferencialmente) um grupo coerente genômicamente de isolados/linhagens individuais que compartilham um alto grau de similaridade em características independentes, testadas comparativamente sob condições altamente padronizadas" (Gevers et al., 2005). Na pratica, uma espécie procariótica é considerada sendo um grupo de linhagens, incluindo a linhagem tipo, que são caracterizada por um alto grau de consistência fenotípica, mostrando 70% de hibridização DNA-DNA (DDH, DNA-DNA Hybrization) e mais de 97% de identidade na sequência do gene 16S do RNA ribossomal (rRNA) (Vandamme et al., 1996; Gevers et al., 2005). Desde a década de 1970, espécies procarióticas foram descritas com base em dados produzidos em experimentos de hibridização DNA-DNA. Nesta abordagem, a similaridade genética entre isolados é avaliada pelo grau em que seus genomas hibridizam sob condições padronizadas. Isolados que demonstram mais de 70% de DDH e valores menores que 5% de diferença em sua temperatura de desnaturação (ΔTm) são considerados como pertencentes à mesma espécie (embora este valor limite pode ser ajustado pelos especialistas que estão familiarizados com a excentricidade de cada um dos grupos taxonômicos). Ao passo que, os isolados que compartilham menos que 50% de DDH pertencem, definitivamente, a espécies diferentes. Para facilitar a identificação são necessárias 13 consistência fenotípica dentro da espécie e as diferenças entre as espécies. A análise comparativa da sequência do gene 16S rRNA é amplamente utilizado para determinar a posição filogenética do isolados. Linhagens que mostram similaridade menores que 97% na sequência do gene 16S rRNA com todos os taxa conhecidos são consideradas como pertencendo a uma nova espécie, já que não existem exemplos em que cepas com este ponto de divergência na sequência do gene 16S do rRNA cumprir os critérios de apresentar mais de 70% de DDH (Gevers et al., 2005). Na prática, a caracterização taxonômica de uma coleção de isolados começam com uma triagem que permite os isolados esteritamente relacionados sejam agrupados e distinguidos de isolados não relacionados. Os métodos de triagem frequentemente utilizados incluem o conteúdo de ácidos graxos celular (um método quimiotaxonômico no qual os lipídeos que estão presentes nas células bacterianas são analisados e usados para delinear agrupamentos) (Welch, 1991; Gevers et al., 2005) e uma ampla gama de métodos de tipagem baseads na análise de DNA, incluindo AFLP (Amplified Fragment Length Polymorphism) entre outro métodos que se utilizam de primers randômicos (Vos, et al., 1996; van Belkum, 1994; Gevers 2005). Estes métodos têm a vantagem de serem relativamente rápidos e fáceis de executar e podem ser automatizados, tornando-os úteis para a triagem padronizada grande número de isolados. Posteriormente, a análise da sequência genética do gene ribossomal 16S rRNA é realizada com representantes dos diferentes grupos, e estas sequências são comparadas com as de espécies conhecidas. Desta forma, a posição filogenética é determinada. Com base nos resultados desta análise, os organismos são selecionados, e devem ser incluídos em experimentos de hibridização DNA-DNA (Gevers et al., 2005). 14 2.4. Identificação e Tipagem Genética Pesquisas continuas têm sido realizadas para definir um conceito de espécie procariótica, devido a particularidades da reprodução destes microrganismos. Stackebrandt (2002) sugeriu uma espécie procariota como "um grupo (de preferência) genomicamente coerente de isolados individuais/cepas compartilhando um alto grau de semelhança em (muitas) características independentes, comparativamente testado sob condições altamente padronizadas". Com esta continua busca por um conceito natural de espécie, baseados em novas abordagens genômicas e no crescente número de genomas sequenciados disponíveis vem mudando continuamente a taxonomia bacteriana e seus conceitos. Considerando que o conceito de espécies é o arcabouço teórico que descreve o que são espécies de bactérias e explica como eles são formados. A definição das espécies é a forma como a concepção é exercida na prática, e consistem de um conjunto de regras de fácil aplicação e estável (Doolittle & Papke, 2006; Konstantinidis et al., 2006). A definição de espécies bacterianas considera espécies de bactérias os grupos de linhagens que são caracterizados por um certo grau de consistência fenotípica, por um significativo grau de hibridação (>70%) DNADNA (DDH) e mais de 97% de identidade sequência do gene 16S da RNA do ribosomal (rRNA) (Ursing et al., 1995; Vandamme et al., 1996; Gevers et. al., 2005). Gevers et. al. (2005) em seu trabalho de reavaliação de espécies procarióticas demonstrou em seu estudo que a utilização do DDH e a análise de sequencias do gene 16S associadas com a utilização da Multilocus Sequence Analysis (MLSA) pode melhorar a caracterização genotípica dos procariotos ao nível intraespecífico com o uso de busca de similaridade entre genes housekeeping. 15 Inúmeras técnicas têm sido utilizadas para a identificação das LAB para uma rápida e precisa identificação e diferenciação inter e intraespecíficas. Para obter uma identificação precisa, técnicas fenotípicas e genotípicas são frequentemente combinadas em uma abordagem polifásica, onde as deficiências de um método é compensado pelo uso de outro método (De Bruyne, 2009). Alguns destes métodos se destacam por já serem consolidados devido à rapidez, praticidade, robustez, reprodutibilidade e eficácia na identificação de microrganismos. Dentre estes podemos citar alguns mais utilizados para o fim de identificar espécies ou linhagens de bactérias: perfil plasmidial, eletroforese em gel de campo pulsátil (PFGE). 2.4.1. Perfil plasmidial Consiste numa técnica utilizada para caracterizar bactérias a partir da análise do padrão criado quando plasmídios são separados, com base no peso molecular, por eletroforese em gel de agarose. Entretanto a falta de estabilidade dos plasmídios por um longo tempo para algumas linhagens em particular, pode ser a sua maior desvantagem (Towner & Cockayne, 1993). Em bactérias lácticas, o perfil plasmidial era considerado útil para a tipagem de linhagens dentro de uma espécie. Contudo, como resultado da instabilidade do DNA extracromossomal, métodos que utilizam o DNA cromossomal, têm sido mais utilizados por demonstrarem melhores resultados (Duffner e O’Connell, 1995, Holzapfel et al., 2001). 16 2.4.2. Eletroforese em campo pulsátil (PFGE) Consiste em um método com alto poder discriminatório e reprodutível, e tem sido usado para diferenciar linhagens de importantes bactérias probióticas, como Bifidobacteria, L. casei, L. acidophilus (Roy et al. 1996; Ferrero et al. 1996; Roussel et al. 1993; Holzapfel et al. 2001). Em muitos estudos com Lactobacillus, o PFGE tem se mostrado como um método poderoso para tipagem de linhagens, sendo também utilizado em estudos epidemiológicos. Contudo consiste em uma técnica laboriosa e cara, e apenas um limitado número de amostras pode ser analisado ao mesmo tempo (Ventura e Zink, 2002). 2.4.3. Polimorfismo de amplificação aleatória do DNA (RAPD) Essa técnica tem como principal vantagem a geração de perfis moleculares sem a necessidade de conhecimento específico do genoma a ser estudado. Contudo, seu aspecto negativo consiste na sua baixa reprodutibilidade, necessitando de condições cuidadosamente controladas (Seseña, 2004). Por ser uma técnica rápida e simples de ser executada ela elimina muitos problemas encontrados por outras técnicas moleculares como o PFGE e perfil plasmidial. Van Reenen (1996) utilizou esta metodologia para diferenciar Lactobacillus plantarum e Lactobacillus pentosus que apresentavam padrões similares para assimilação de açucares, contudo apresentaram padrões distintos para o RAPD. Outra desvantagem é o alto nível de padronização e controles dos parâmetros, como concentrações adequadas de DNA, magnésio, primer e enzima, assim como o número de ciclos da PCR (Bassam et al., 1992). 17 2.4.4. Análise de Enzimas de Restrição (REA) A utilização de endonucleases de restrição para a digestão do DNA cromossomal gera um padrão de fragmentos de restrição são separados em gel de agarose em uma eletroforese convencional. O padrão de bandas gerado por esta técnica é de difícil análise visual, sendo necessário a utilização de programas computacionais de análise multivariável (Charteris et al. 1997). O método de REA tem sido utilizada e conseguido bons resultados para a diferenciação de linhagens de L. acidophilus (Roussel et al. 1993) L. casei e L. rhamnosus (Ahrné e Molin, 1997), e L. reuteri (Ahrné e Molin, 1997; Stahl e Molin, 1994). 2.4.5. Ribotipagem Consiste na hibridização do tipo Southern Blotting de fragmentos de restrição do rRNA por . Em geral, o padrão obtido é mais estável e de melhor interpretação que o padrão conseguido pela REA (Charteris et al. 1997). A alta reprodutibilidade é outra vantagem desta técnica e a possibilidade de se utilizar uma sonda universal para todas as espécies, levando-se em consideração a similaridade entre os genes ribossomais (Pot et al. 1993; Grimont et al. 1986; Holzapfel et al. 2001). A ribotipagem vem sendo frequentemente utilizada na identificação e caracterização de linhagens e diferentes espécies de LAB encontradas em diferentes processos industriais como: fabricação do queijo mussarela (Morea et al. 1998), na produção de whisky (Simpson et al. 2001), trato vaginal (Pavlova et al. 2002), processo de fermentação do vinho (Du Plessis et al., 2004), produção de álcool combustível (Skinner e Leathers, 2004) entre outros. 18 2.4.6. Análise de Restrição do DNA Ribossomal Amplificado (ARDRA) A técnica de ARDRA (Amplified Ribosomal DNA Restriction Analysis) consiste na análise do perfil de digestão do produto da PCR da região ITS do loco de rDNA. O perfil gerado por ARDRA é distinto entre os grupos, podendo se diferenciar dentro de um mesmo grupo. Esta técnica tem se mostrado uma ferramenta importante na identificação de bactérias, e de linhagens dentro das espécies, pois nem sempre os métodos clássicos baseados nas características bioquímicas e/ou morfofisiológicas geram resultados claros e confiáveis (Dubernet et al. 2002; Collado e Hernández, 2007). Assim como a ribotipagem, a ARDRA faz uso da análise dos genes ribossomais que carregam consigo informações úteis sobre a relação evolutiva e de parentesco entre as bactérias (Miteva et al. 2001). Esta metodologia combina facilidade, rapidez, especificidade e sensibilidade, sendo ela precisa para a identificação de bactérias e se mostrando bastante eficiente na diferenciação das espécies dos gêneros Lactobacillus, Streptococcus e Bifidobacterium (Miteva et al. 2001; Moreira et al. 2005; Collado e Hernández, 2007). Três tipos de rRNAs são encontrados nos ribossomos das bactérias (23S, 16S e 5S). Os genes codificantes destes rRNAs são separados por sequências espaçadoras (ISRs) e são organizados em operons que podem variar de 1 a 11 por célula bacteriana. O mais comum arranjo destes genes para as diferentes subunidades dentro do operon segue a seguinte ordem 16S-23S-5S, são raras as exceções nas quais os genes não se encontram dispostos desta forma (Gürtler e Stanisich, 1996; Pisabarro et al. 1998; Roth et al. 1998; García-Martinez et al. 1999). Estes genes são essenciais para a sobrevivência dos organismos e são altamente 19 conservados entre as espécies bacterianas. O comprimento das regiões espaçadoras entre os genes do rRNA 16S-23S (ISRs), que são amplificados pela ribotipagem por PCR, podem variar entre as diferentes espécies ou dentro da mesma espécie (García-Martinez et al. 1999). As variações no comprimento das ISRs 16S-23S podem ser explicadas pelo fato de existir números e tipos de genes diferentes do tRNA que pode-se encontrar dentro destas regiões (Gürtler e Stanisich, 1996); além da presença de sequencia de reconhecimento de enzimas, como a ribonuclease III, esta enzima está envolvida no processo de splicing para a produção de ribossomos maduros (Bram et al. 1998); e boxA, sequência que tem um papel anti-terminador durante a transcrição (Harvey et al. 1988). O restante desta região consiste em sequências de genes não essenciais, e estas sequências estão sujeitas a frequentes eventos de inserção-deleção (García-Martinez et al., 1999). É sabido que a sequência espaçadora 16S-23S do DNA ribossomal exibe uma grande quantidade de elementos, com comprimentos variados e sequências conservadas com papeis funcionais definidos, como por exemplo, genes de tRNAs (Ruiz et al. 2000). A técnica de ARDRA-PCR foi utilizada com sucesso na diferenciação das espécies de bactérias produtoras de ácido acético destituídas de variações intraespecíficas indicando que a alta variabilidade da sequência 16S-23S ITS nestas bactérias pode ser utilizadas para fazer a identificação e classificação taxonômica de linhagens ou de grupos relatados dentro das próprias linhagens de bactérias (Ruiz et al. 2000; Shang et al. 2003). Para as espécies de Lactobacillus a utilização de sondas espécie-especificas para a diferenciação intraespecífica de todas as linhagens para L. delbrueckii conseguiu diferenciar com sucesso três linhagens de L. delbrueckii: a L. delbrueckii 20 subsp. bulgaris de L. delbrueckii subsp. delbrueckii/L. delbrueckii subsp. lactis pela análise do perfil de restrição do 16S gerado com a enzima EcoRI (Miteva et al. 2001). A metodologia da ARDRA constitui um especifico e reprodutível caminho para a identificação de LAB ao nível de espécie. Além disso, a maior parte dos gêneros co-isolados com Lactobacillus pôde ser identificada com precisão através da ARDRA da 16S-23S rRNA (Moreira et al. 2005). Esta região permite discriminar o gênero Lactobacillus devido ao seu padrão de amplificação, apresentando três bandas, correspondendo às três regiões espaçadoras (pequena, media e grande regiões espaçadoras), similar as encontradas nas espécies dos gêneros Pediococcus, Carnobacterium, Leuconostoc e Weissella. Estas diferenças nos tamanhos são devido a inserção do gene do tRNA-Ala dentro do médio espaçador ou dos genes dos tRNA-Ala e tRNA-Ile dentro do espaçador maior (Figura 1). Bifidobacteria, Enterococci, Streptococci e Staphylococci apresentam padrões de amplificação com uma banda ou duas com tamanhos diferentes, também devido a inserção dos genes dos tRNA (Moreira et al. 2005). 16-1A 16S 23-1B 23S ITS (p) tRNA tRNA AL A AL A ITS 5S ITS (m) tRNA IL E ITS (g) Inserção de tRNAs Figura 2. Mapa físico da região 16S-23S ITS do rDNA típico do gênero Lactobacillus mostrando a inserção dos tRNAs da alanina e isoleucina. Modificado de Lucena (2010) 21 O gênero Lactobacillus apresenta um perfil de amplificação da região 16S23S ITS com três bandas com a banda menor com cerca de 503 pb (Figura 2). Um estudo realizado por Lucena (2010) de linhagens industriais mostrou (Fermentec Ltda) o perfil de amplificação típico para Lactobacillus. 1Kb FT338B FT400B FT025B FT008B FT337B FT124B Figura 3. Amplificação da região espaçadora 16S-23S do agrupamento de genes de rDNA das amostras de bactérias lácticas da Fermentec Ltda. As linhagens estão representadas na parte superior do gel. Modificado a partir de Lucena (2010) Segundo Moreira et al. (2005) o padrão de amplificação e o perfil de digestão dos três espaçadores tem permitido a identificação de diferentes espécies de Lactobacillus, o que indica ser essa uma metodologia fácil e rápida para identificação de bactérias lácticas. Com isso, a técnica de ARDRA poderá ser utilizada na análise da população bacteriana associada a produção de etanol para a identificação dos contaminantes bacterianos presentes no processo de fermentação alcoólica. Por se tratar de uma técnica rápida, com alta sensibilidade e precisa para a identificação de LAB. 22 2.4.7. Sequênciamento de DNA A utilização de macromoléculas como as sequências de DNA tem se mostrado uma ótima ferramenta para a identificação e diferenciação de espécies intimamente relacionadas. A décadas a o DNA tem sido descrita como sendo um documento que registra as mudanças evolutivas das espécies e sendo utilizado para da diversidade filogenética e a relação evolutiva entre diferentes espécies. Diversos estudos têm sido feito e demonstrado que o uso de gene codificadores de proteínas são como marcadores filogenéticos têm sido utilizados como marcadores filogenéticos para a classificação e identificação de bactérias lácticas. A utilização de genes informativos, como o gene 16S rRNA, são considerados verdadeiros cronômetros evolutivos por estarem presentes em todos os seres vivos pois a comparação de sua sequência entre diversos organismos permite inferências filogenéticas entre os mesmos. Isto é possível porque além de estarem presentes em todos seres vivos, são conservados do ponto de vista evolutivo mas apresentam regiões com variação de nucleotídeos nas diferentes espécies, o que permite a comparação de organismos mesmo filogeneticamente distantes. A adoção de técnicas que utilizam o gene 16S rDNA para identificar e até mesmo classificar uma bactéria foi sugerida e incentivada pelo comitê de sistemática bacteriana em 2002 (Stackebrandt et. al. 2002). Desde 2003 esta abordagem é amplamente utilizada para estudar comunidades microbianas complexas como solo, ar e água (Hongoh et. al. 2003). O gene 16S é comumente considerado como um confiável marcador filogenético para atribuir relações evolutivas entre espécies do gênero Lactobacillus 23 (Schleifer & Ludwig, 1995). Este gene ainda é o alvo mais comumente utilizado em estudos de diversidade bacteriana. Este gene é um marcador universal altamente conservado com funções bem definidas desde os primórdios da evolução e é relativamente não afetado por pressões ambientais. Isto, junto ao tamanho do gene, faz com que ele seja um bom relógio evolutivo (Kimura et al., 1997; Mohania et al., 2008). Mesmo sendo considerado um marcador universal bem conservado existem deficiências associadas ao seu uso. Primeiro, por ser bem conservado ele limita o poder de resolução. Segundo, o gene 16S rRNA aparece em diferentes espécies de bactérias em diferentes números cópias do gene (Mohania et al., 2008). Para superar estes problemas a utilização de genes classificados como housekeeping, genes constitutivos que são requeridos para a manutenção das funções celulares básicas, vem surgindo como uma alternativa para a resolução destes problemas (Naser et al., 2007). Estudos recentes in silico baseados em genomas completos têm providos as bases para o estabelecimento de uma serie de genes housekeeping que podem predizer precisamente o parentesco genômico aumentar a precisão da identificação das espécies. A necessidade por marcadores genômicos alternativos que forneçam mais altos níveis de discriminação do que o gene 16S rRNA têm levado a um sequenciamento sistemático de genes housekeeping (Coenye et al., 2005; Gevers et al., 2005; Konstatinidis & Tiedje, 2005; Naser et al., 2005a). Os estudos de análises de sequências de loco único (SLSA - Single-locus sequence analysis) focam genes codificadores de proteínas que tem evolução relativamente lenta, porem mais rapidamente do que os genes 16S rRNA, para obter uma alta resolução taxonômica (Figura 3). 24 Figura 4. Árvore filogenética feita a partir de sequências do gene 16S mostrando a posição do T Lactobacillus hammesii sp. nov. strains LP38 e LP39 entre alguns lactobacilli. (Reproduzido de Valcheva et al., 2005) Para ser utilizado para discriminação de espécies, é ideal que os gene esteja presente em uma única copia no genoma, evoluir com maior rapidez do que os genes rRNA e ser amplamente distribuídos entre os genomas bacterianos. Aqueles genes nos quais a recombinação pode conferir uma vantagem seletiva, ou genes ligados estreitamente, devem ser evitados. Além disso, estes genes devem ser informativos e com um adequado grau de resolução e fornecer variabilidade para diferenciar espécies de um gênero em particular (Naser et al., 2007). A utilização de genes housekeeping tal como o gene que codifica a subunidade α da RNA polimerase (gene rpoA) e a enzima fenilalanina-tRNA sintetase bacteriana (gene pheS) têm se mostrado como um robusto sistema para a identificação de todas as espécies reconhecidas de Enterococcus (Naser, et al., 2005b) e de todas as espécies do gênero Lactobacillus (Naser et al., 2007). A partir 25 da análise dos genes pheS e ropA é possível fazer a diferenciação efetiva das espécies de Lactobacillus com um alto nível de discriminação quando comparado com o gene 16S rRNA. RecA é uma pequena proteína essencial para o reparo e manutenção do DNA. Está envolvida no processo de recombinação homóloga do DNA, indução do sistema SOS (um sistema de reparo do DNA pós-replicação), e indução dos mecanismos de reparação de lesões no DNA. Devido ao seu papel fundamental, o gene recA é ubíquo, e seu produto tem sido proposto como um marcador filogenético para espécies muito próximas (Felis et al., 2001; Torriani et al., 2001). O uso desta metodologia para a diferenciação de três espécies do grupo Lactobacillus plantarum (L. plantarum, L. pentosus e L. paraplantarum) teve êxito e mostrou um alto poder discriminatório no uso deste gene para separar espécies de difícil diferenciação, tais como Bifidobacterium longum e Bifidobacterium infantis. Validando a utilização deste gene como marcador filogenético-taxonômico para espécies relacionadas, abrindo uma nova possibilidade para uma rápida e segura identificação de LAB importantes de uso industrial. Silvers et al. (2003) determinou as sequências de nucleotídeos do gene atpD que codifica a subunidade β da ATPase das bactérias Oenococcus oeni, Leuconostoc mesenteroides subsp. mesenteroides, Pediococcus damnosus, Pediococcus parvulus, Lactobacillus brevis e Lactobacillus hilgardii. A árvore obtida a partir destas sequências indicaram que O. oeni e L. mesenteroides subsp. mesenteroides formaram um grupo bem separados de P. damnosus e P. parvulus e das duas espécies de Lactobacillus. Tapp et al. (2003) em seus estudos, do gene rnpB, que é universalmente presente em bactérias Gram-positivas com baixo conteúdo de G+C e, que codifica a 26 subunidade de RNA da endoribonuclease P, revelou que o mesmo se mostrou adequado para analises filogenéticas de taxa relativamente próximos e como uma potencial ferramenta para discriminação de espécies dentro do gênero Streptococcus. Em outro estudo, o uso de sequências parciais do gene rpoB, que codifica a subunidade altamente conservada da RNA polimerase bacteriana. As sequências do gene rpoB mostraram um maior poder discriminatório do que sequências do gene 16S rRNA para a identificação de LAB dos gêneros Streptococcus, Enterococcus, Gemella, Abiotrophia e Granulicatella (Drancourt et al., 2004). 2.4.8. Rep-PCR A técnica Rep-PCR faz uso de sequências oligonucleotídicas iniciadoras complementares de sequências de DNA repetitivas muito conservadas e presentes em numerosas cópias no genoma da maioria das bactérias Gram-negativas e de várias bactérias Gram-positivas. Foram identificadas três famílias de sequências repetitivas: 1) as sequências REP (Repetitive Extragenic Palindromic elements) com 35-40 pares de bases, as quais são altamente conservadas em várias espécies bacterianas; 2) as sequências ERIC (Enterobacterial Repetitive Intergenic Consensus elements) de 124-127 pares de bases, as quais contêm um elemento repetitivo central invertido altamente conservado e que estão localizados nas regiões extragênicas do genoma bacteriano; 3) o elemento BOX de 154 pares de bases. A amplificação das sequências entre cada um destes elementos repetitivos têm sido utilizadas para gerar perfis de DNA de várias espécies bacterianas, tanto 27 Gram-positivas quanto Gram-negativas (Vuyst et al., 2008). Sequências com unidades de repetição (GTG)5 aparecem distribuídas amplamente nos genomas de diversos grupos de bactérias. A amplificação das sequências destes elementos repetitivos gera padrões de impressões genéticas (DNA fingerprints) específicos para diversas espécies de Lactobacillus (Gevers et al., 2001) e Enterococcus (Švec et al., 2005), permitindo uma especiação e tipagem rápida, precisa e confiável. A Rep-PCR consiste em uma técnica com alto poder discriminatório, baixo custo, adequado para discriminação de linhagens e pode ser considerada como uma ferramenta confiável para a classificação e tipagem de um grande numero de bactérias Gram-negativas e várias bactérias Gram-positivas (Gevers et al., 2001). Hyytiä-Trees et al. (1999) sugeriu que um adequado nível de discriminação entre linhagens de Lactobacillus sakei pode ser conseguido com o uso combinado da Rep-PCR usando primers BOX e REP e RAPD. Enquanto que, Sohier et al. (1999) demonstrou a aplicabilidade da rep-PCR para identificar as espécies de Lactobacillus hilgardii e Lactobacillus brevis. A técnica de Rep-PCR utilizando o iniciador (GTG)5 é uma ferramenta rápida, de fácil perfomance e, reprodutível para a diferenciação de uma grande variedades de Lactobacillus associados a alimentos no nível de espécie, subespécie e, potencialmente, ate o nível de linhagem, com um protocolo de desempenho simples. Esta técnica consiste em uma ferramenta genotípica promissora para a especiação e tipagem rápida e confiável de Lactobacillus entre outras LAB em indústrias de fermentação (Gevers et al., 2001, Lucena et al., 2010). Vuyst et al. (2008) fez a utilização do primer (GTG)5 que permitiu a identificação da impressão genética das bactérias ácido acético (AAB - Acetic Acid Bacteria) durante um dia de trabalho. Fazendo uso desta ferramenta para a 28 classificação e identificação de AAB no nível de espécies, bem como a para a caracterização destas bactérias ao nível intraespecífico. A utilização do primer (GTG)5 pode aumentar o conhecimento a cerca da ecologia biodiversidade tanto das LAB como das AAB e ajudar a determinar com mais precisão o seu comportamento de crescimento e benéficos ou indesejáveis papeis durante várias etapas da fermentação de alimentos. Diante disto, a taxonomia dos procariotos vem evoluindo juntamente com os avanços nas áreas de Genética de Populações de microrganismos, Ecologia, Genômica, Transcripitônica e na facilidade de obtenção de dados sequênciamento. Isto faz com que os conceitos de espécie sejam revistos. Avanços na área molecular leva a crer que o futuro da taxonomia e sistemática microbiana terá como base a abordagem de análise de sequências de DNA (Gevers et al. 2005). Assim esta abordagem pode ser utilizada para o estudo de populações complexas de microganismos como a do processo de fermentação etanólica industrial. O conhecimento das espécies bacterianas que entram no processo fermentativo contribuirá para um maior controle das contaminações bacterianas que diminuem o rendimento da produção de etanol. Isto trará benefícios econômicos para o setor industrial. 29 3. Objetivos 3.1 Geral Este trabalho teve como principal objetivo a identificação de bactérias presentes no caldo de cana-de-açúcar e destilarias da região Nordeste do Brasil. 3.2 Específicos Identificar quais grupos bacterianos entram no processo fermentativo do caldo de cana-de-açúcar. Verificar quais as bactérias lácticas mais comuns quais problemas podem ser identificados como sendo causados por sua presença durante a fermentação. Demonstrar a eficácia das técnicas de ARDRA e Sequênciamento de DNA para identificação empregadas neste trabalho 30 4. Material e Métodos 4.1. Isolamento, manutenção e identificação bacteriana Amostras do caldo de cana-de-açúcar de quatro destilarias localizadas nos estados da Paraíba e Pernambuco, região Nordeste do Brasil, foram coletadas durante o período da safra de 2007-2008. As coletas foram realizadas nas usinas Giasa (Pedras de Fogo-PB), Japungu e Miriri (Santa Rita-PB) e Trapiche (SirinhaémPE) onde 100mL do caldo de partida foi coletado em sacos estéreis e transportados em gelo para o laboratório onde foram executados os experimentos. Inicialmente amostras foram diluídas em solução salina 0,85% estéril até 3x10-4 e semeadas em placas de Petri contendo o meio de cultura MRS® sólido acrescido de ciclohexamida (0,2 gL-1) para inviabilizar o crescimento de fungos. As placas foram incubadas em jarras de anaerobiose a 37ºC com geradores de anaerobiose Anaerocult A (Merk) (Skinner e Leathers, 2004). 4.1.1 Isolamento e purificação das Culturas Após o crescimento, foram selecionadas as placas nas quais havia um crescimento de aproximadamente 100 colônias com uma boa distribuição na placa. As colônias foram caracterizadas segundo a sua morfologia, e o isolamento realizado por amostragem estratificada segundo cada morfotipo. Os isolados foram mantidos em glicerol 30% a -20oC. As colônias foram semeadas em meio MRS líquido para o crescimento e posterior extração do material genético das mesmas. 31 4.2. Tipagem Genética 4.2.1. Extração de DNA O método extração de DNA foi adaptado do protocolo de Moreira et al. (2005). Resumidamente, os isolados obtidos foram semeados em 10mL do meio MRS líquido em tubos Falcon™ de 15mL. As células foram centrifugadas por 10min a 4.000 rpm, e o sedimento celular foi ressuspenso em 1ml de LiCl 5M, transferidos para microtubos de 1,5mL e incubados por 1h sob agitação constante. Após incubação, as células foram centrifugadas a 4.000 rpm em microcentrífuga de bancada. O sedimento foi ressuspenso em 1ml do tampão TE10 (Tris 50mM, EDTA 10mM, pH 8,0) acrescido de 3 L de solução de lisozima (10mg/mL) e incubados por 1h e posteriormente centrifugados por 10min a 4.000 rpm. O sedimento foi novamente ressuspensos em TE10 (540 µL) acrescido de 60µL de solução de SDS (10%) para permitir a lise celular (entre cada fase de ressuspensão o sedimento foi lavado em água mili-Q). Os tubos foram centrifugados mais uma vez e o sobrenadante foi transferido para outro microtubo onde foi acrescido de clorofane (fenol-clorofórmio + álcool isoamílico, 1:1) na quantidade de 600μL por microtubo e centrifugado por 10min a 14.000 rpm, ao sobrenadante retirado e transferido para um novo microtubo onde foi acrescido 600μL de clorofil (clorofórmio-álcool isoamílico, 24:1). O DNA foi precipitado em 600μL de álcool isopropanol 100%, centrifugado a 4°C a 14.000 rpm por 30min. Mais uma vez ressuspendido em 500μL de etanol 70% e centrifugado a 4°C a 14.000 rpm por 10min e, por fim ressuspendido em 100μL de água estéril. 32 4.2.2. ARDRA-PCR A amplificação da região espaçadora 16S-23S do gene do rRNA seguiu o protocolo descrito por Moreira et al. (2005), utilizando o primer 16-1A, e o primer 231B, (Tabela 2) (Tilsala-Timisjarvi e Alatossava, 1997). Para a amplificação realizou-se uma reação em cadeia da polimerase (PCR) com as seguintes condições: 10pmol de cada primer, 0,2 mM de cada dNTP, tampão de reação (1X), com 1,5mM de MgCl2 e 5U de Taq DNA polimerase (Invitrogen Life Technologies) para um volume final de 50µL. A amplificação consistiu de um ciclo de desnaturação de 2 minutos a 94°C, seguido de 35 ciclos de desnaturação a 94°C por 30seg, anelamento a 55°C por 1min, extensão a 72°C por 1min e extensão final a 72°C por 10min. Os produtos da amplificação foram separados em gel de agarose 1,4% submetidos a 5 V/cm por 45min em tampão TBE 0,5X, corados em brometo de etídeo, e visualizados em transiluminador ultravioleta. Os produtos de amplificação foram digeridos com 12 enzimas de restrição (SphI, NcoI, NheI, SspI, SfuI, EcoRV, DraI, VspI, HincII, EcoRI, HindIII, AvrII) (Fermentas) e os fragmentos de digestão foram separados em gel de agarose 1,4% submetidos a 5 V/cm por 45min em tampão TBE 0,5X, corados em brometo de etídeo, e visualizados em transiluminador ultravioleta. Os produtos de digestão foram comparados com o banco de dados genéticos descrito por Moreira et al. (2005) (Anexo 1). 4.2.3. rep-PCR Para realizar a reação de amplificação dos elementos repetitivos do DNA foi utilizado primer (GTG)5 segundo Lucena et al. (2010). Na reação de amplificação foi 33 utilizado 0,2 pmol do primer (GTG)5; 0,2 mM de dNTP; 3 mM MgCl2; 0,025 µg/µL de BSA (Bovine Serum Albumin - soro de albumina bovina)e 1 U Taq polimerase (Invitrogen). O programa de amplificação foi composto por um período de desnaturação a 95°C por 7min, seguido por 30 ciclos de desnaturação a 95°C por 1min, anelamento a 40°C por 1min, extensão a 65ºC por 8min seguidos por um período de extensão final de 16min. O produto da PCR foi separado em gel de agarose 1,5% submetido a 5 V/cm em tampão TBE 0,5X, corado em brometo de etídeo. As imagens do foram capturadas usando um sistema de captura de imagens PhotoCapture™. A similaridade entre os padrões de amplificações foram determinadas através de análises visuais das imagens. 4.2.4. Sequênciamento dos genes 16S rRNA e pheS O gene 16S rRNA foi amplificado por PCR utilizando os primers 27F e 519R, desenhados para o anelamento nas extremidades do gene e amplamente utilizados, que amplificam um fragmento de aproximadamente 492 pb (Hauben et al. 2005) (Tabela 1). O programa térmico consistiu de 1 ciclo de desnaturação a 95°C por 3 minutos, seguidos por 30 ciclos de desnaturação a 95°C por 1min; anelamento a 51°C por 1min e 30s; extensão a 72°C por 3min e uma extensão final a 72°C por 30min no termociclador Biocycler (MJ96+). Para o gene pheS foram utilizados os primers 21-F, e 22-R ou 23-R (Tabela 2) (Naser et al. 2005b). As reações para amplificações foram feitas utilizando 0,5 µM de cada primer, 0.2 mM de cada dNTP, tampão de reação (1X) com 1,5 nM de MgCl2 e 1U de Taq polimerase (Invitrogen). As amplificações foram feitas em no termociclador Biocycler (MJ96+) programado para 1 ciclo de desnaturação de 5 34 minutos a 95°C, seguidos por 3 ciclos de desnaturação 95°C por 1 minuto, anelamento a 46°C por 2 minutos e 15 segundos, extensão a 72°C por 1 minuto e 15 segundos e 30 ciclos de desnaturação a 95°C por 35 segundos, anelamento a 46°C por 1 minuto e 15 segundos, extensão a 72°C por 1 minuto e 15 segundos, e uma extensão final de 7 minutos a 72°C. Os produtos da PCR foram purificados com o kit Wizard® SV Gel and PCR Clean-Up System (Promega). Cerca de 50 a 100 ng do produto da purificação foram misturados com a reação de marcação BigDye™ Terminator v3.1 Cycle Sequencing Kit (Applied Biosystems), seguindo as instruções do fabricante na presença de um dos primers descritos na concentração de 1,2µM. O sequênciamento foi realizado na Plataforma Genômica do Laboratório Central do Centro de Ciências Biológicas da Universidade Federal de Pernambuco, utilizando o sequenciador 3500 Genetic Analyser (Applied Biosystems - USA). A verificação da qualidade das sequências geradas foi feita pelo programa Sequencing Analysis (Applied Biosystems), que gerou arquivos com o eletroferograma de cada sequência. Inicialmente as sequências fora editadas através do programa Pregap4 pertencentes ao Standen Package (Standen, 1998) e submetidas à busca de similaridade no banco de dados do NCBI, utilizando a ferramenta Blast N (Altschul et al., 1997). A identificação bacteriana foi assumida se a sequência de consulta mostrou similaridade >90% com a sequência de nucleotídeos da linhagem tipo para o gene pheS (Naser et al., 2007) e similaridade > 97% para o gene 16S (Gevers et al., 2005). Após a edição foi feito o alinhamento múltiplo utilizando a ferramenta ClustalW do programa MEGA 4 (Tamura et al., 2007), que também foi utilizado para a construção e visualização das árvores filogenéticas geradas. Os agrupamentos 35 baseados nas inferências filogenéticas foram construídos aplicando o algoritmo Neighbour-Joining (Saitou e Nei, 1987) com testes estatísticos de bootstrap com 2000 replicações. Tabela 2. Primers utilizados nas reações de PCR e sequenciamentos. Primer Sequência (5'→3')a Posição Referencia b Tilsala-Timisjarvi e Alatossava, 1997 16-1A GTCGGAATCGCTAGTAATCG 1361 23-1B GGGTTCCCCCATTCGGA 127 b Tilsala-Timisjarvi e Alatossava, 1997 21-F CAYCCNGCHCGYGAYATGC 557 c Naser et al. 2005b 22-R CCWARVCCRAARGCAAARCC 1031c Naser et al. 2005b c Naser et al. 2005b 23-R GGRTGRACCATVCCNGCHCC 968 27F AGAGTTTGATCCTGGCTCAG 8-27c Hauben et al. 2005 519R GTATTACCGCGGCTGCTG 536-519c Hauben et al. 2005 a Y:C ou T; N:A,C,G ou T; H:A, C ou T; W:A ou T; R:A ou G; V:A, C ou G Posição de ligação no cromossomo de L. casei c Posição de ligação no cromossomo de E. coli b 36 5. Resultados 5.1. Análise Molecular das Bactérias isoladas do Caldo de Cana Como forma de identificação os perfis de ARDRA obtidos neste trabalho foram comparados com um banco de dados gerados por Moreira et al. (2005) e Lucena (2010). Foram analisados neste trabalho através da ARDRA um total de 141 isolados oriundos do caldo de partida das quatro destilarias estudadas durante a safra 2007-2008, sendo 25 pertencendo a Giasa; 52 isolados a Japungu; 44 a Miriri e 20 da Trapiche. Pelo uso da técnica de ARDRA foi possível identificar 33,33% do total de isolados de todas as destilarias (Anexo 2). Figura 5. Gêneros de LAB encontrados no caldo de partida das destilarias Japungu, Miriri e Giasa durante a safra 2007/2008. Neste estudo foi possível encontrar no caldo de alimentação do processo de fermentação seis gêneros de LAB (Figura 5). A maioria dos isolados encontrados encontradas pertenceram ao gênero Lactobacillus, que é comumente encontrado durante a produção de álcool nas destilarias do Nordeste brasileiro (Lucena et al., 2010). 37 A diversidade de LAB encontradas nas destilarias estudadas foi grande. Foram encontradas 15 espécies de bactérias utilizando as técnicas descritas na seção anterior. Destacam-se as espécies L. fermentum, Leuc. mesenteroides que apresentaram a maior abundancias nas amostras coletadas, cada um representou 24% dos isolados identificados e W. confusa que teve uma frequência também elevada (20%) porém menor que as primeiras (Figura 6). As demais espécies foram encontradas em uma frequência bem abaixo das bactérias que predominaram durante o período de estudo. Figura 6. Distribuição percentual das espécies de bactérias lácticas identificadas utilizando as técnicas de ARDRA e sequênciamento do genes 16S e pheS. 38 5.1.1. Destilaria Giasa Nesta destilaria foram feitas três coletas durante o período da safra, e puderam ser identificados através da análise do perfil de restrição do rDNA amplificado 65% dos isolados (Anexo 2), sendo o gênero Lactobacillus o mais frequente, representado pelas espécies L. fermentum (44%) e L. delbrueckii (4%) seguidas por Lactococcus, representado por Lac. lactis (16%). Cerca de 36% dos isolados desta destilaria não puderam ser identificados utilizando a ARDRA (Figura 7). Figura 7. Bactérias lácticas identificadas a partir da análise do perfil de restrição do rDNA amplificado ARDRA. N=25 Os isolados não identificados foram submetidos a análise do sequênciamento do genes 16S e pheS. A identificação destes isolados se deu primeiramente com o sequênciamento do gene 16S, com as sequências dos isolados apresentando similaridade de 97% ou mais com sequências depositadas no Genebank. Dos isolados que tiveram o gene 16S sequenciados 95% foram identificados como pertencendo a espécie L. fermentum, enquanto que 5% foram identificados como Oenococcus sp. (Figura 8). 39 O isolado GCP7.2.1 foi identificado como sendo um Oenococcus sp. devido a problemas de sequência e foi o único com similaridade abaixo de 97% de similaridade com a espécie mais próxima ao mesmo (Oenococcus kitaharae). Figura 8. Bactérias Lácticas identificadas a partir da análise de sequências do gene 16S. Figura 9. Árvore filogenética construída a partir do gene pheS utilizando o método NeighbourJoining dos isolados da destilaria Giasa. Os isolados identificados como sendo L. fermentum a partir da análise do sequênciamento do gene 16S foram confirmados pelo sequênciamento do gene pheS como pertencentes a esta espécie (Figura 9) onde pode-se observar a formação de um agrupamento composto pelos isolados sequenciados. Nesta nova análise foi possível identificar o isolado GCP7.3.15 como pertencendo a espécie L. fermentum, devido a esta 40 análise da sequência do gene pheS. A Tabela 3 mostra os isolados identificados pela análise das sequências dos genes 16S e pheS. O sequênciamento se mostrou uma ótima ferramenta para a identificações destes isolados. Durante a safra foi encontrada uma baixa diversidade bacteriana (Figura 7), fato que pode ser explicado pelo pequeno número de colônias que foram isoladas. A dinâmica da população bacteriana foi variável, onde não foi possível encontrar as mesmas espécies ao longo das três coletas. Tabela 3. Isolados identificados a partir do sequênciamento dos genes 16S e pheS Isolado Marcador Identificação GCP7.3.7 16S Lactobacillus fermentum GCP7.3.8 16S Lactobacillus fermentum GCP7.3.14 16S Lactobacillus fermentum GCP7.3.15 pheS Lactobacillus fermentum GCP7.3.16 16S Lactobacillus fermentum GCP7.3.17 16S Lactobacillus fermentum GCP7.3.18 16S Lactobacillus fermentum GCP7.3.19 16S Lactobacillus fermentum Figura 10. Dinâmica da população bacteriana da destilaria Giasa durante a safra 2007/2008 41 5.1.2. Destilaria Japungu Três coletas foram realizadas nesta destilarias e após o isolamento foram obtidos 54 isolados. A maior parte dos isolados não puderam ser identificados pela ARDRA, apenas 35% deles foram identificados (Figura 11). Quatro gêneros de LAB foram encontrados nas coletas realizadas nesta destilaria. Houve uma predominância do gênero Leuconostoc (56%) seguidos por Wissella (38%), Lactobacillus (4%) e Staphylococcus (2%). Grande parte dos isolados não apresentou o perfil de amplificação da região espaçadora típica do gênero Lactobacillus (Figura 2), mas apresentaram diversos perfis de ARDRA que não haviam sido observados no banco de dados já estabelecido na literatura. Figura 11. Bactérias lácticas identificadas pela análise do perfil de digestão do rDNA amplificado. N=54 Os isolados não identificadas foram submetidas ao sequênciamento do gene 16S para uma nova tentativa de identificação. A partir da análise do sequênciamento foram 42 identificados todos as bactérias lácticas que não possuíam perfis de ARDRA no banco de dados (Tabela 4). Tabela 4. Isolados identificados através da análise das sequências parciais dos genes 16S e pheS durante a safra 2007/2008. Isolados Marcador Identificação JCP7.1.1 pheS Weissella cibaria JCP7.1.2 16S Leuconostoc garlicum JCP7.1.3 pheS Leuconostoc garlicum JCP7.1.4 pheS Weissella cibaria JCP7.1.5 16S Weissella cibaria JCP7.1.7 16S Weissella cibaria JCP7.1.8 16S Leuconostoc citreum JCP7.1.9 16S Leuconostoc garlicum JCP7.1.12 16S Leuconostoc garlicum JCP7.1.14 16S Staphylococcus epidermidis JCP7.1.16 16S Leuconostoc garlicum JCP7.1.17 16S Leuconostoc citreum JCP7.1.20 pheS Weissella cibaria JCP7.1.21 16S Weissella confusa JCP7.2.2* 16S Weissella confusa JCP7.2.3 16S Leuconostoc garlicum JCP7.2.8* 16S Weissella confusa JCP7.2.9* 16S Weissella confusa JCP7.2.10 16S Weissella confusa JCP7.2.11* 16S Weissella confusa JCP7.2.12* 16S Weissella confusa 43 JCP7.2.14* 16S Leuconostoc citreum JCP7.2.15* 16S Weissella confusa JCP7.2.16* 16S Leuconostoc citreum JCP7.2.17* 16S Leuconostoc citreum JCP7.2.18 16S Leuconostoc citreum JCP7.2.19 16S Leuconostoc citreum JCP7.2.20* 16S Leuconostoc citreum JCP7.3.3 16S Leuconostoc mesenteroides JCP7.3.7 pheS Leuconostoc mesenteroides JCP7.3.14 16S Leuconostoc mesenteroides JCP7.3.16* 16S Weissella cibaria JCP7.3.20 16S Leuconostoc mesenteroides * Isolados que tiveram suas identificações confirmadas pelo sequenciamento do gene pheS. A dinâmica das bactérias lácticas nas coletas realizadas durante a safra de 2007/2008 é mostrada na Figura 12. Figura 12. Dinâmica da população bacteriana da destilaria Japungu durante a safra 2007/2008 44 Analisando a Figura 12 pode-se observar que há um aumento na diversidade das bactérias nesta destilaria, principalmente na primeira coleta onde predominou o isolado identificado como W. cibaria (46%) seguidos por Leuc. mesenteroides (31%), Leuc. citreum, w. confusa e S. epidermidis todos com 8% dos isolados identificados. Na segunda coleta houve uma queda no numero de espécies, mas com um aumento na frequência de W. confusa (62%) e Leuc. citreum (38%) das espécies identificadas. Na terceira coleta verificou-se que o Leuc. mesenteroides foi a espécie de maior frequência com de 73% seguidos pelo aparecimento do L. fermentum, que só havia sido identificado pela ARDRA, representando 23% dos isolados seguido pela W. cibaria identificada com 4% dos isolados identificados. Um dos fatores que pode explicar este aumento é por esta destilaria receber cana de vários produtores, forma como é feita a coleta por cada um destes produtores e o contaminação dos equipamentos responsáveis pela moagem da cana. Durante a safra foram identificadas bactérias pertencentes à espécie Leuconostoc garlicum. Esta foi descrita por Kim e Kyung (2003), isoladas a partir do alho utilizado na culinária (Allium sativum) porém não tiveram seu artigo publicado e nem seu epíteto reconhecido mas já existe inúmeras sequências depositadas no Genebank. Não existe estudos anteriores que tenham encontrado esta espécie no caldo de cana para produção do etanol. Com estudo filogenético feito a partir das sequências obtidas foi possível obter a árvore filogenética observada na Figura 13 a partir do agrupamento tipo Neighbor Joining. 45 Nela é pode-se observar que os resultados obtidos pelo sequenciamento são robustos e consistentes com os resultados da análise utilizando a ferramenta BLAST do GeneBank. Figura 13. Árvore filogenética construída a partir das sequências de nucleotídeos do gene 16S, utilizando o agrupamento do tipo Neighbor joinning, dos isolados encontrados no caldo de cana da destilaria Japungu durante a safra de 2007/2008. 46 5.1.3 Destilaria Miriri Durante o período da safra fora efetuadas três coletas nesta destilaria, espécies de três gêneros foram encontradas durante esta safra. Houve um predomínio do gênero Weissella (46%), seguido por Lactobacillus e Leuconostoc com 38% e 16% das espécies encontradas, respectivamente. Em uma primeira análise feita pela ARDRA de todos os isolados apenas 32% foram identificados de acordo com seu perfil de restrição do DNA ribossomal amplificado (Figura 14). A grande maioria não pode ser identificada devido a divergências entre os perfis encontrados neste trabalho e na literatura. Porém este método pode identificar com grande precisão as espécie L. nagelli, L. salivarus e L. fermentum. Figura 14. Bactérias lácticas identificadas pela análise do perfil de digestão do rDNA amplificado. N=54 Os 68% dos isolados não identificados foram submetidos ao sequenciamento dos genes pheS e 16S. E a partir da análise das sequências obtidas através da busca de similaridade entre sequências do Genebank foram identificadas as bactérias lácticas mostradas na tabela 5. 47 Tabela 5. Isolados identificados a partir do sequênciamento dos genes 16S e pheS oriundos da destilaria Miriri durante a safra 2007/2008 Isolado Marcador Identificação MCP7.1.1 16S Weissella cibaria MCP7.1.4 pheS/16S Weissella confusa MCP7.1.5 16S Leuconostoc mesenteroides MCP7.1.6 16S Leuconostoc pseudomesenteroides MCP7.1.7 16S Weissella confusa MCP7.1.9 16S Leuconostoc pseudomesenteroides MCP7.1.11 16S Leuconostoc lactis MCP7.1.13 16S Leuconostoc pseudomesenteroides MCP7.1.14 16S Leuconostoc pseudomesenteroides MCP7.1.17 16S Leuconostoc pseudomesenteroides MCP7.1.19 pheS/16S Weissella confusa MCP7.1.20 pheS/16S Weissella confusa MCP7.3.1 pheS16S Weissella confusa MCP7.3.2 pheS/16S Weissella confusa MCP7.3.3 pheS/16S Weissella confusa MCP7.3.4 pheS/16S Weissella confusa MCP7.3.5 pheS/16S Weissella confusa MCP7.3.6 pheS/16S Weissella confusa MCP7.3.7 pheS/16S Weissella confusa MCP7.3.8 pheS/16S Weissella confusa MCP7.3.9 pheS/16S Weissella confusa MCP7.3.10 pheS/16S Weissella confusa MCP7.3.11 pheS/16S Weissella confusa MCP7.3.12 16S Leuconostoc lactis 48 Durante a safra foi possível observar que houve uma queda no numero de espécies de LAB encontradas (Figura 15). A diversidade na primeira coleta foi a maior observada entre as destilarias estudadas com seis espécies encontradas. A espécies Leuc. pseudomesenteroides foi a predominante com cerca de 36% dos isolados seguidos por W. confusa, (22%), L. salivarus e L. nagelii ambos com 14% e W. cibaria e Leuc. mesenteroides com 7% cada. Na segunda coleta só foi encontrado isolados pertencentes a espécie L. fermentum, isso se deve ao fato dela ter apresentado um número reduzido de isolados coletados (N=09). Figura 15. Diversidade da população bacteriana presente no caldo de cana da destilaria Miriri durante a safra 2007/2008. Apenas duas espécies foram encontradas na terceira coleta com a predominância da bactéria W. confusa (92%) e a espécie L. fermentum encontrada em 8% dos isolados desta coleta. A partir da análise filogenética, através da construção de arvores filogenéticas baseadas nas sequências de nucleotídeos obtidas através do sequenciamento do gene 49 16S foi possível fazer a identificação das espécies bacterianas que não apresentaram o perfil de amplificação da região espaçadora dos genes 16S e 23S diferentes do apresentado pelas bactérias do gênero Lactobacillus (Figura 17). A análise da árvore da maior consistência à análise feita através da busca de similaridade entre as sequências depositadas no Genebank. Figura 16. Árvore filogenética construída a partir das sequências do gene 16S dos isolados da destilaria Miriri utilizando o agrupamento Neighbor Joining. 50 5.1.4 Destilaria Trapiche Nesta destilaria foi realizada apenas uma coleta durante a safra, com um total de 20 isolados dos quais apenas dois não obtiveram crescimento no meio liquido. Três gêneros foram encontrados com Leuconostoc se destacando dentre os demais com onze isolados identificados representando 75% dos mesmos seguidos por Lactobacillus (15%) e Weissella (10%). Através da ARDRA foram identificados 17% dos isolados (Anexo 2). Todos foram pertencentes a espécie L. plantarum. Os outros 83% dos isolados encontrados não possuíam perfil de restrição do rDNA amplificado no banco de dados, sendo necessário fazer uma análise de sequências de nucleotídeos. As identificações são mostradas na tabela 6. Tabela 6. Isolados identificados a partir do sequênciamento dos genes 16S e pheS encontrados no caldo de partida da destilaria Trapiche. Isolado TCP7.1.1 TCP7.1.2 TCP7.1.3 TCP7.1.4 TCP7.1.5 TCP7.1.6 TCP7.1.7 TCP7.1.8 TCP7.1.9 TCP7.1.10 TCP7.1.11 TCP7.1.13 TCP7.1.14 TCP7.1.15 TCP7.1.16 TCP7.1.17 Marcador 16S/pheS 16S 16S 16S 16S 16S 16S 16S 16S 16S 16S 16S 16S pheS 16S 16S Identificação Weissella confusa Leuconostoc mesenteroides Leuconostoc mesenteroides Leuconostoc mesenteroides Leuconostoc mesenteroides Lactobacillus plantarum Leuconostoc mesenteroides Leuconostoc mesenteroides Lactobacillus plantarum Lactobacillus plantarum Leuconostoc mesenteroides Leuconostoc mesenteroides Leuconostoc mesenteroides Weissella paramesenteroides Leuconostoc mesenteroides Leuconostoc mesenteroides 51 Figura 17. Árvore filogenética construída a partir das sequências do gene 16S dos isolados da destilaria Trapiche utilizando o agrupamento Neighbor Joining. 52 5.2 Análise Intraespecífica dos isolados A técnica de rep-PCR com o uso do primer (GTG)5 foi utilizada com a finalidade de diferenciar ao nível intraespecífico os isolados das espécies W. confusa. e Leuc. Mesenteroides identificados na safra 2007/2008. Este isolados foram os que apresentaram uma maior abundância durante as análises, ficando atrás apenas dos isolados de L. fermentum (Figura 6). 5.2.1 Weissella confusa Esta espécie de bactéria foi encontrada apenas nas destilarias Japungu, Miriri e Trapiche. Os perfis de amplificação da região espaçadora 16S-23S obtidos destes isolados foram diferentes dos encontrados no gênero Lactobacillus, apresentando uma quarta banda entre o primeiro e o segundo espaçador com aproximadamente 620pb (Figura 18). Além disso, seu espaçador menor apresentou um tamanho superior com aproximadamente 580pb contrastando com o perfil característicos dos Lactobacillus onde o menor espaçador apresenta aproximadamente 503pb. Isso se mostrou uma constante nos isolados do gênero Weissella, podendo ser descrito como uma característica que os diferencia dos Lactobacillus. 1 2 3 4 5 100pb 600bp Figura 18. Perfil de amplificação da região espaçadora 16S-23S dos isolados identificados como pertencentes ao gênero Weissella. Marcador de peso molecular (100bp Biolabs) 53 Estes isolados apresentaram diferentes perfis de amplificação e de restrição do rDNA amplificado, por isso, foi feita uma análise intraespecífica de alguns dos isolados representativos encontrados. Para isso, foi utilizada a técnica de rep-PCR com a utilização do primer (GTG)5 com a finalidade de diferenciar estes isolados que foram detectados nas destilarias estudadas. Foi observado que mesmo em isolados de uma mesma coleta existe diferenças entre os mesmos sugerindo uma diferenciação intraespecífica. Na figura 20 pode-se ver que em coletas feitas na três destilarias que os isolados de W. confusa apresentam perfis de rep-PCR semelhantes, como no caso dos isolados 6, 7, 8, 9, 13 e 14 que apresentam um perfil bem característico e distinto dos demais. Mesmo tendo este perfil característico eles apresentaram mudanças sutis que não são suficientes para afirmar que pertencem a linhagens distintas. Os isolados 2 e 3 formam outro grupo distinto, com conjuntos de bandas bem diferenciados. Da mesma forma que os isolados anteriores, os isolados 10, 11 e 12 também apresentam um perfil diferente. Apesar de também apresentarem diferenças entre si estas não são relevantes e pode-se afirmar que pertencem a uma mesma linhagem. Os isolados 1, 4 e 5 apresentaram perfis totalmente distinto entre si e os demais, com conjuntos de bandas singulares a cada isolado. Estes pode-se dizer que são linhagens de W. confusa distintas das outras encontradas. 54 1 2 3 4 5 6 7 8 9 10 11 12 13 14 100pb Figura 19. Perfis representativos de rep-PCR dos isolados das destilarias Japungu, Miriri e Trapiche Identificados como W. confusa. (1- TCP7.1.1; 2- JCP7.2.9; 3- JCP7.2.10; 4- JCP7.1.21; 5- MCP7.3.17; 6- MCP7.3.1; 7- MCP7.3.2; 8- MCP7.3.3; 9- MCP7.3.4; 10- MCP7.3.5; 9- MCP7.3.6; 10- MCP7.3.7; 11MCP7.3.8; 12- MCP7.3.9; 13- MCP7.3.10; 14- MCP7.3.11). 5.2.2 Leuconostoc mesenteroides Esta espécie também foi identificada em três das destilarias estudadas (Japungu, Miriri e Trapiche). Bem como a espécie W. confusa, os isolados de Leuc. Mesenteroides, apresentaram um perfil de amplificação da região espaçadora 16S-23S distintos, apresentando uma única banda com tamanho aproximado de 620pb (Figura 20). O isolado 5 tem o perfil do gênero Weissella. Através do rep-PCR foi possível analisar seus perfis e verificar que os isolados de destilarias diferentes apresentaram perfis distintos. Na figura 22 onde mostra o rep-PCR de isolados das destilarias Japungu, Miriri e Trapiche fica evidente a diferença entre os perfis. Os isolados 5, 6 e 7 são os que mais se 55 assemelham entre si onde pode-se dizer que são pertencentes a uma mesma linhagem. Os outros isolados ao que parece são linhagens diferentes de Leuc. mesenteroides. 1 2 3 4* 5 100pb Figura 20. Perfil de amplificação da região espaçadora 16S-23S dos isolados identificados como pertencentes ao gênero Leuconostoc. Marcador de peso molecular (100pb Biolabs). (1 - TCP7.1.7; 2 - TCP7.1.8; 3 - TCP7.1.9; 4 - TCP7.1.10; 5 - TCP7.1.11). * Isolado identificado como Weissella confusa 1 2 3 4 5 6 7 8 9 10 100pb Figura 21. Perfis representativos de rep-PCR dos isolados das destilarias Japungu e Trapiche Identificados como Leuc. mesenteroides (100pb Biolabs). (1- MCP7.1.5; 2- JCP7.3.3; 3- JCP7.3.14; 4- JCP7.3.20; 5- TCP7.1.2; 6- TCP7.1.3; 7- TCP7.1.4; 8- TCP7.1.17; 9- TCP7.1; TCP7.1.5) 56 6. Discussão Os resultados obtidos neste trabalho demonstraram que LABs são comumente encontradas no processo fermentativo e, que no caldo de partida utilizado por destilarias produtoras de etanol do Nordeste do Brasil há uma predominância das bactérias lácticas pertencentes ao gênero Lactobacillus. Estes resultados são inédito quanto a entrada do substrato no processo fermentativo mas ao mesmo tempo coerentes, pois coincidem com os dados encontrados na literatura onde foram estudados a produção de etanol em destilarias nos Estados Unidos com a utilização do milho como substrato (Skiner and Leathers, 2004) e no Brasil utilizando a cana-de-açúcar (Lucena et al. 2010). Foram encontradas no caldo de partida seis espécies de lactobacilos, destas destaca-se o L. fermentum, esta espécie é a principal responsável por grandes quedas na produção e rendimento da produção do etanol devido a capacidade que algumas linhagens desta bactéria têm de induzir a floculação e a perda de viabilidade quando em sinergia com cálcio, das leveduras dando origem ao que se chama de "pé-de-cuba" causador de entupimentos de tubulações e centrífugas de separação (Yokoya & OlivaNeto, 1991; Ludwig et al. 2001), em alguns casos o L. plantarum podem causar o mesmo efeito floculante do L. fermentum dependendo da linhagem e densidade da bactéria no processo (Garcia, 2000; Alcarde, 2001). Outros estudos demonstraram que a queda da concentração final do etanol pode chegar a 17% quando a contaminação chega a 10 8 CFU/mL de L. fermentum em alguns casos pode se chegar a 22% na queda da produção, aumento do desvio de carboidratos para a produção de glicerol e acido lático, queda de 55% da viabilidade da levedura e redução na formação de massa celular de levedura (Thomas et al., 2001; Bischoff et al., 2008; Beckner et al., 2011). 57 No inicio da fermentação o L. fermentum não foi a espécie mais abundante identificada nas destilarias, porém estudo pretéritos mostram que esta espécie tende a ter um aumento com no decorrer da fermentação e que ela é bastante adaptadas as condições do processo. Além desta, outras espécies de Lactobacillus identificadas neste estudo já haviam sido detectadas no processo fermentativo anteriormente, como: L. nagelii e L. satsumensis que são organismos versáteis, que possuem um metabolismo homofermentativo anaeróbico facultativo e com capacidade de fermentar pentoses e hexoses (ribose e arabinose) a acido láctico. Foi demonstrado por Chin e Ingledew (1994) os efeitos das LAB sobre a S. cerevisiae pela inoculação de L. fermentum e L. delbrueckii, onde foi verificado uma queda na viabilidade da levedura. Também foram identificados os lactobacilos L. delbruekii, L. plantarum e L. salivarus foram encontrados nas coletas realizadas mas com uma menor frequência, mas que são comuns na fermentação alcoólica em destilarias do Nordeste do Brasil (Lucena et al. 2010). Na descrição da microbiota da tradicional fermentação alcoólica no Vietnã ficou evidente que as LAB tiveram um destaque, onde se destacaram as espécies de LAB Pediococcus pentosaceus, L. plantarum, L. brevis, W. confusa e W. paramesenteroides. Além das espécies que são produtoras de amilase (Bacillus subtilis, B. circulans, B. amyloliquefaciens e B. sporothermodurans), bactérias produtoras de acido acético (Acetobacter orientalis e A. pasteurianus) e alguns patógenos de plantas (Burkholderia 58 ubonensis, Ralstonia solanacearum e Pelomonas puraquae) foram também detectados (Thanh et al., 2008). Existe inúmeros relatos da presença de Leuconostoc (Skinner-Nemec, Nichols & Leathers, 2007; Amorim, 2005; Skinner & Leathers, 2004; Oliva-Neto & Yokoya, 1998), e alguns de Weissella (Passoth, Blonqvist & Schnürer, 2007; Skinner & Leathers, 2004) no processo de produção do bioetanol. Algumas destas espécies foram comuns na fermentação nas destilarias estudadas, como as W. confusa e W. paramesenteroides. Outras espécies encontradas foram a W. cibaria esta não havia sido encontrada na fermentação alcoólica. As espécies do gênero Leuconostoc são facilmente encontrados no inicio da fermentação mas elas raramente causam grandes infecções, por apresentar uma baixa resistência ao etanol. Kaji (1989) simulou uma fermentação mista entre a S. cerevisiae e as bactérias L. fermentum e Leuc. mesenteroides subsp. mesenteroides, apenas os lactobacilos puderam ser recuperados após a fermentação. A causa da não recuperação do Leuconostoc foi atribuída ao efeito inibitório do etanol, que a 3% foi capaz de reduzir a velocidade especifica máxima de crescimento e, em concentrações superiores a 6% houve a interrupção total do crescimento. Lucena (2010) em seus estudos sobre a diversidade genética de bactérias láticas em destilarias de álcool combustível encontrou espécies de Leuconostoc apenas nas primeiras coletas. Os resultados aqui obtidos mostram que estes organismos estão presentes no caldo de partida, porém não permanecem no processo. Um dado interessante observado nas analises foi a identificação da "espécie" Leuconostoc garlicum na destilaria Japungu (dados não mostrados). Estes isolados 59 apresentaram uma similaridade acima 97% do seu gene 16S com sequências depositadas no Genebank, todavia este nome taxonômico genérico não foi publicado a tempo para a submissão das sequências ao Genebank. A espécie foi descrita por Kim e Kyung (2003) contudo não foi publicado (dados do NCBI). Nossos dados mostram que a diversidade bacteriana encontra no caldo de partida é composta basicamente por espécies que não permanecem ao longo da fermentação. Portanto espécies como o Leuc. mesenteroides, Leuc. psendomesenteroides, Leuc citreum, Leuc. garlicum, W. cibaria, W. confusa, W. paramesenteroides, Lac. Lactis, Staphylococcus epidermidis e O. kitaharae que não afetam drasticamente a produção do bioetanol. No presente trabalho também foi avaliado a variabilidade intraespecífica de isolados de Leuc. mesenteroides e W. confusa a partir da utilização da técnica de repPCR com o uso do primer (GTG)5. Foram escolhidas estas duas espécies por ser estas as mais bem distribuídas entre as destilarias, as mais abundantes não-lactobacilos e também apresentaram perfis de amplificação da região espaçadora 16S-23S diferentes aos perfis dos lactobacilos (Figuras 19 e 21). Esta técnica foi viável para esta análise intraespecífica dos isolados oriundos do processo fermentativo. O Leuc. mesenteroides é conhecido por produzir dextrana a partir de sacarose (Kaji, 1989) isto ainda não havia sido feito com estas espécies. Nos perfis encontrados claramente foi observado que existem diferentes linhagens de W. confusa, porém os dados são conclusivos pois se trata de um novo achado tendo que ser feito estudo minuciosos sobre o efeito de cada linhagem na fermentação, da mesma forma ocorreu com os isolados de Leuc. mesenteroides, porém se sabe que estas espécies não permanecem no processo por muito tempo por não tolerarem a altas concentrações de etanol. 60 7. Conclusões A diversidade de bactérias presentes no caldo de partida utilizado no processo de fermentação do bioetanol, apresentou o gênero Lactobacillus como o principal contaminante do processo, seguido pelos gêneros Leuconostoc e Weissella. A técnica de ARDRA, baseada na análise do perfil de digestão da amplificação da região espaçadora 16S-23S, se mostrou efetiva na identificação dos lactobacilos, mas pouco eficaz na identificação de outras espécies devido ao banco de dados que ainda é pequeno. Esta técnica não pode ser utilizada isoladamente para identificação de todas as espécies bacterianas presentes no caldo de cana. O sequenciamento do gene pheS para a identificação das espécies se mostrou eficaz em apenas alguns casos, pois assim como no caso da ARDRA o banco de sequências depositadas ainda é pequeno. Sendo assim, foi necessário o sequenciamento do gene 16S que foi de maior precisão. Neste estudo, obtivemos diferentes perfis genotípicos de Leuc, mesenteroides e W. confusa, com a utilização do rep-PCR, demonstrando que esta técnica pode ser utilizada como marcador intraespecífico destas espécies. Mais estudos são necessários para uma melhor compreensão do papel de cada linhagem no processo. 61 8. Referências Bibliográficas Abbott DA, Hynes SH, Ingledew WM (2005) Growth rates of Dekkera/Brettanomyces yeasts hinder their ability to compete with Saccharomyces cerevisiae in batch corn mash fermentations. Appl Microbiol Biotechnol 2005, 66:641-647. Ahrné S and Molin, G. (1997) Restriction endonuclease analysis of total chromosomal DNA of Lactobacillus. Microecol Ther. 26: 27-30. Alcarde VE (2001) Avaliação de parâmetros que afetam a floculação de leveduras e bactérias isoladas de processos industriais de fermentação alcoólica. Universidade Estadual de Campinas - Ciências de Alimentos. 91p. Tese (Doutorado). Altschul SF, Madden TL, Schaffer AA, Zhang J, Zhang Z, Miller W and Lipman DJ (1997) Gapped BLAST and PSI-BLAST: a new generation of protein database search programs. Nucleic Acids Res 25: 3389-3402. Amorim HV, Oliveira AJ (1981) Infecção na Fermentação: como evitá-la. Açúcar e Álcool. Fermentec v.1, n 5, p. 12-18. Basso LC, Oliveira AJ, Orelli VD, Campos AD, Gallo CR, Amorim HV (1993) Dominance of wild yeast over industrial yeast strains evaluated by karyotiping technique. Yeast Newsletter 52: 50-53. Basso LC e Amorim HV (1996) Habilidade fermentativa de leveduras isoladas do processo industrial. Relatório Anual de Pesquisa em Fermentação Alcoólica. v 1, Fermentec, Piracicaba, São Paulo, Brasil, pp 52-76. Bassam BJ, Caetano-Anollés G, Gresshoff P M (1992) DNA amplification fingerprinting of bacteria. Applied Microbiology Biotechnology, 38: 70-76. Beckner M, Ivey ML, Phister TG (2011) Microbial contamination of fuel ethanol fermentations. Lett Appl Microbiol 2011 Oct;53(4):387-94. Bischoff KM, Skinner-Nemec KA and Leathers TD (2007) Antimicrobial susceptibility of Lactobacillus species isolated from commercial ethanol plants. J Ind Microbiol Biotechnol 34: 739–744. 62 Björkroth J and Korkeala H (1996) rRNA gene restriction patterns as a characterisation tool for Lactobacillus sake producingropy slime. Int J Food Microbiol 30: 293-302. Bram RJ, Young RA, Steitz JA (1980) The ribonuclease III site flanking 23S sequences in the 30S ribosomal precursor RNA of Escherichia coli. Cell 19: 393-401. Chagnaud, P., Machinis, K., Coutte, L. A., Marecat, A., Mercenier, A. (2001) Rapid PCR – based procedure to identify lactic acid bacteria: application to six common Lactobacillus species. J Microbiol Methods 44: 139-148. Charteris WP, Kelly PM, Morelli L and Collins JK (1997) Selective detection, enumeration and identification of potentially probiotic Lactobacillus and Bifidobacterium species in mixed bacterial population. Int J Food Microbiol 35: 1-27. Cherubin RA (2003) Efeito da viabilidade da levedura e da contaminação bacteriana na fermentação alcoólica. Univ. de São Paulo Piracicaba/Escola Sup. de Agricultura Luiz de Queiroz - Agronomia. 124p. Tese (Doutorado) Chin PM and Ingledew WM (1994) Effect of lactic acid bacteria on wheat mash fermentations prepared with laboratory backset. Enz Microbiol Technol, v. 16, p.311317. Ciani M, Maccarelli F and Fatichenti F (2003) Growth and fermentation behaviour of Brettanomyces/Dekkera yeasts under different conditions of aerobiosis. World J Microbiol Biotechnol 19, 419–422. Clementino MM, Filippis I, Nascimento CR, Branquinho R, Rocha CL and Martins O (2001) PCR analysis of tRNA intergenic spacer, 16S-23S internal transcrobed spacer, and randomly amplified polymorphic DNA reveal inter- and intraspecific relationships of Enterobacter cloacae strains. J Clin Microbiol 39: 3865-3870. Coenye T, Spilker T, Reik R, Vandamme P and Lipuma JJ.(2005) Use of PCR analyses to define the distribution of Ralstonia species recovered from patients with cystic fibrosis. J Clin Microbiol. Jul;43(7):3463-6. 63 Collado, M. C., Hernández, M., (2007) Identification and Differentiation of Lactobacillus, Streptococcus and Bifidobacterium species in fermented milk products with bifidobacteria. Microbiol Res. 162: 86-92. de Bruyne K. (2009) Applocation of multilocus sequence analysis and matrix assisted laser desorption/ionization-time-of-flight mass spectrometry in molecular taxonomy of lactic acid bacteria. Univesiteit Gent - Departament of Biochemistry and Microbiology. Tese (Doutorado). de Miniac M (1989) Contamination des fermentations alcooliques industrielles par les levures du genre Brettanomyces. Ind Aliment Agric 106, 559–563. de Souza-Liberal AT, Basílio AC, do Monte Resende A, Brasileiro BT, da Silva-Filho EA, de Morais JO, Simões DA and de Morais MA Jr. Identification of Dekkera bruxellensis as a major contaminant yeast in continuous fuel ethanol fermentation. J Appl Microbiol. 2007;102:538–547. Doolittle WF and Papke RT (2006) Genomic and the bacterial species problem. Genome Biol 7, 116. Drancourt, M., Roux, V., Fournier, P.E., Raoult, D. (2004) rpoB gene sequence-based identification of aerobic Gram-positive cocci of the genera Streptococcus, Enterococcus, Gemella, Abiotrophia and GranulicatellaI. J Cli Microbiol 42, 497-504. Du Plessis, H. W., Dicks, L. M. T., Pretorius, I. S., Lambrechts, M. G., du Toit, M. (2004) Identification of lactic acid bacteria isolated from South African brandy base wines. Int J Food Microbiol 91: 19-29. Dubernet, S., Desmasures, N., Guéguem, M., (2002) A PCR-based method for identification of lactobacilli at the genus level. FEMES Microbiol Lett 214: 271-275. Duffner F and O’Connell M (1995) Comparative evaluation of plasmid profile and ribotyping in the analysis of Lactobacillus plantarum strain heterogeneity in silage. J Appl Bacteriol 78: 20-27. Ferrero M, Cesena C, Morelli L, Scolari G and Vescovo M (1996) Molecular characterization of Lactobacillus casei strains. FEMS Microbiol Lett 140:215–219. 64 Garcia CE (2000) Efeito do nível de contaminação de bactérias isoladas de processo industrial de fermentação alcoólica, na floculação de levedura. Univ. de São Paulo/Escola Sup. de Agricultura Luiz de Queiroz - Ciência e Tecnologia de Alimentos. 80p. Dissertação (Mestrado). Gallo CD (1989) Contaminantes bacterianos em mosto e dornas de fermentação alcoólica. In: Eguchi SY, Yokoya F, Canhos VF and Gallo CR Pontos Criticos Microbiologicos em Usinas de Açúcar e Álcool. Campinas: Fundação Tropical de Pesquisas André Tosello, , 18p. Gallo CR (1992) Identificação de bactérias contaminantes da fermentação alcoólica. STAB - Açúcar Alcool e Subprodutos, Piracicaba, Fermentec v. 10, n. 5, p. 30-34. Gevers D, Huys G and Swings J (2001) Applicability of rep-PCR fingerprinting for identification of Lactobacillus species. FEMS Microbiol Lett. 205(1):31-6. Gevers D, Cohan FM, Lawrence JG, Spratt BG, Coenye T, Feil EJ, Stackebrandt E, Van de Peer Y, Vandamme P, Thompson FL and Swings J. (2005) Opinion: Re-evaluating prokaryotic species. Nat Rev Microbiol 3:733-739. Grimont F and Grimont PDA (1986) Ribosomal ribonucleic acid gene restriction as potential taxonomic tools. Ann Inst Pasteur Microbiol 137B:165–175. Gürtner V and Stanish A (1996) New approaches to typing and identification of bacteria using the 16S-23S rDNA spacer region. Microbiol 142: 3-6. Harvey S, Hill CW, Squires C, and Squerea CL (1988) Loss of the spacer loop sequence from the rrnB operon in the Escherichia coli K12 subline that bears the relA1 mutation. J Bacteriol 170: 1235-1238. Hauben L, Vauterin L, Swings J, Moore ER (1997) Comparison of 16S Ribosomal Sequences of All Xantomonas Species. Int J Syst Bacteriol. Vol. 47 No. 2. p. 328-335. Holzapfel WH, Haberes P, Guisen R, Björkroth J and Schillinger U (2001) Taxomomy and important features of probiotic microorganism in food and nutrition. Am J Clin Nutr 73: 365-373. 65 Hongoh Y, Yuzawa H, Ohkuma M and Kudo T (2003) Evaluation of primers and PCR condition for the analysis of 16S rRNA genes from a natural environment. FEMS Microbiol Lett Oxford, v.221, p.299-304. Hyytiä-Trees E, Lyhs U, Korkeala H and Bjorkroth J (1999) Characterisation of ropy slimeproducing Lactobacillus sakei using repetitive element sequence-based PCR. Int J Food Microbiol. 50, 215-219. Kim MH and Kyung KH (2003) Leuconostoc garlicum sp. nov. isolated from garlic (NCBI http://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=255248) (Não publicado) Kimura K, McCartney AL, McConnell MA and Tannock GW (1997) Analysis of fecal populations of bifidobacteria and lactobacilli, and investigation of the immunological responses of their human hosts to the predominant strains. Appl Environ Microbiol 63: 3394– 3398. Klein G, Pack A, Bonaparte C and Reuter G. (1998) Taxonomy and physiology of probiotic lactic acid bacteria. Int J Food Microbiol 41: 103-125. Kosaric N, Weiczorek A, Cosentino GP, Magee RJ and Prenosil JE Ethanol fermentation. In: REHN HJ and Reed G Eds. Biotechnol - A Comprehensive Treatise in 8 volumes. Weinhein, Verlag Chemie, 1983. V.3, p.257-385. Konstantinidis KT, Ramette A and Tiedje JM (2006) The bacterial species definition in the genomic era. Philos Trans R Soc Lond B Biol Sci 361, 1929-1940. Lopes ACS, Rodrigues JF, Clementino MBM, Miranda CAC, Nascimento APA and Morais MA Jr (2007) Application of PCR ribotyping and tDNA-PCR for Klebsiella pneumoniae identification. Mem Inst Oswaldo Cruz 102: 827-832. Lucena BTL (2010) Diversidade de Bactérias Lácticas Presentes em Destilarias de Álcool Combustível. Univ. Fed. Pernambuco - UFPE. 22p. Tese (Doutorado). Lucena BT, dos Santos BM, Moreira JL, Moreira AP, Nunes AC, Azevedo V, Miyoshi A, Thompson FL, de Morais MA Jr. (2010) BMC Microbiol nov 23;10:298. 66 Ludwig KM, Oliva-Neto P, and Angelis DF (1997) Qualificação da floculação de Sacaromyces cerevisiae por bactérias contaminantes da fermentação alcoólica. Ciênc Tecnol Aliment Campinas, v.21, n.1, p. 63-68. Ludwig KM, Oliva-Neto P and Angelis DF (2001) Quantification of Saccharomyces cerevisiae flocculation by contaminant bacteria from alcoholic fermentation. Ciênc Tecnol Aliment, v21, p.63-68. Makanjuola DB, Tymon A and Springham DG (1992) Some Effects of lactic acid bacteria on laboratory-scale yeast fermentation. Enz Microbiol Technol v.14, n,4, p,350-357. Miteva V, Boudakov I, Ivanova-Stoyancheva G, Marinova B, Mitev V and Mengaud J (2001) Differentiation of Lactobacillus deubrueckii subspecies by ribotyping and amplified ribosomal DNA restriction analysis (ARDRA). J Appl Microbiol 90: 909-918. Mohania D, Nagpal R, Kumar M, Bhardwaj A, Yadav M, Jain S, Marotta F, Singh V, Parkash O and Yadav H. (2008) Molecular approaches for identification and characterization of lactic acid bacteria. J Dig Dis. nov;9(4):190-8. Morea M, Barizzi F, Cappa F and Cocconcelli PS (1998) Molecular characterization of Lactobacillus community in tradicional processing of mozzarella cheese. Int J Food Microbiol 43: 53-60. Moreira JLS, Mota RM, Horta MF, Teixeira SMR, Neumann E, Nicoli JR and Nunes AC (2005) Identification to the species level of Lactobacillus isolated in probiotic prospecting studies of human, animal or food origin by 16S-23S rRNA restriction profiling. BMC Microbiol 5: 15-23. Narendranath N V, Hynes S H, Thomas K C e Ingledew W M (1997) Effects of Lactobacilli on yeast-catalyzed ethanol fermentations. Applied and Environmental Microbiology 63: 4158-4163. Narendranath NV, Thomas KC and Ingledew WM (2000) Urea Hydrogen Peroxide Reduces the Numbers of Lactobacilli, Nourishes Yeast, and Leaves No Residues in the Ethanol Fermentation. Appl Environ Microbiol 66(10): 4187-4192. 67 Naser S, Thompson FL, Hoste B, Gevers D, Vandemeulebroecke K, Cleenwerck I, Thompson CC, Vancanneyt M and Swings J (2005a) Phylogeny and identification of enterococci using atpA gene sequence analysis. J Clin Microbiol 43, 2224-2230. Naser S, Thompson FL, Hoste B, Gevers D, Dawyndt P, Vancanneyt M and Swings J (2005b) Aplication of multilocus sequence analysis (MLSA) for rapid identification of Enterococcus species based on rpoA and pheS genes. Microbiol 151, 2141-2150. Naser S, Dawyndt P, Hoste B, Gevers D, Vandemeule-broecke K, Cleenwerck I, Vancanneyt M and Swings J (2007) Identification of lactobacilli by pheS and rpoA gene sequence analyses. Int J Syst Evol Microbiol 57: 2777-2789. Niederhauser C, Höfelein C, Allmann M, Burkhalter P, Lüthy J and Candrian U (1994) Random amplification of polymorphic bacterial DNA: evaluation of 11 oligonucleotides and application to food contaminated with Listeria monocytogenes. J Appl Bacteriol 77: 574-582. Nour M (1998) 16S-23S and 23S-5S intergenic spacer regions of lactobacilli: nucleotide sequence, secondary structure and comparative analysis. Res Microbiol 149: 433-448. Oliva-Neto P, Yokoya F (1994) Evaluation of bacterial contamination in a fed-batch alcoholic fermentation process. World J Microbiol Biotechnol v. 10, n.6, p.697-699. Oliva-Neto P and Yokota F (1997) Effects of nutritional factors on growth of Lactobacillus fermentum mixed with Sacharomyces cerevisiae in alcoholic fermentation. Rev. Microbiol. v.28, p.25-31. Passoth V, Blomqvist J and Schnürer J (2007) Dekkera bruxellensis and Lactobacillus vini Form a Stable Ethanol-Producing Consortium in a Commercial Alcohol Production Process. Appl Environ Microbiol 73: 4354–4356. Pavlova SI, Kilic AO, So JS, Nader-Macias ME, Simões JA and Tao L (2002) Genetic diversity of vaginal lactobacilli from women in different countries based on 16S rRNA gene sequences. J Appl Microbiol 92: 451-459. 68 Pisabarro A, Correia A and Martín J, (1998) Characterization of the rrnB operon of the plant pathogen Rhodococcus fascians and targeted integrations of exogenous genes at rrn loci. Appl Environ Microbiol 64, 1276-1282. Pot B, Hertel C, Ludwig W, Descheemaker P, Kersters K and Schleifer KH (1993) Identification and classification of Lactobacillus acidophilus, L. gasseri and L. johnsonii strains by SDS PAGE and rRNA-targeted oligonucleotide probe hybridization. J Gen Microbiol 139:513–7. Rondini MAT (1985) Isolamento, Caracterização e Identificação de Bactérias Contaminantes de Dornas de Fermentação nas Destilarias de Etanol. Piracicaba. Univ. São Paulo - Escola Superior de Agricultura Luiz de Queiroz. p.37 Tese (Mestrado). Roth A, Fischer M, Hamid ME, Michalke S, Ludwig W and Mauch H (1998) differentiation of Phylogenetically related slowly growing mycobacteria based on 16S-23S rRNA gene internal transcribed spacer sequences. J Clin Microbiol v.36, p.139-147. Roussel Y, Colmin C, Simonet JM and Decaris B (1993) Strain characterization, genome size and plasmid content in the Lactobacillus acidophilus group (Hansen and Mocquot). J Appl Bacteriol 74:549–56. Roy D, Ward P and Champagne G (1996) Differentiation of bifidobacteria by use of pulsed-field gel electrophoresis and polymerase chain reaction. Int J Food Microbiol 29:11–29. Ruiz A, Poblet M, Mas A and Guillamón JM (2000). Identification of acetic acid bacteria by RFLP PF PCR-amplifield 16S rDNA intergenic spacer. Int J Syst Evol Microbiol. 50, 1981-1987 Schell DJ, Dowe N, Ibsen KN, Riley CJ, Ruth MF and Lumpkin RE (2007) Contaminant occurrence, identification and control in a pilot-scale corn fiber to ethanol conversion process. Bioresour Technol 98: 2942-2948. 69 Schleifer KH and Ludwig W (1995) Phylogenetics relationships of acid lactic bacteria. In The Genera of Lactic Acid Bacteria ed. Wood, B.J.B. and Holzapfel, W.H. vol. 2, p. 718. Blackie Academic Professional. Schwan RF, Mendonça AT, Silva-Jr JJ, Rodrigues V and Wheals AE (2001) Microbiology and physiology of Cachaça (Aguardente) fermentation. Antonie van Leeuwenhoek, Dordrecht, v.79, p.89-96, 2001. Seseña S, Sánchez I and Palop L (2004) Genetic diversity (RAPD-PCR) of lactobacilli isolated from “Almagro” eggplant fermantations from two seasons. FEMS Microbiol Lett 238: 159-165. Shang S, Fu J, Dong G, Hong W, Du L and Yu X (2003) Estabilishment and analysis of specifc DNA patterns in 16S-23S rRNA gene spacer regions for differentiating different bacteria. Chin Med J 116 (1), 129-133. Silva N (1998) Influência do resfriamento em torres sobre a microflora do caldo de cana no processo de produção de álcool. Campinas. Univ. Campinas - Faculdade de Engenharia de Alimentos. p.117. Dissertação (Mestrado). Silva-Filho EA (2003) Caracterização genética de populações de leveduras em destilarias de álcool combustível com vistas à seleção de linhagens promissoras para a expressão heteróloga de genes de interesse industrial. Recife. Univ. Fed. Pernambuco. Tese (Doutorado) p.50. Silvers M, Uermosi C, Fehlmann M and Krieger S (2003) Cloning, sequence analysis and expression of the FaF0-ATPase beta-subunit from wine lactic acid bactéria. Sys Appl Microbiol 26, 350-356. Simpson KL, Pettersson B and Priest FG (2001) Characterization of lactobacilli from Scotch malt whisky distilleries and description of new Lactobacillus ferintoshensis sp. Nov., a new species isolated from malt whisky fermentation. Microbiology 147: 10071016. Skinner KA and Leathers TD (2004) Bacterial Contaminants of fuel ethanol production. J Ind Microbiol Biotechnol 31: 401-408. 70 Sohier D, Coulon J and Lonvaud-Funel A (1999) Molecular identification of Lactobacillus hilgardii and genetic relatedness with Lactobacillus brevis. Int J Syst Bacteriol 49, 1075-1081. Spacov ICG, Silva SAM, Morais Jr MA, Morais MMC (2006) Polymorphism of the rDNA and tDNA loci in clinical isolates of Pseudomonas aeruginosa: a perspective for molecular epidemiology surveillance. Gen Mol Biol 29: 722-729. Stackebrandt E, Frederiksen W, Garrity GM, Grimont PA, Kämpfer P, Maiden MCJ, Nesme X, Rosselló R, Swings J, Trüper HG, Vauterin L, Ward AC and Whitman WB (2002). Report of the ad hoc committee for re-evaluation of the species definition in bacteriology. Int J Syst Evol Microbiol 52, 1043-1047. Stahl M and Molin G (1994) Classification of Lactobacillus reuteri by restriction endonuclease analysis of chromosomal DNA. Int J Syst Bacteriol. 44: 9–14. Staden R, Beal KF and Bonfield JK (1998) The Staden Package. Comp Meth Mol Biol, 132, 115-130. Tamura K, Dudley J, Nei M and Kumar S (2007) Mega4: Molecular Evolutionary Genetics Analysis (MEGA) Software version 4.0. Mol Biol Evol 24: 1596-1599. Thomas KC, Hynes SH and Ingledew WM (2001) Effect of lactobacilli on yeast growth, viability and batch and semi continuos alcoholic fermentation of corn mash. J Appl Microbiol, v.90, p.819-828. Tilsala-Timisjarvi A and Alatossava T (1997) Development of oligonucleotide primers from the 16S-23S rRNA intergenic sequences for identifying different dairy and probiotic lactic acid bacteria by PCR. Int J Food Microbiol 35:49-56. Towner KJ and Cockayne A (1993) Molecular methods for microbial identification and typing. Chapman & Hall, London. Using JB, Russellomora RA, Garciavaldes E and Lalucat J (1995) Taxonomic note - a pragmatic approuch to the nomenclature of phenotypically similar genomic groups. Int J Syst Bacteriol 45, 604. 71 Valcheva R, Korakli M, Onno B, Prévost H, Ivanova I, Ehrmann MA, Dousset X, Gänzle MG and Vogel RF (2005) Lactobacillus hammesii sp. nov., isolated from French sourdough. Int J Syst Evol Microbiol. mar v. 55, n.2, p. 763-767. van Belkum A (1994) DNA fingerprinting of medically important microorganisms by use of PCR. Clin. Microbiol. Rev. 7, 174-184. van Reenen CA and Dicks LMT (1996) Evaluation of numerical analysis of random amplified polymorphic DNA (RAPD)-PCR as a method to differentiate Lactobacillus plantarum and Lactobacillus pentosus. Curr Microbiol 32:183-187. Ventura M and Zink R (2002) Specific identification and molecular typing analysis of Lactobacillus johnsonii by using PCR – based methods and pulsed-field gel electrophoresis. FEMS Microbilol Lett 217: 141-154. Vos P, Hogers R, Bleeker M, Reijans M, van de Lee T, Hornes M, Frijters A, Pot J, Peleman J and Kuiper M (1995) AFLP: a new technique for DNA fingerprinting. Nucleic Acids Res. nov 23, 4407-4414. Vuyst L, Camu N, De Winter T, Vandemeulebroecke K, Van de Perre V, Vancanneyt M, De Vos P and Cleenwerck I. (2008) Validation of the (GTG)(5)-rep-PCR fingerprinting technique for rapid classification and identification of acetic acid bacteria, with a focus on isolates from Ghanaian fermented cocoa beans. Int J Food Microbiol. jun 30;125(1):79-90. Wheals EA, Basso LC, Alves DMG and Amorim HV (1999) Fuel ethanol after 25 years. Trends Biotechnol 17: 482-487. Welch DF (1991) Application of cellular fatty acid analysis. Clin. Microbiol. Rev. 4, 422438. Yokoya F and Oliva-Neto P (1991) Características da floculação de leveduras por Lactobacillus fermentum. Rev. Microbiol 22(1), 12-16. 72 9. Anexos 9.1 Quadro comparativo das bactérias do gênero Lactobacillus 9.2 Lista de isolados identificados a partir do caldo de cana das destilarias estudadas neste trabalho 73 Anexo 1 Quadro comparativo das bactérias do gênero Lactobacillus SphI + + + + + + + + + + + + + + + + + + - NcoI + + + + + + + + + + + + + + + NheI + + + - SspI + + + + + + - SfuI + + + + + + + + EcoRV + + + + + + + + + - DraI + + + + + + + + + + + + - VspI + + + + + + + + + + + + + + + + + + - HincII + + + - EcoRI + + + + + + + + + - HindIII + + + + + + + + + + + + + + + + + + + + + + + + - AvrI + + + + + + + + + - Identificação L. acidophilus L. agilis L. alimentarius L. amylovorus L. animalis L. brevis L. casei L. camelliae L. colehominis L. crispatus L. delbrueckii 75 SphI + + + + + + + + + + + + + + + + + + - NcoI + + + + - NheI - SspI + + + + + + + + + + + + - SfuI - EcoRV + + + + + + + + + - DraI + + + + + + + + + VspI + + + + + + + + + + + + + + + + + + + + + - HincII + + + + + + + - EcoRI - HindIII + + + + + + + + + + + + + + + + + AvrI + + + - IDENTIFICAÇÃO L. "diolivorans like" L. farciminis L. ferintoshensis L. fermentum L. frumenti L. fructivorans L. gasseri L. hilgardii L. jensenii L. johnsonii L. murinus 76 SphI + + + + + + + + + + + + + + + + + + + + + + + + + + + + + + NcoI + + + + + + + + + + NheI + + + - SspI + + + + + + + + + - SfuI + + + +/- EcoRV + + + + + + - DraI + + + + + + + + + - VspI + + + + + + + + + + + + + + + + + + + + + + + + HincII + + + + + + + + + +/- EcoRI + + + - HindIII + + + + + + + + + - AvrI - IDENTIFICAÇÃO L. mucosae L. nageli L. panis L. pantheris L. paralimentarius L. paraplantarum L. pentosus L. perolens L. plantarum L. pontis L. reuteri - 77 SphI + + 0 + + + + + + + + + NcoI 0 + + + - NheI 0 + + + - SspI 0 + + + + + + - SfuI 0 + + + - EcoRV + + + 0 - DraI + + + + + 0 + + + + + + + + + + + + +/+/+/- VspI 0 + + + + + + - HincII + + 0 + - EcoRI 0 + + + HindIII + + + + + 0 + + + + - AvrI 0 - IDENTIFICAÇÃO L. rhamnosus L. rossii L. ruminis L. safranciscencis L. sakei L. salivarus L. vaginalis L. vini Modificada a partir de Moreira et al. (2005) e Lucena (2010). 78 Anexo 2 Lista de isolados identificados a partir do caldo de cana das destilarias estudadas neste trabalho Isolados Identificação Local de Coleta Metodologia para Identificação GCP7.1.1 GCP7.1.2 GCP7.1.3 GCP7.1.4 GCP7.3.1 GCP7.3.2 GCP7.3.3 GCP7.3.2 GCP7.3.4 GCP7.3.5 GCP7.3.6 GCP7.3.7 GCP7.3.8 GCP7.3.9 GCP7.3.10 GCP7.3.11 GCP7.3.12 GCP7.3.13 GCP7.3.14 GCP7.3.15 GCP7.3.16 GCP7.3.17 Lactococcus lactis Lactococcus lactis Lactococcus lactis Lactococcus lactis Lactobacillus fermentum Lactobacillus fermentum Lactobacillus fermentum Lactobacillus fermentum Lactobacillus fermentum Lactobacillus fermentum Lactobacillus fermentum Lactobacillus fermentum Lactobacillus fermentum Lactobacillus fermentum Lactobacillus fermentum Lactobacillus fermentum Lactobacillus fermentum Lactobacillus fermentum Lactobacillus fermentum Lactobacillus fermentum Lactobacillus fermentum Lactobacillus fermentum Destilaria Giasa - PB Destilaria Giasa Destilaria Giasa Destilaria Giasa Destilaria Giasa Destilaria Giasa Destilaria Giasa Destilaria Giasa Destilaria Giasa Destilaria Giasa Destilaria Giasa Destilaria Giasa Destilaria Giasa Destilaria Giasa Destilaria Giasa Destilaria Giasa Destilaria Giasa Destilaria Giasa Destilaria Giasa Destilaria Giasa Destilaria Giasa Destilaria Giasa ARDRA ARDRA ARDRA ARDRA ARDRA ARDRA ARDRA ARDRA ARDRA ARDRA ARDRA Sequênciamento Sequênciamento ARDRA ARDRA ARDRA ARDRA ARDRA Sequênciamento Sequênciamento Sequênciamento Sequênciamento 79 GCP7.3.18 GCP7.3.19 GCP7.3.20 JCP7.1.1 JCP7.1.2 JCP7.1.3 JCP7.1.4 JCP7.1.5 JCP7.1.6 JCP7.1.7 JCP7.1.8 JCP7.1.9 JCP7.1.10 JCP7.1.11 JCP7.1.12 JCP7.1.14 JCP7.1.15 JCP7.1.16 JCP7.1.17 JCP7.1.20 JCP7.1.21 JCP7.2.2 JCP7.2.3 JCP7.2.8 JCP7.2.9 JCP7.2.10 JCP7.2.11 JCP7.2.12 Lactobacillus fermentum Lactobacillus fermentum Lactobacillus fermentum Weissella cibaria Leuconostoc garlicum Leuconostoc garlicum Weissella cibaria Weissella cibaria Leuconostoc mesenteroides Weissella cibaria Leuconostoc citreum Leuconostoc garlicum Leuconostoc mesenteroides Leuconostoc mesenteroides Leuconostoc garlicum Staphylococcus epidermidis Leuconostoc mesenteroides Leuconostoc garlicum Leuconostoc citreum Weissella cibaria Weissella confusa Weissella confusa Leuconostoc garlicum Weissella confusa Weissella confusa Weissella confusa Weissella confusa Weissella confusa Destilaria Giasa Destilaria Giasa Destilaria Giasa Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Sequênciamento Sequênciamento ARDRA Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento ARDRA Sequênciamento Sequênciamento Sequênciamento ARDRA ARDRA Sequênciamento Sequênciamento ARDRA Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento 80 JCP7.2.14 JCP7.2.15 JCP7.2.16 JCP7.2.17 JCP7.2.18 JCP7.2.19 JCP7.2.20 JCP7.3.1 JCP7.3.2 JCP7.3.3 JCP7.3.4 JCP7.3.5 JCP7.3.6 JCP7.3.7 JCP7.3.8 JCP7.3.9 JCP7.3.10 JCP7.3.12 JCP7.3.13 JCP7.3.14 JCP7.3.15 JCP7.3.16 JCP7.3.17 JCP7.3.18 JCP7.3.19 JCP7.3.20 MCP7.1.1 MCP7.1.2 Leuconostoc citreum Weissella confusa Leuconostoc citreum Leuconostoc citreum Leuconostoc citreum Leuconostoc citreum Leuconostoc citreum Lactobacillus fermentum Lactobacillus fermentum Leuconostoc mesenteroides Leuconostoc mesenteroides Leuconostoc mesenteroides Leuconostoc mesenteroides Leuconostoc mesenteroides Leuconostoc mesenteroides Leuconostoc mesenteroides Leuconostoc mesenteroides Leuconostoc mesenteroides Leuconostoc mesenteroides Leuconostoc mesenteroides Leuconostoc mesenteroides Weissella cibaria Leuconostoc mesenteroides Leuconostoc mesenteroides Leuconostoc mesenteroides Leuconostoc mesenteroides Weissella cibaria Lactobacillus nagelli Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Japungu - PB Destilaria Miriri - PB Destilaria Miriri - PB Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento ARDRA ARDRA Sequênciamento ARDRA ARDRA ARDRA Sequênciamento ARDRA ARDRA ARDRA ARDRA ARDRA Sequênciamento ARDRA Sequênciamento ARDRA ARDRA ARDRA Sequênciamento Sequênciamento ARDRA 81 MCP7.1.3 MCP7.1.4 MCP7.1.5 MCP7.1.6 MCP7.1.7 MCP7.1.9 MCP7.1.10 MCP7.1.11 MCP7.1.12 MCP7.1.13 MCP7.1.14 MCP7.1.17 MCP7.1.19 MCP7.1.20 MCP7.2.1 MCP7.2.3 MCP7.2.9 MCP7.2.10 MCP7.2.15 MCP7.2.16 MCP7.2.17 MCP7.2.19 MCP7.2.20 MCP7.3.1 MCP7.3.2 MCP7.3.3 MCP7.3.4 MCP7.3.5 Lactobacillus nagelli Weissella confusa Leuconostoc mesenteroides Leuconostoc pseudomesenteroides Weissella confusa Leuconostoc pseudomesenteroides Lactobacillus salivarus Leuconostoc lactis Lactobacillus salivarus Leuconostoc pseudomesenteroides Leuconostoc pseudomesenteroides Leuconostoc pseudomesenteroides Weissella confusa Weissella confusa Lactobacillus fermentum Lactobacillus fermentum Lactobacillus fermentum Lactobacillus fermentum Lactobacillus fermentum Lactobacillus fermentum Lactobacillus fermentum Lactobacillus fermentum Lactobacillus fermentum Weissella confusa Weissella confusa Weissella confusa Weissella confusa Weissella confusa Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB ARDRA Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento ARDRA Sequênciamento ARDRA Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento ARDRA ARDRA ARDRA ARDRA ARDRA ARDRA ARDRA ARDRA ARDRA Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento 82 MCP7.3.6 MCP7.3.7 MCP7.3.8 MCP7.3.9 MCP7.3.10 MCP7.3.11 MCP7.3.12 MCP7.3.20 TCP7.1.1 TCP7.1.2 TCP7.1.3 TCP7.1.4 TCP7.1.5 TCP7.1.6 TCP7.1.7 TCP7.1.8 TCP7.1.9 TCP7.1.10 TCP7.1.11 TCP7.1.13 TCP7.1.14 TCP7.1.15 TCP7.1.16 TCP7.1.17 TCP7.1.18 TCP7.1.19 TCP7.1.20 Weissella confusa Weissella confusa Weissella confusa Weissella confusa Weissella confusa Weissella confusa Leuconostoc lactis Lactobacillus fermentum Weissella confusa Leuconostoc mesenteroides Leuconostoc mesenteroides Leuconostoc mesenteroides Leuconostoc mesenteroides Lactobacillus plantarum Leuconostoc mesenteroides Leuconostoc mesenteroides Lactobacillus plantarum Lactobacillus plantarum Leuconostoc mesenteroides Leuconostoc mesenteroides Leuconostoc mesenteroides Weissella paramesenteroides Leuconostoc mesenteroides Leuconostoc mesenteroides Lactobacillus plantarum Lactobacillus plantarum Lactobacillus plantarum Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Miriri - PB Destilaria Trapiche - PE Destilaria Trapiche - PE Destilaria Trapiche - PE Destilaria Trapiche - PE Destilaria Trapiche - PE Destilaria Trapiche - PE Destilaria Trapiche - PE Destilaria Trapiche - PE Destilaria Trapiche - PE Destilaria Trapiche - PE Destilaria Trapiche - PE Destilaria Trapiche - PE Destilaria Trapiche - PE Destilaria Trapiche - PE Destilaria Trapiche - PE Destilaria Trapiche - PE Destilaria Trapiche - PE Destilaria Trapiche - PE Destilaria Trapiche - PE Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento ARDRA Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento Sequênciamento ARDRA ARDRA ARDRA 83 10. Curriculum Lattes Billy Manoel dos Santos Curriculum Vitae Outubro/2012 84 Billy Manoel dos Santos Curriculum Vitae ______________________________________________________________________________________ Dados pessoais Nome Billy Manoel dos Santos Filiação Valdemiro Manoel dos Santos e Irenice Maria dos Santos Nascimento 11/05/1982 - Recife/PE - Brasil Carteira de Identidade 5193768 ssp - PE - 08/04/1999 CPF 037.521.584-09 ______________________________________________________________________________________ Formação acadêmica/titulação 2010 - 2012 Mestrado em Genética. Universidade Federal de Pernambuco, UFPE, Recife, Brasil Título: Identificação Molecular de bactérias Lácticas Presentes no Caldo de Cana-deaçúcar, Ano de obtenção: 2012 Orientador: Marcos Antônio de Morais Júnior Co-orientador: Brígida Thays Luckwu de Lucena 2004 - 2008 Graduação em Ciências Biólógicas / bach.. Universidade Federal de Pernambuco, UFPE, Recife, Brasil Título: IDENTIFICAÇÃO DE BACTÉRIAS LÁCTICAS ISOLADAS DA FERMENTAÇÃO ALCOÓLICA INDUSTRIAL PELA TÉCNICA DE ARDRA-PCR Orientador: Marcos Antônio de Morais Júnior ______________________________________________________________________________________ Formação complementar 2012 - 2012 Curso de curta duração em Uso Progressivo da força e Tecnologias Não Letais. Academia Integrada de Defesa Social, ACIDES, Brasil 2012 - 2012 Curso de curta duração em Uso Diferenciado da Força. Secretaria Nacional de Segurança Pública, SENASP, Brasil 2012 - 2012 Curso de curta duração em Enfrent. da Expl. Sex. de Crianças e Adolescentes. Secretaria Nacional de Segurança Pública, SENASP, Brasil 2012 - 2012 Curso de curta duração em Inglês 1. Secretaria Nacional de Segurança Pública, SENASP, Brasil 2012 - 2012 Curso de curta duração em Formação de Formadores. Secretaria Nacional de Segurança Pública, SENASP, Brasil 2011 - 2011 Curso de curta duração em Aspectos Jurídicos da Abordagem Policial. Secretaria Nacional de Segurança Pública, SENASP, Brasil 2010 - 2010 Curso de curta duração em Aconselhamento Genético: aspectos práticos. Sociedade Brasileira de Genética, SBG, Ribeirao Preto, Brasil 2010 - 2010 Curso de curta duração em Violência, Criminalidade e Prevenção. Secretaria Nacional de Segurança Pública, SENASP, Brasil 2010 - 2010 Curso de curta duração em Genética Forense. Sociedade Brasileira de Genética, SBG, Ribeirao Preto, Brasil 85 Inglês V. Instituto de Apoio a Fundação Universidade de Pernambuco, IAUPE, Recife, Brasil Ano de interrupção: 2010 2010 - 2010 Curso de curta duração em Polícia Comunitária. Secretaria Nacional de Segurança Pública, SENASP, Brasil 2009 - 2009 Curso de curta duração em Investigação criminal 1. Secretaria Nacional de Segurança Pública, SENASP, Brasil Bolsista do(a): Secretaria Nacional de Segurança Pública 2009 - 2009 Curso de curta duração em Mulher Vítima de Violência Doméstica. Secretaria Nacional de Segurança Pública, SENASP, Brasil Bolsista do(a): Secretaria Nacional de Segurança Pública 2009 - 2009 Ingês III. Instituto de Apoio a Fundação Universidade de Pernambuco, IAUPE, Recife, Brasil 2009 - 2009 Inglês IV. Instituto de Apoio a Fundação Universidade de Pernambuco, IAUPE, Recife, Brasil 2009 - 2009 Curso de curta duração em Mediação de Conflitos 1. Secretaria Nacional de Segurança Pública, SENASP, Brasil Bolsista do(a): Secretaria Nacional de Segurança Pública 2008 - 2008 Curso de curta duração em Gerenciamento de Crises. Secretaria Nacional de Segurança Pública, SENASP, Brasil Bolsista do(a): Secretaria Nacional de Segurança Pública 2008 - 2008 Curso de curta duração em Utilização de SNPs em Genética Forense. Sociedade Brasileira de Genética, SBG, Ribeirao Preto, Brasil 2008 - 2008 Inglês II. Instituto de Apoio a Fundação Universidade de Pernambuco, IAUPE, Recife, Brasil 2008 - 2008 Inglês I. Instituto de Apoio a Fundação Universidade de Pernambuco, IAUPE, Recife, Brasil 2008 - 2008 Curso de curta duração em Técnicas e Tecnol. Não Letais na Atuação Policial. Secretaria Nacional de Segurança Pública, SENASP, Brasil Bolsista do(a): Secretaria Nacional de Segurança Pública 2008 - 2008 Curso de curta duração em Uso Progressivo da Força. Secretaria Nacional de Segurança Pública, SENASP, Brasil Bolsista do(a): Secretaria Nacional de Segurança Pública 2005 - 2005 Curso de curta duração em Proc de Mat Biológico p/ Incl e cortes em Parafin. Universidade Federal de Pernambuco, UFPE, Recife, Brasil ______________________________________________________________________________________ Atuação profissional 1. Universidade Federal de Pernambuco - UFPE ____________________________________________________________________________ Vínculo institucional 86 2010 - Atual 2007 - 2008 Vínculo: Aluno de Mestrado , Enquadramento funcional: Pesquisador , Carga horária: 36, Regime: Parcial Vínculo: Aluno de Iniciação Científica , Enquadramento funcional: Pesquisador , Carga horária: 20, Regime: Parcial ____________________________________________________________________________ Atividades 04/2007 - Atual Pesquisa e Desenvolvimento, Centro de Ciências Biológicas Linhas de pesquisa: Identificação Molecular de Bactérias Lácticas 04/2007 - 12/2008 Estágio, Centro de Ciências Biológicas, Departamento de Genética Estágio: Biologia Molecular 2. Polícia Militar de Pernambuco - PMPE ____________________________________________________________________________ Vínculo institucional 2006 - Atual Vínculo: Servidor público , Enquadramento funcional: Policial Militar , Carga horária: 48, Regime: Integral ______________________________________________________________________________________ Linhas de pesquisa 1. Identificação Molecular de Bactérias Lácticas ______________________________________________________________________________________ Áreas de atuação 1. Genética Molecular e de Microorganismos Producão ______________________________________________________________________________________ Produção bibliográfica Artigos completos publicados em periódicos 1. SOUZA, RAFAEL BARROS, Santos, Billy Manoel dos, FÁTIMA RODRIGUES DE SOUZA, RAQUEL, SILVA, PAULA KATHARINA NOGUEIRA DA, LUCENA, BRÍGIDA THAIS LUCKWU, MORAIS, MARCOS ANTONIO The consequences of Lactobacillus vini and Dekkera bruxellensis as contaminants of the sugarcane-based ethanol fermentation. Journal of Industrial Microbiology and Biotechnology. , v.39, p.1 - 8, 2012. 2. LUCENA, BRIGIDA TL, DOS SANTOS, Billy M, MOREIRA, JOÃO LS, MOREIRA, ANA PAULA B, NUNES, ALVARO C, AZEVEDO, VASCO, MIYOSHI, ANDERSON, THOMPSON, FABIANO L, DE MORAIS, MARCOS Diversity of lactic acid bacteria of the bioethanol process. BMC Microbiology (Online). , v.10, p.298 - , 2010. 87
Baixar