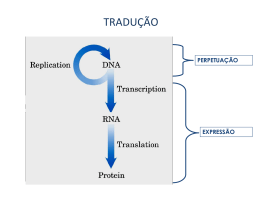
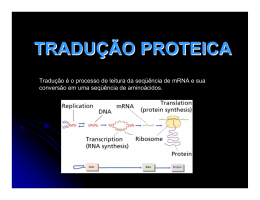
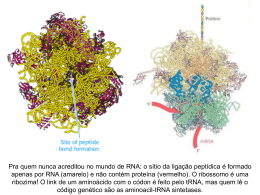
UNIDADE 03 METABOLISMO DE PROTEÍNA Disciplina de Biociências I DB-110 Área de Bioquímica Profa. Cínthia P. M. Tabchoury O QUE É QUE VOCÊS SE LEMBRAM DE PROTEÍNA?? Biossíntese de Proteínas I – Introdução: 70 proteínas ribossômicas, 20 ou + enzimas ativadoras de aa, + 12 enzimas auxiliares, fatores de iniciação, alongação e terminação, + 40 tRNA = cerca de 300 moléculas envolvidas. II – Síntese Proteica: a) descoberta; b) Características; c) Ativação dos aa; d) Iniciação; e) Alongamento; f) Terminação; g) Enrolamento e Processamento. Introdução As proteínas devem ser sintetizadas em resposta às necessidades da célula, transportadas e degradadas quando a necessidade cessar; A síntese das proteínas é o mais complexo dos mecanismos biossintetizantes; Pode ser responsável pelo gasto de até 90% da energia química usada por 1 célula para todas as reações biossintetizantes; Apesar da complexidade, as proteínas são sintetizadas em velocidades extrema/e altas. Ex: cadeia polipeptídica com 100 resíduos de aa – 5 segundos Sítio da síntese protéica é o ribossomo citosol Lúmen do RE ribossomo Ribossomo: são compostos de rRNA e proteínas. Possuem 2 subunidades que se encaixam de modo que uma fenda é formada entre elas por onde passa o mRNA durante o processo de síntese protéica. aminoácido Sítio de ligação do aa adaptador Trinca de nucleotídeos codificando para uma aa Os aa estão ativados: são ligados ao tRNA, formando os aminoacil-tRNAs. Códon de iniciação, AUG, sinaliza o início das cadeias polipeptídicas. 3 trincas nucleotídicas, UAA, UAG, UGA, não codificam qualquer aminoácido e sinalizam o fim da síntese da cadeia polipeptídica. segunda letra do códon Primeira letra do códon (extremidade 5’) O código genético é degenerado, significando que um certo aminoácido pode ser especificado por mais de um códon; Degenerado não significa imperfeito; O código genético não é ambíguo, pois nenhum códon especifica mais de um aa; Quando um aa possui códons múltiplos, a diferença entre os códons está, geralmente, na 3a base. Ex: alanina é codificado pelas trincas GCU, GCC, GCA e GCG tRNA funciona como um adaptador, reconhece uma seqüência nucleotídica curta no mRNA. Uma trinca nucleotídica específica no tRNA interage com uma trinca códon específica no mRNA através de pontes de H de bases complementares. Os 2 RNAs são pareados antiparalelamente, a primeira base do códon (ler na direção 5’3’) pareando com a terceira base do anticódon. A síntese proteica é composta por 5 etapas: ativação dos aa, iniciação, alongamento, terminação/liberação e enrolamento/processamento. Os tRNAs são relativamente pequenos e consistem de 1 fita simples de RNA enrolada; Há pelo menos 1 espécie de tRNA para cada aa; Todos tRNAs possuem a seqüência extremidade 3’ onde se liga ao aminoácido. CCA na Há ativação do aminoácido para formação da ligação peptídica e a ligação do aa a um tRNA adaptador que direciona sua colocação. aminoácido Classe I aminoacil-tRNA sintetases Classe II aminoacil-tRNA sintetases A identidade do aminoácido ligado ao tRNA não é checada no ribossomo; A ligação do aminoácido correto é essencial para a fidelidade da síntese de proteínas; A enzima aminoacil-tRNA discrimina e liga-se ao seu específico; sintetase aminoácido Esta enzima ainda possui uma função de revisão, onde os aminoácidos são checados em um segundo centro ativo e os incorretos são hidrolisados. INICIAÇÃO –ESTÁGIO 2 Um aminoácido específico inicia a síntese protéica. A síntese das proteínas começa na extremidade aminoterminal e são alongadas pela adição seqüencial de resíduos de aa até a extremidade carboxilaterminal. O complexo de iniciação se forma em 3 etapas com o gasto da hidrólise de GTP a GDP e Pi. Subunidade 30S Códon de iniciação Subunidade 50S P designa o sítio peptidil. A designa o sítio aminoacil. Subunidade 50S próximo códon ALONGAMENTO – ESTÁGIO 3 Ligações peptídicas formadas nesta etapa; Complexo de iniciação (70S) são próximo códon próximo aminoaciltRNA complexo de iniciação; próximo aminoacil-tRNA especificado pelo códon seguinte no mRNA; fatores de alongamento; o 2º aminoacil se liga no sítio A, o que é acompanhado pela hidrólise de GTP a GDP e Pi. Ligação do próximo aminoaciltRNA ALONGAMENTO fMet-tRNAfMet AminoaciltRNA2 Formação da ligação peptídica; Peptidil transferase: ribozima; dipeptidil-tRNA no sítio A e o tRNA(met) descarregado ligado ao sítio P. Formação da ligação peptídica tRNAfMet deacilado Dipeptidil tRNA2 ALONGAMENTO tRNAfMet deacilado Translocação: ribossomo movese 1 códon na direção da extremidade 3’; Dipeptidil tRNA2 O 3º códon do mRNA agora está no sítio A e o 2º códon no sítio P; Movimento do ribossomo ao longo do mRNA requer a energia fornecida pela hidrólise de outra molécula de GTP. Translocação próximo aminoacil-tRNA Cadeia polipeptídica sempre permanece ligada ao tRNA do último aa que foi inserido; Direção do movimento do ribossomo TERMINAÇÃO – ESTÁGIO 4 Término polipeptídio especial; Fator de liberação se liga da síntese do requer um sinal sinalizada por um dos 3 códons de terminação no mRNA; fatores de terminação; liberação União polipeptidiltRNA hidrolisada ou hidrólise da ligação peptidiltRNA terminal; liberação do polipeptídio livre e do último tRNA; dissociação do ribossomo 70S nas 2 subunidades. Dissociação dos componentes Subunidades ribossomais que chegam POLISSOMOS Agregados de 10 a 100 ribossomos; vários ribossomos podem traduzir um único mRNA, permitindo um uso altamente eficiente do mesmo. Cadeia polipeptídica em crescimento Direção da tradução Nas bactérias, há um acoplamento muito estreito entre a transcrição e a tradução; inicia-se a tradução antes que a transcrição se complete. Enrolamento e Processamento Modificações nos carboxilaterminal; grupos amino Perda da seqüência sinalizadora; Modificações de aminoácidos individuais; Ligação de cadeias laterais de carboidratos; Adição de grupo isoprenil; Adição de grupos prostéticos; Formação das ligações cruzadas de dissulfeto. e Síntese proteica é uma função central na fisiologia celular e o alvo primário de antibióticos e toxinas. Sítio P peptidil-tRNA Sítio P puromicina A puromicina inibe a translocação através da formação da peptidilpuromicina. Peptidil transferase A tetraciclina bloqueia o sítio A. A cicloheximida é uma toxina que bloqueia a transferência do peptídio dos ribossomos 80S (ribossomos eucarióticos) mas não do 70S (ribossomos bacterianos). O cloranfenicol bloqueia a transferência do peptídio. A estreptomicina em baixas concentrações leva a erro de leitura e em altas concentrações inibe a iniciação da síntese protéica. http://www.wiley.com/legacy/college/boyer/0470003790/animations/animations.htm
Download