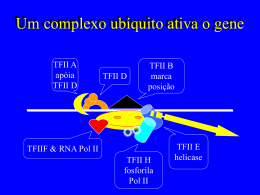
Controle da expressão gênica Como são usados os elementos na seqüência de DNA? Em que condições o produto de cada gene é feito? Quando feito, o que ele faz? O Genoma de quase todas as células do organismo é igual Diferenciação celular depende de mecanismos de memória celular Uma célula humana típica expressa cerca de 10.000 genes de seus aproximadamente 25.000 genes. O padrão de abundância de cada mRNA é muito típico de cada tipo de célula. Para um mesmo tipo de célula, a abundância de cada mRNA varia de acordo com a fase de desenvolvimento, com o estado funcional, com o ciclo circadiano. No DNA, existem seqüências específicas de nucleotídeos que são alvos dos fatores de transcrição: Elementos Regulatórios (“cis-acting elements”) Os fatores de transcrição são denominados “trans-acting elements” – pois são codificados por genes localizados em locais distintos daquele ocupado pelo gene que está sendo regulado. Os fatores reguladores reconhecem sequências de acordo com os grupos químicos expostos nas incisuras maior e menor do DNA dupla fita As diversas combinações das bases expõem grupos químicos de forma distinta, especialmente na incisura maior Fator regulador Elemento cis reconhecido Reconhecem o sítio por interações de fraca intensidade: pontes de H, interações eletrostáticas, interações hidrofóbicas Motivos observados nos domínios de interação com o DNA Helix-turn-helix Homeodomain – 3 hélices alfa Zinc-finger Beta sheet Alças que entram nas incisuras do DNA (p53) Ziper de leucina Frequentemente formam dímeros Reconhecimento de sequências palindrômicas • Os primeiros estudos dos mecanismos de regulação da expressão de genes foram realizados em bactérias • Bactérias: o controle gênico tem como consequência o ajuste metabólico da célula para que essa se adapte a alterações nutricionais do meio. • O controle gênico é feito por fatores de transcrição. Promotor em genes bacterianos Operator: elemento de regulação ao qual se liga o regulador do operon Figure 7-35 Molecular Biology of the Cell (© Garland Science 2008) No operador pode se ligar um regulador helix-turn-helix Figure 7-36 Molecular Biology of the Cell (© Garland Science 2008) Um regulador pode funcionar como ativador em um promotor e como inbidor em outro. Operator mais próximo do promotor em 2 pb Figure 7-38 Molecular Biology of the Cell (© Garland Science 2008) Jacques-Monod Francois Jacob Estudo do Lac Operon Figure 7-39 Molecular Biology of the Cell (© Garland Science 2008) • O controle da transcrição em eucariotos é bastante mais complexa No promotor são integrados todos os inputs regulatórios do gene PROMOTORES DE EUCARIOTOS Lenhard B, Sandelin A, Carninci P Nature Rev 13:233,2012 Tipo I Promotores com TATA-box têm sítio de início de transcrição bem definido. Correspondem a 10-12% dos genes, são genes regulados e expressos em tecidos bem diferenciados Fatores gerais de transcrição e Pol II Fatores específicos de transcrição (em CRM) • Dependem principalmente de módulos regulatórios cis (CRM) para sua regulação • Sítio de início de transcrição coberto por nucleossomos • Distribuição adjacente de nucleossomos sem padrão definido Lenhard B, Sandelin A, Carninci P Nature Rev 13:233,2012 Tipo II Ilhas CpG parecem ser o promotor “default”em mamíferos CpG - início de transcrição em pontos variáveis Promotores com ilhas CpG pequenas Promotores com início de transcrição disperso Sem TATA-box Genes de expressão constitutiva (79-88% dos genes)- housekeeping Promotores ricos em sequências consensuais para Sp1 • Core promoter desprovido de nucleossomos • Região imediatamente à montante, onde também pode iniciar-se a transcrição, desprovida de nucleossomos • Possuem poucos “enhancers” próximos ao core-promotor Lenhard B, Sandelin A, Carninci P Nature Rev 13:233,2012 Uma região livre de nucleossomos (NFR) próxima ao sítio de início de transcrição (TSS) é uma característica comum dos promotores tipo II/III (Transcription Start Site) Venters BJ & Pugh BF. 2009 Tipo III CpG grandes, estendendo-se pelo gene • Regulados durante o desenvolvimento Genes ativos durante o desenvolvimento • Regulados por vários enhancers localizados a distâncias variáveis do TSS (distantes até em megabases) • Região livre de nucleossomos no TSS Lenhard B, Sandelin A, Carninci P Nature Rev 13:233,2012 Lenhard B, Sandelin A, Carninci P Nature Rev 13:233,2012 Genes relacionados ao desenvolvimento, quando inativos estão próximas à lâmina nuclear. Ao serem ativados, movem-se no núcleo. PROMOTORES TCT (com polipiridimidinas) CpG overlapping Promotores de genes de proteínas altamente expressas envolvidas com tradução do mRNA: • proteínas ribossomais • proteínas de início e alongamento de tradução Elementos essenciais na regulação da taxa de transcrição dos genes em eucariotos: Fatores de transcrição específicos para certas sequências Organização dos nucleossomos Remodelagem da cromatina acoplada à montagem dos fatores gerais de transcrição Estados de fosforilação da cauda C-terminal (CTD – C Terminal Domain) da RNA polimerase II (Pol II) associados a modificações na cromatina Fatores de transcrição que se ligam a sequências específicas (enhancers) MEDIATOR TFIID TBP Enhancers: Elementos no DNA que ativam a transcrição independente de direção, localização e distância em relação ao promotor do gene. Regulador sequência específicos Complexos remodeladores de cromatina: Remodelam nucleossomos com gasto de ATP Não possuem especificidade, devem ser recrutados pelos fatores seq-específicos Por força mecânica deslocam o octâmero de histonas ou o remove Deslizamento (sliding) Quatro famílias de remodeladores de cromatina: • SWI/SNF (SWItch Sucrose Non-Fermentable) •ISWI (Imitation SWItch) • CHD (Chromodomain-Helicase DNA-biding protein) • INO80/SWR1 (INOsitol requiring 80/ SWI/SNF Related protein 1) Bromodomínio (lisinas acetiladas) Cromodomínio (lisinas metiladas) REMODELADORES DE CROMATINA • Família SWI/SNF: ativadores de transcrição • Remoção de histonas ou do octâmero inteiro • Importantes para remoção de nucleossomos junto aos promotores • Remodelagem de nucleossomos em genes que são rapidamente ativados • Presentes no promotor e no corpo dos genes ativos • SWI/SNF, RSC, parte dos complexos de acetilação SAGA e p300 ATPase Bromodomínio Bromodomínio: liga-se a lisinas metiladas REMODELADORES DA CROMATINA • ISWI ATPase SANT SLIDE • Implicados no alongamento do transcrito • Observados no corpo dos genes altamente transcritos • Trocam histonas dos nucleossomos • Propicial deslizamento de nucleossomos REMODELADORES DA CROMATINA • CHD • Colocalizam-se com RNA Pol II • Participam nos passos iniciais do alongamento do transcrito • Propiciam o deslizamento de nucleossomos Chromodomínio ATPase Domínio de ligação ao DNA Complexo SAGA tem como um de seus componentes Chd1 CHD1 interage com chaperona de histonas (FACTS – facilitates chromatin transcription) CHD1 interage dom DSIF CHD1 interage com H3K4me3 Associado a transcrição: tem atividade de acetilase de histonas, de remodelador de cromatina, reconhecimento H3K4me3 e deubiquitinação REMODELADORES DE CROMATINA • INO80/SWR1 ATPase • Atuam no alongamento da transcrição realizando troca de histonas SWR1 H2A INO80 H2A.Z (Remodel the Structure of Chromatin) Ativação Substituição das histonas H2A por H2A.Z Nucleossomo removido Variantes de Histonas H3 H2A Chaperonas na substituição de histonas Chaperonas de histonas: • Proteínas com forte afinidade por regiões específicas na superfície de H2A:H2B ou de H3:H4 • Propiciam a desmontagem do complexo • Não dependem de ATP Chaperonas que facilitam a transcrição: • Chaperonas de H2A:H2B Nap1 (Nucleosome assembly protein 1) FACT (FACilitates chromatin Transcription) • Nap1 interage com remodeladores de cromatina (Chd1, RSC) e facilita a desmontagem dos dímeros H2A:H2B • Nap1 interage com nucleossomos com H3K14Ac desmontando-os • FACT segue a Pol II durante o alongamento e interage com outros fatores de alongamento • Chaperonas de H3:H4 • Asf1 (antisilence factor 1) • Spt6 (Supressor of Ty homolog 6) • Ambos facilitam o alongamento da transcrição • Asf1 facilita a remoção e a deposição de H3 no promotor e no corpo do gene • Ast1 se associa com a histona acetilase que acetila H3K56Ac, que facilita a troca de H3 e H4 • Spt6 facilita a reincorporação de H3 nos nucleossomos após a passagem da Pol II • Chaperona de H2A.Z • Chz1 (levedura; parte do complexo de remodelagem Swr1) • Chaperonas de H3.3 • HIRA (HIstone Regulator A protein interage com o remodelador Chd1 • Daxx (Death domain associated protein) Ativação da transcrição por tipos de histonas • O complexo remodelador Swr1 troca os dímeros H2A:H2B para formar nucleossomos com o dímero H2A.Z:H2B na extremidade 5’ dos genes • As chaperonas HIRA e Daxx troca H3 por H3.3 nos nucleossomos próximos ao promotor na extremidade 5’ do gene • A duas substituições, H2A.Z e H3.3, torna os nucleossomos mais instáveis, facilitando a remoção dos mesmos do promotor Elementos essenciais na regulação da taxa de transcrição dos genes em eucariotos: Organização dos nucleossomos Remodelagem da cromatina acoplada à montagem dos fatores gerais de transcrição Fatores de transcrição específicos para certas sequências Estados de fosforilação da cauda C-terminal (CTD – C Terminal Domain) da RNA polimerase II (Pol II) associados a modificações na cromatina Os reguladores sequência-específicos orquestram múltiplos aspectos da transcrição pelos seguintes mecanismos: 1 - Recrutamento de remodeladores da cromatina 2 - Recrutamento de complexos modificadores da cromatina 3 - Recrutamento dos fatores gerais de transcrição 4 - Recrutamento da RNA Pol II via complexo Mediator Modificações pós-tracicionais nas caudas das histonas são essenciais na regulação da taxa de transcrição Modificações em lisinas (Lys – K) Modificações pós-transcricionais em histonas que ativam a transcrição • Alteração direta da estrutura da cromatina • Acetilação : • A acetilação neutraliza cargas positivas das lisinas, reduzindo a força de interação entre DNA e histona • Acelitação das caudas de H2A e H2B reduz a interação do DNA com as díades • Acetilação das caudas de H3 e H4 impede formação de estruturas de ordem superior (a cauda de H4 usualmente faz a conexão entre nucleossomos vizinhos) • Ex: H4K16Ac Aumentado em genes do cromossomo X para cpmesenção de dosagem • Recrutamento de complexos remodeladores da cromatina devido a marcas nas histonas • Histona H3 acetilada interage com bromodomínios presentes em subunidades dos complexos remodeladores SWI/SNF e RSC • H3K4me3 recruta Chd1, uma proteína com cromodomínio, para a extremidade 5’ de genes ativos • H3K4me3 + H4K16Ac recruta a subunidade com domínio PHD, BPTF, do complexo ISWI • H3K36me3 recruta histonas-deacetilases com cromodomínio para o corpo do gene, necessárias para a remontagem dos nucleossomos após a passagem da Pol II e para inibir sítios de início de transcrição no corpo do gene. “PARylation” como ativador da transcrição • PARP – Poly-(ADP Ribose) Polymerase • Usa NAD+ para adicionar ADP-riboses em proteínas • Estruralmente similar a ácido nucléico, mas dois grupos fosfato com carga negativa • Pode adicionar até ~ 200 unidades de DAP-ribose linearmente ou com ramificações • Proteínas usualmente modificadas: proteínas de reparo do DNA, fatores de transcrição e histonas • Induzem forte relaxamento da cromatina e depleção de nucleossomos • Marcas de histonas que podem ativar PARP: • Fosforilação C-terminal de H2A • H2AK5Ac Fatores de transcrição que se ligam a sequências específicas (enhancers) MEDIATOR TFIID TBP Enhancers: Elementos no DNA que ativam a transcrição independente de direção, localização e distância em relação ao promotor do gene. Enhancers modulam a transcrição por: • Recrutar enzimas modificadoras de histonas • Recrutar complexos remodeladores de cromatina dependentes de ATP • Recrutar proteínas que substituem histonas • Facilitar o recrutamento da RNA Pol II • Afrouxamento da cromatina • Maior acessibilidade a outras proteínas regulatórias • Formação do PIC • Facilitação do início da transcrição e do alongamento A especificidade que determina a diferenciação celular: Quais enhancers irão participar da regulação? • Informações epigenéticas nos enhancers,adicionadas antes da ativação do gene, permitem a seleção dos enhancers a serem utilizados a cada momento. • Quais moléculas regulam a deposição de marcas epigenéticas em enhancers específicos durante o desenvolvimento e diferenciação celular? Fatores que se ligam a DNA Sequências específicas do DNA Estado de metilação do DNA As marcas H3K4me1/2 e H3.3/H2AZ nos enhancers (além de outras menos conservadas) resultam em afrouxamento da cromatina e depleção de nucleossomos no local, com consequente ativação rápida do gene via recrutamento eficiente de fatores de transcrição e de complexos remodeladores da cromatina Chin-Tong Ong & Victor G. Corces EMBO Reports 13(5):423, 2012 Fatores de transcrição chaves que participam no processo de diferenciação: • Células Tronco Embrionárias Humanas: hESC • Para manter os estado pluripotente: Oct4, Sox2 e Nanog Inibe Diferenciação neural ectodérmica Sox2 Oct4 Ativa Diferenciação mesodérmica Sinais de diferenciação modulam contínua e assimetricamente os níveis de Oct4 e Sox2, alterando seus padrões de ligação no genoma e as decisões quanto ao destino celular Marcação de enhacers para GR (Glucocorticoid receptor): GRE Grau de metilação do DNA e fatores específicos Chin-Tong Ong & Victor G. Corces EMBO Reports 13(5):423, 2012 Células tronco embrionárias p300: histona acetil transferase (HAT) PU.1 – Fator de diferenciação de células da linhagem hematopoiética Oct4 LSD1: (histona demetilase) PU.1 modifica o enhacer com marcas de ativação Chin-Tong Ong & Victor G. Corces EMBO Reports 13(5):423, 2012 A disponibilidade dos enhancers varia com o tipo de célula FoxA1 liga-se a seu enhancer marcado com H3K4me1/2 Mama: FoxA1 recruta ER Próstata: FoxA1 recruta AR Chin-Tong Ong & Victor G. Corces EMBO Reports 13(5):423, 2012 A integração dos sinais provenientes dos enhancers é feita pelo complexo co-ativador MEDIATOR (Mediador) • Por meio do MEDIATOR, um ativador pode influenciar no efeito do outro • Presente desde leveduras a mamíferos, o MEDIATOR é complexo multiprotéico com mais de 1 MDa • O complexo possui 4 domínios: cabeça (head), cauda média (mid tail) e dois domínios cinases • Adquire uma configuração estendida ao contato com RNA Pol II • Elevada flexibilidade conformacional • Duas formas: • Grande (TRAP ou ARC-L) com com o subdomínio Cdk8 (MED12,MED13, ciclina C e Cdk8) • Pequena, sem Cdk8 MEDIATOR: um fator geral de transcrição • Localizado em regiões intergênicas de genes ativos e inativos e em regiões codificantes de muitos genes • Associado a enhancers • Recruta Pol II e propicia a formação do PIC (Pré-Inciation Complex) • Domínio head – sítio multipartite de interação com TBP (TATA Binding Protein) Mediator tem 26 subunidades MED1 a MED26 • MEDIATOR tem mais ações além de recrutar Pol II: • Permite a interação de fatores de transcrição ligados a enhancers com os fatores gerais de transcrição Borggrefe T & Yue X. Seminars in Cell and Developmental Biology. 22:759-768, 2011 1 Um exemplo de sequência de eventos na ativação de um gene Promotor do gene interferon humano 2 2 3 3 Acetyl transferase 4 4 SWI/SNF 5 TFIID Primeiro fator geral de transcrição a se ligar no promotor. Faz parte do complexo TBP Montagem dos fatores gerais de transcrição • O mediador é recrutado precomente na montagem do PIC • Mediador recruta a acetilase p-300 (subunidade de SAGA) • p-300 acetila todos os alvos e de dissocia do promotor • A montagem do PIC continua Fatores gerais de transcrição: TFIID (com TATA biding protein – TBP e TAFs), TFII A, - B, -D, -E, -F e –H Mediator SAGA TF Ac Ac A TFIID TBP B F Ac Ac E PIC = Pré-Iniciation Complex Complexo remodelador • TFIIB e TFIIF são necessários para o recrutamento da RNA Pol II • TFIIE junta-se após Pol II • TFIIE recruta TFIIH e modula sua atividade de helicase e cinase NC2 Mot1 Mediator TF TF Ac TFIID TBP Pol II B F Ac E H PIC pronto para dar a largada CTD Em células tronco embrionárias, > 55% dos genes silenciosos têm RNA Pol II no promotor (Poised or stalling Pol II ) • Após transcrever os primeiros 30-50 nts iniciais, Pol II faz uma pausa • DSIF e NELF inibem a progressão A pausa é essencial para a montagem do complexo de alongamento completo P-Ser5 Ac Ac DSIF H Transcrito inicial NELF Pol II Ac Ac DSIF: DRB Sensitivity Inducing Factor NELF: Negative regulator factor O complexo NELF/DSIF inibe a transcrição depois que o transcrito já atingiu ~30 nts BRCA-1 DSIF A ER-a B E C/D AP-1 NELF: subunidades A, B, C/D e E NELF recruta fatores reguladores da transcrição A pausa induzida por NELF e DSIF parece fundamental para a montagem de todo o complexo de alongamento produtivo YSPTSPS YSPTSPS YSPTSPS Pol II CTD com 52 repetições YSPTSPS 2 P YSPTSPS 5 P YSPTSPS YSPTSPS Pol II Sub-unidade cinase (Kin 28) do TFIIH fosforila CTD em Ser5 e a polimerase sai do promotor mRNA P-Ser5 PAF SAGA TF Ac TBP Ac B Rad6 TFIID F E Fatores Gerais de Transcrição são deixados para trás Fatores de alongamento Bre1 COMPASS H Pol II Complexo PAF conecta Ser5-P a uma rede de modificdores de histonas YSPTSPS YSPTSPS YSPTSPS Pol II Proteínas envolvidas com: modificações em histonas, alongamento, término e processamento do RNA Os complexos ligados à Pol II CTD vão sendo trocados, de acordo com o estado de fosforilação do CTD Pol II CTD conecta-se a escritores, leitores e apagadores do código de histonas Ssu72 fosfatase P YSPTSPS P Cdk1 P-TEFb P YSPTSPS P Positive-elongation factor b P YSPTSPS Pol II Mecanismos epigenéticos • Metilação do DNA • Modificações de Histonas DNA metiltransferases: DNMT1 - DNMT3a - DNMT3b Metilação do DNA permite a herança do padrão de metilação por várias gerações Herança epigenética Células diferenciadas: • Metilação de citosinas ocorre quase que exclusivamente em dinucleotídoes CpG • Ilhas CpG nas regiões promotoras de genes ativos não estão metiladas • Ilhas CpG metiladas ocorrem mais em regiões intragênicas ou intergênicas • Metilação CpG ocorre em promotores alternativos e em promotores de genes inativos • O Padrão de metilação se altera em certas doenças: cancer genoma hipometilado Células tronco: • 25% das 5mC não estão em contexto CG • Metilação mais frequente no corpo dos genes e próximo ao sítio de início de transcrição • Com a diferenciação celular, esse padrão desaparece • Padrão de modificações em histonas também é herdado, contribuindo para a manutenção do estado da cromatina em células diferenciadas Mecanismos que produzem formas epigenéticas de herança num organismo Herança epigenética é perdida na ocasião da formação de células germinativas DNA metiltransferases são direcionadas para o DNA pelas modificações em histonas: • H3K9m e H3K27m • H3K9 atrai HP1 • HP1 recruta DNMT • HP1 recruta HMT garantindo a marca de metilação H3K9 na fita nova Andrew P. Feinberg & Benjamin Tycko 2004 Fêmea Alelo silenciado(imprint ed) do gene A Alelo expresso do gene A Ambos os pais expressam O Mesmo alelo do gene A Macho Cromossomo herdado dos pais Célula somática Célula somática Nas células germinativas o imprinting é removido Meiose Imprinting é restabelecido de forma sexo-específica A prole irá diferir quanto ao alelo do gene A que é expresso Como identificar “Transcription Factor Binding Sites”- TFBS? Ensaios bioquímicos: • DNA footprint • Eletroforetic mobility assay • Capture of transcription-factor-bound sequences DNA footprint Figure 7-27a Molecular Biology of the Cell (© Garland Science 2008) Como identificar “Transcription Factor Binding Sites”- TFBS – ocupados “in vivo"? No genoma humano, de cada 500 TFBS para um dado fator, apenas 1 é ocupado. Ex: GATA-biding factor 1 (GATA1): 8 milhões no genoma; 15.000 ocupados em células eritróides DNA precipitado com a cromatina pode ser identificado por hibridização de arrays ou sequenciado Imunoprecipitaçãoda cromatina para identificação de modificações em histonas Chromatin Immunoprecipitation (ChIP) Immunoprecipitação de histonas + DNA crosslinked ChIP-Chip: DNA ligado a histonas precipitadas é identificado por hibridização de microarrays ChIP e sequenciamento (Next Generation Sequencing) ChIP-Seq Genoma de mamíferos: ~ 1.000 TF As sequências de reconhecimento são curtas e variáveis Enhancers em geral têm mais de um sítio de ligação para TFs: “cisregulatory module” Podem estar próximos ao promotor ou tão distantes quanto 1 Mpb CRM: Cis Regulatory Module Hardison RC & Taylor J. Nature Rev 13:469, 2012 CTCF é a proteína que se liga aos “insulators” impedindo a ação de "enhancers" sobre determinados promotores Impedem também a propagação da heterocromatina Hardison RC & Taylor J. Nature Rev 13:469, 2012 Chip-Seq para 4 fatores de transcrição (TF) em um segmento de DNA. Nem todos os sítios (TFBS) estarão ocupados Quantificação das sequencias de DNA imunoprecipitadas com o anticorpo específico para o fator de transcrição. Hardison RC & Taylor J. Nature Rev 13:469, 2012 Comparação das sequências: região codificante conservada Hardison RC & Taylor J. Nature Rev 13:469, 2012 Avaliação funcional Transfecção Cultivo das células Inserção da sequência contendo o suposto enhancer em vetor de Lise das células e medida da expressão no qual o gene repórter é atividade da Luciferase controlado por um promotor fraco Atividade de Luciferase em relação à atividade basal Hardison RC & Taylor J. Nature Rev 13:469, 2012 A avaliação funcional pode ser feita no animal inteiro Identificar os tecidos que podem ativar o enhancer Identificar a fase do desenvolvimento em que é ativado Hardison RC & Taylor J. Nature Rev 13:469, 2012
Download