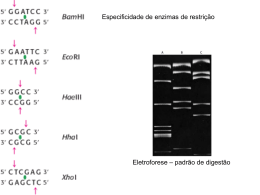
Genome sequence Genome size does not correlate well with gene number or with apparent organism complexity Closely related organisms can have genome sizes that vary by 100x Human genome is 30x smaller than some plant genomes Número de clones teoricamente necessários para representar o genoma Tamanho do genoma (pb) Tamanho do fragmento clonado (pb) 2 X106 2 X107 3 X109 Bacteria Fungo mamíferos 5 X103 400 4.000 600.000 10 X103 200 2.000 300.000 20 X103 100 1.000 150.000 40 X103 50 500 75.000 N= ln (1-P) ln (1 - f ) Seqüênciamento do genoma por “shotgun” Genoma aberto Clonagem e seqüênciamento Clones “shotgun” Bioinformática Alinhamento dos clones Genoma seqüênciado 5’...ATCGGTACCAGGCTCCTCAGAGT...3’ 3’...TAGCCATGGTCCGAGGAGTCTCA...5’ Construção da biblioteca shotgun • Fragmentos de DNA ramdômicos podem ser obtidos usando o processo HydroShear (GeneMachines Inc., USA). • DNA sonicado para construção biblioteca shotgun kb 11,0 7,0 6,0 5,0 4,0 3,0 2,0 1,5 1,0 In shotgun approach, DNA is randomly broken into fragments, and each is sequenced w/out knowing where in the genome the fragment comes from Maria Aparecida Fernandez- DBC - UEM SEQUENCIAMENTO DE DNA Bancada Bioinformática 1.cromossomo ou cosmídeo 2.fragmentação _ enzimática _ nebulização 3.clonagem _ pUC18 4.seqüenciamento retro-alimentação ACCTGATGCCGACGGG 5.montagem 6.finalização Contig 1 Contig 2 Cosmídeo 7.anotação Maria Aparecida Fernandez- DBC - UEM iniciador Automated DNA Sequencing If the chain terminators are labeled instead of the primers, all four reactions can occur in the same tube. SEQUENCIAMENTO DE DNA Maria Aparecida Fernandez- DBC - UEM Typical output of an automated sequencer Estrutura da Rede de Computadores Laboratório de bioinformática PC1 INTERNET Servidor e-mail Web FTP Firewall Pentium III 750 MHz PC2 PC3 Switcher PCn Instituições participantes do projeto Maria Aparecida Fernandez- DBC - UEM Sun 450 Enterprise 4 processadores 4 GB RAM 300 GB HD Montagem do genoma • Phred – identificação de bases (base calling) e valores de qualidade • Cross-match – identificação de seqüências de vetor • Phrap – montagem das seqüências contíguas (contigs) • Consed – análise e edição dos contigs • Finalização – fechamento de falhas (Scaffold), determinação de genes (BLAST/Glimmer) e anotação (Artemis/Sequin) GENOMAS SEQUENCIADOS Bacterial genome Eukaryotic DNA: chromosome Band pattern of human chromosomes Genes in genome The organization of genes on a human chromosome Human genome sequence Comparison of genomes Dogma central da biologia molecular Replicação do DNA Reparo de DNA 5´ Recombinação genética 3´ DNA 3´ Transcrição Síntese de RNA Transcrição 5´ Tradução 5´ 3´ RNA Síntese protéica Tradução H2N COOH Proteína Gene Definição Molecular Seqüência de DNA que codifica uma proteína Não acredito que vocês estão satisfeitos com esta definição!!! Gene Falhas da definição molecular Alguns genomas são constituídos de RNA e não de DNA. Alguns genes produzem RNA (tRNA e rRNA) e não proteínas. Algumas regiões não-codantes são importantes para produção de RNA e proteínas. Gene “ Open Reading Frame” - ORF É a seqüência de nucleotídeos que codifica os aminoácidos de uma proteína. A definição molecular de gene é mais ampla do que apenas a seqüência codante. Finding the regions in a DNA sequence that encode a protein: Computer programs designed to identify ORFs Gene Definição molecular atual Toda seqüência de nucleotídeos necessária para a síntese de uma cadeia polipeptídica ou de RNA funcionais. Gene Regiões não-codantes Regulatórias Sítios de ligação da RNA polimerase Sítios de ligação dos fatores transcricionais Íntrons Sítios de Poliadenilação - poliA Gene Seqüência codante ATG Promotor E1 I1 E2 Cauda de poliadenina I2 E3 PoliA Interruptor do gene Seqüência não-codante Fatores transcricionais RNA polimerase Splicing Introns can be recognized by conserved sequences at the junctions that are required for their removal All parts of the genome are subject to mutation, but not all parts are subject to natural selection Sequences that do not encode protein are not under selective pressure and thus diverge more rapidly Comparative genomics is our most powerful tool for identifying the exons of expressed genes Random mutations leads to extensive sequence variation between humans and mice at all sites that are NOT under selection. Because of functional constraints, the exons in genes stand out as islands of conservation. Distribution of sequence types in the human genome Average human gene is about 20x larger than genes in E. coli or even in yeast Gene density is much higher in compact yeast genome relative to human The partly reflects the compression of gene regulatory sequences into short regions just upstream of the gene. Human regulatory elements can be spread out over tens of 1000s of basepairs The C. elegans genome was completed in 1998 and is predicted to contain 19,000 genes; 3x more than the unicellular eukaryote, S. cerevisiae
Download

















![[TA]7 - Biblioteca Digital do IPB](http://s1.livrozilla.com/store/data/000350244_1-74c23704413cd5a20bb1c551e9d0a0e1-260x520.png)

