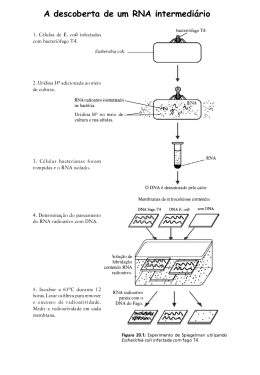
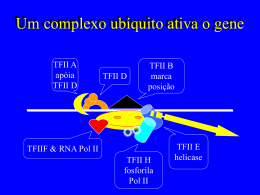
Transcrição do RNA em Organismos Procariotos 1-Aspectos Gerais 2-RNA Polimerase DNA Dependente 3-Região Promotora 4-Transcrição em E. coli 5-mRNA policistrônico Edmar Vaz de Andrade www.ufam.edu.br/~edandrade 1-Aspectos Gerais Comparação entre os processos de transcrição e replicação • Replicação do DNA • Direção da síntese: 5’-3’ • Cromossomo inteiro é copiado • Requer iniciador • As duas fitas da dupla hélice são copiadas • Transcrição do RNA • Direção da síntese: 5’-3’ • Segmentos gênicos são copiados • Não requer iniciador • Somente uma fita serve como molde em um dado momento 1-Aspectos Gerais • Tipos de RNAs transcritos – mRNA: codifica a seqüência de aminoácidos de cadeias polipeptídicas. – tRNA: transporta um aminoácido específico durante a síntese protéica. – rRNA: interage com proteínas constituindo os ribossomos (maquinário da síntese protéica). – RNAs reguladores: regulação de diferentes processos celulares 2-RNA Polimerase DNA Dependente Bolha de transcrição Fita codificadora RNA polimerase Re-anelamento Desanelamento Fita molde DNA-RNA híbrido, 8 pb Sítio ativo Direção da transcrição Transcrição pela RNA polimerase na Echerichia coli RNA polimerase DNA dependente 2-RNA Polimerase DNA Dependente Fita 5’-3’: fita codificadora Fita 3’-5’: fita molde RNA transcrito O RNA é transcrito a partir da fita molde e sua sequência é idêntica a da fita codidificadora (U/T) 2-RNA Polimerase DNA Dependente Polimerização mediada pela RNA polimerase 3-Região Promotora Espaçadores TTGACA Região -35 N17 TATAAT N6 Região a ser transcrita Região -10 Sítio de início de transcrição (+1) Sequência conservada de promotores de E. coli reconhecidos pela subunidade sigma 70 2-RNA Polimerase DNA Dependente Genes essencias para a manutenção celular – house keeping genes Genes regulados por nitrogênio Genes relacionados com a adaptação ao estresse térmico Genes relacionados com quimiotaxia e síntese de flagelos Genes relacionados com a adaptação ao estresse térmico extremo Subunidades sigma descritas em E. coli [Sadhale P, Verma J and Naorem A 2007 Basal transcription machinery: role in regulation of stress reponse in eukaryotes; J. Biosci. 32 569–578] 3-Região Promotora • Eficiência da ligação da RNA polimerase ao promotor – Sequência nucleotídica do promotor – Espaçamento entre as regiões -10 e -35 – Distância entre as regiões -10 e -35 e o sítio de início de transcrição 3-Região Promotora Reconhecimento do promotor pela subunidade sigma em E. coli [Sadhale P, Verma J and Naorem A 2007 Basal transcription machinery: role in regulation of stress reponse in eukaryotes; J. Biosci. 32 569–578] 4-Transcrição em E. coli Etapa de início da transcrição 4-Transcrição em E. coli Etapa de elongação da transcrição 4-Transcrição em E. coli Etapa de término da transcrição 4-Transcrição em E. coli Etapa de término da transcrição independente da proteína ρ (ro) 5-mRNA policistrônico Decodificação de vários genes a partir de um único mRNA Transcrição do RNA em Organismos Eucariotos 1-Aspectos gerais 2-Complexo de iniciação 3-Processamento Pós-Transcricioanal 4-DNA polimerase RNA dependente 1-Aspectos gerais mRNA monocistrônico 1-Aspectos gerais Tipos de RNA polimerase em eucariotos • RNA polimerase I – Transcrição de rRNA • RNA polimerase II – Transcrição de mRNA • RNA polimerase III – Transcrição de tRNA, rRNA e RNAs reguladores 1-Aspectos gerais Seqüências reguladoras Seqüência TATA Seqüência Inr Promotores reconhecidos pela RNA polimerase II de eucariotos. 2-Complexo de iniciação Dobramento da duplahélice quando associada a proteína TBP Fatores gerais de transcrição (TF) Reconhecimento da região promotora pela TBP (Proteína Ligante da Região TATA) 2-Complexo de iniciação Domínio N-terminal Inr Fatores gerais de transcrição (TF) Reconhecimento da região promotora pela TBP (Proteína Ligante da Região TATA) 2-Complexo de iniciação Interação da RNA polimerase II com o promotor TFIIF: posiciona a RNA polimerase ao promotor e ao sítio Inr 2-Complexo de iniciação Sítio de ancoragem para TFIIH TFIIH Helicase e fosforilação do domínio C-terminal de RNA Pol II Conclusão da etapa de iniciação 3-Processamento Pós-Transcricional Processamento do prémRNA eucarioto 3-Processamento Pós-Transcricional 7-metilguanilato Ligação 5’-5’ fosfato Estrutura do quepe 5’ metilado do mRNA de eucariotos 3-Processamento Pós-Transcricional Ponto de forquilha Sítio de corte e emenda 5’ Sítio de corte e emenda 3’ Região rica em bases pirimidinas Seqüências conservadas que regulam o splicing nos pré-mRNAs de eucariotos 3-Processamento Pós-Transcricional 1a reação de transesterificação Mecanismo geral do splicing 2a reação de transesterificação 3-Processamento Pós-Transcricional Reconhecimento dos sinais de poli(A) por fatores protéicos Clivagem e poliadelilação do prémRNA eucariótico Ligação da poli(A) polimerase (PAP) 3-Processamento Pós-Transcricional Poliadenilação lenta (≈ 12 resíduos) Clivagem no sítio de poli(A) Clivagem e poliadelilação do prémRNA eucariótico 3-Processamento Pós-Transcricional PABII: regula a atividade da enzima poli(A) polimerase Proteína II de ligação à poli(A) PABII Clivagem e poliadelilação do pré-mRNA eucariótico Quando não mencionado, as figuras apresentadas foram retiradas, com ou sem modificações, das seguintes referências: 1. LODISH, H; DARNELL, J. E. e BALTIMORE, D. 2005. “Biologia Celular e Molecular”, 5ª edição. Artmed Editora SA. Porto Alegre-Brasil. (www.whfreeman.com/lodish) 2. LEHNINGER, A. L. NELSON, D. L e COX, M. M. 2000. “Principles of Biochemistry”. 3ª Edition, Worth Publitions.
Baixar