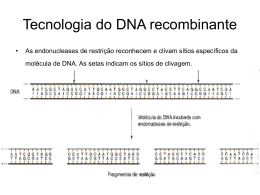
Manual da oficina prática de Genética, Genoma e Biotecnologia Terceiro Módulo www.odnavaiaescola.org deste Módulo Introduação à Bioinformática Conteúdo 1) Síntese de c-DNA Em 1988 o governo americano lançou o primeiro banco de dados público contendo sequências de DNA dos mais diversos organismos. Este repositório de seqüências recebeu o nome de Centro Nacional para Informação em Biotecnologia (NCBI-National Center for Biotechnology Information). Hoje este centro tem várias ramificações no mundo inteiro e além do banco de dados propriamente dito, o NCBI proporciona um grande número de ferramantas de informática e recursos para auxiliar o cientista na pesquisa genética. A utilização cada vez maior da informática no estudo da pesquisa biólogica e ainda mais específicamente no estudo dos genes deu origem à disciplina conhecida como bioinformática. A bioinformática é ainda considerada uma recente subdivisão da biotecnologia e representa o “casamento” da biotecnologia com a informática. De modo simples, bioinformática consiste no depósito e análise de sequências genéticas em bancos de dados e consequente manipulação e análise destas sequências com a utilização de software específicos. O estabelecimento de bancos de dados públicos possibilita que os cientistas possam ter acesso à informação proveniente de outros laboratórios e possam também trocar e compartilhar seqüências genéticas. Todos os dias novas seqüências são depositadas no banco de dados púbicos do NCBI, o chamado GenBank. A bioinformática tem revolucionado o desenvolvimento da pesquisa médicobiólogica e tem possibilitado que novas descobertas relevantes sejam realizadas quase todos os dias. Hoje a maioria dos biólogos envolvidos com a pesquisa genética não estão restritos apenas ao trabalho de laboratório devendo os mesmos dedicar grande parte do seu tempo à bioinformática. 2) 3) 4) 4) Genbank e banco de sequências Pesquisa BLAST, Comparação de sequências Mapa de restrição Anotação de genomas Este texto foi escrito por Francisco Prosdocimi (transcrito com permissão do autor). Este artigo foi baseado, principalmente, no artigo da Revista Nature Reviews, volume 2, julho de 2001, páginas 493-505, Genome annotation: from sequence to biology, que foi escrito por Lincoln Stein. Por milhares de anos, rabinos têm trabalhado sobre os textos do Torah, tentando fazer com que esse livro sagrado, tão difícil de ser entendido, seja traduzido num conjunto de regras e condutas mais claras, de forma a montar uma versão compreensível desse texto, conhecida como Talmud. Com o passar do tempo, a grande quantidade de anotação contida no Talmud chegou a ultrapassar, de longe, o tamanho do texto original, o Torah. A mesma analogia pode ser feita para os genomas. A seqüência de DNA é uma fonte rica de informações sobre a biologia dos organismos, mas deve ser traduzida e anotada de forma correta para que possamos obter corretamente essas informações. A anotação genômica consiste num processo de vários passos, havendo, mais ou menos, três Todos os direitos reservados à DNA Goes to School, Inc 2003 1 categorias básicas: a anotação em nível de nucleotídeos, anotação em nível de proteínas e anotação em nível de processo. Na anotação em nível de nucleotídeos procura-se encontrar a localização física das seqüências de DNA e descobrir onde estão os genes, RNAs, elementos repetitivos, etc. Na anotação em nível protéico procura-se descobrir a provável função dos genes, identificando quais são aqueles que determinado organismo possui e quais ele não possui. Já a anotação em nível de processo procura identificar as vias e processos nos quais diferentes genes interagem, montando uma anotação funcional eficiente. ® Chico On Line. Prosdocimi, F. 2001-2. Como as seqüências estão depositadas no GenBank? Para que você possa realizar as atividades interativas você tem que entender como as seqüências de DNA e RNA estão depositadas no GenBank. As sequências estão sempre depositadas em forma de fita única, mesmo que seja um fragmento de DNA. Isso porque quando o DNA é transcrito para a forma de RNA apenas uma das fitas é transcrita, a que chamamos de fita senso. A outra fita complementar à molécula de DNA, chamada de fita anti-senso não é transcrita para a forma de RNA. Deste modo, apenas a fita senso de DNA é depositada no banco de dados. É claro que isso é válido para os casos onde o gene é conhecido. Quando não se conhece o gene, qualquer uma das fitas pode ser a fita depositada. Mas não se preocupe porque só trabalharemos com genes conhecidos. Então, o primeiro ponto que você tem que ter em mente é que a fita de DNA depositada é a fita senso, que é transcrita para a forma de RNA. O segundo ponto que você deve saber é que a molécula de RNA mensageiro, apesar de, na realidade, corresponder à fita complementar do DNA , não está depositada no Genbank desta forma. Ao invés disso, o RNA mensageiro depositado no GenBank é semelhante à fita senso de DNA. Então qual é a diferença entre a fita senso de DNA e fita de RNA mensageiro depositadas no GenBank? A diferença está no fato de que o RNA mensageiro constitui apenas os exons da molécula de DNA pois os introns são removidos durante o processo de edição do RNA. Deste modo o RNA mensageiro é sempre menor que a fita senso de DNA. Veja, o exemplo abaixo. As letras em maiúscula correspondem aos exons de um determinado gene e as letras em minúscula correspondem aos introns. Vamos supor que um fragmento de DNA de uma determinada fita senso seja da seguinte forma: ATGCGTTACCTTAGtcagtctcatcTCAGGTGTCatcttttcttaCTGATC O RNA mensageiro correspondente à esta fita senso estará depositado no GenBank da seguinte forma: ATGCGTTACCTTAGTCAGGTGTCCTGATC 2 É importante que você faça as seguintes observações: · O RNA mensageiro constutui apenas os exons da fita original de DNA · O RNA mensageiro NÃO está na forma de fita complementar · O RNA mensageiro não está depositado com suas bases de uracil (que é o que na realidade ocorre), mas sim com as bases de timina A explicação deste fato está na síntese de DNA complementar, ou cDNA. A maioria das seqüências de RNA que estão no GenBank foram obtidas através da síntese de DNA complementar. O cDNA é uma molécula similar ao DNA porém esta é sintetizada no laboratório. A síntese de cDNA é realizada com a transcriptase reversa, uma enzima de origem viral que tem a capacidade de produzir uma molécula de DNA dupla fita a partir da cópia de uma molécula de RNA. Ou seja, como o nome diz, a transcriptase reversa faz o caminho contrário daquele percorrido pela RNA polimerase, que a partir de uma molécula dupla fita de DNA produz RNA. Veja a figura abaixo. A molécula de DNA produzida pela transcriptase reversa é chamada de DNA complementar (ou cDNA) e é formada pelos mesmos compostos encontrados na molécula de DNA (açucar desoxirribose, grupos fosfato, ponte de hifdrogênio e as 4 bases nitrogenadas, adenina, timina, guanina e citosina). A descoberta desta enzima viral e de seu mecanismo de ação abriu um importante caminho na biologia molecular. A molécula de RNA é extremamente instável e dependendo do RNA em questão o número de cópias pode estar muito reduzido. Deste modo, a possibilidade em se trabalhar com moléculas de cDNA ao invés de RNA tem facilitado muito o trabalho do cientista no laboratório. O cDNA é uma molécula estavél, de fácil manuseio e sua multiplicação é bastante simples. 3 Atividade 1: Procurando um gene no GenBank- Seqüência de Imagens Passo 1 4 Passo 2 Passo 3 5 Passo 4 Passo 5 6 Passo 6 7 Atividade: Cachorro, o melhor amigo do homem Passo 1 Passo 2 8 Passo 3 Atividade: Em buusca da mutação da anemia falciforme Passo 1 9 Passo 2 Passo 3 10 Mapas de Restrição Veja abaixo os mapas de restrição que você irá obter no exercício Cortando com o Webcuter Exercício 1: Polimorfismo do gene ALDR 11 Exercício 2: Seqúência do gene VLCS (Very Long Chain Synthetase) Após realizar a atividade no computador, tente responder às seguintes perguntas: 1) O que é bioinformática? 2) Se o DNA é uma fita dupla, por que as sequências aqui apresentadas só têm uma fita? 3) A partir da realização da atividade “ Cachorro, o melhor amigo do homem..”, você concluiria que o homem e os cachorros apresentam 91% de similaridade em seu DNA? 4) O que é mapa de restrição e como o mesmo pode ser usado? 5) O que é o GenBank? 12 Tabela de Aminoácidos 13 Bioinformática, a profissão do futuro Renato Sabbatini (disponível em http://www.epub.org.br/correio/cp990430.htm) Nos últimos anos, as seções de oferta de empregos na área biomédica têm estado lotadas de anúncios à procura de um profissional ainda desconhecido da maioria das pessoas: o especialista em bioinformática. Esta é uma ciência novissima (existe há menos de 10 anos), que tem como objetivo desenvolver e aplicar técnicas computacionais no estudo da genética, da biologia molecular e da bioquímica. Entre outras coisas, ela é essencial para a construção de bases de dados contendo informações sobre os genes e proteínas dos organismos vivos, para a descoberta de novos genes, e de novos medicamentos. Usando alta tecnologia, o bioinformata é muito valorizado pela crescente demanda e pelo ainda pequeno número de pessoas capazes de preenchê-las. Os maiores empregadores são as universidades, empresas farmacêuticas e de informática, institutos de pesquisas privados e do governo. Os salários iniciais são altos, e um especialista com muitos anos de experiência pode ganhar muito dinheiro, particularmente nos grandes laboratórios multinacionais. O verdadeiro oceano de dados que está sendo gerado por projetos como o Genoma Humano, e a expansão no setor de biotecnologia médica, são os principais responsáveis por esta explosão na demanda por bioinformatas. Existem hoje grandes repositórios de dados genéticos e bioquímicos, como o GenBank (que contém todas as seqüências de DNA conhecidas até hoje e que ocupa o equivalente a mais de 100 CD-ROMs). O Projeto Genoma Humano, por exemplo, tem como objetivo descobrir o código genético dos cerca de 80.000 genes humanos e utilizar esse conhecimento para tratar doenças genéticas. Onde encontrar um bioinformático, ou como formá-lo? É uma tarefa ainda bastante difícil. Geralmente é um biólogo ou médico que tem grandes conhecimentos de informática, genética e bioquímica. Quase não existem cursos sobre esse tema, e a maioria dos profissionais da área é autodidata. Antes de tudo, é preciso ter uma cabeça interdisciplinar, ou seja, ter múltiplos talentos, tais como matemática, estatística, computação, instrumentação e bioengenharia, genética, biologia, etc. O Brasil também se ressente da falta desses profissionais, embora já estejamos embarcados em projetos genômicos grandes e multi-institucionais, como o Genoma da Xylella fastidiosa (um microorganismo que ataca os laranjais, causando a doença chamada “amarelinho”, de grande importância econômica para o estado de São Paulo) e o Projeto Genoma e Câncer, ambos da FAPESP (Fundação de Amparo à Pesquisa do Estado de São Paulo), que montou uma organização para o sequenciamento genético (ONSA). A universidade precisa urgentemente se movimentar para formar esse profissional crucial para a medicina do próximo século! No exterior (como na Baylor University, no Texas), já existem até cursos em nível de graduação sobre bioinformática. E aqui? Tirando uns poucos centros, como o da UNICAMP (Grupo de Biologia Computacional do Instituto de Computação, Centro de Biologia Molecular e Engenharia Genética, Núcleo de Informática Biomédica), estamos ainda no começo. Mas, sem dúvida, é a profissão do futuro. 14 Bioinformática: manual de utilização João Paulo Kitajima “Experiência é o nome que damos a nossos erros.” Oscar Wilde A bioinformática é uma área fascinante. Vejo nela a confluência de meus vários ideais de profissão: a multidisciplinaridade e o equilíbrio entre teoria e prática. O contato com biólogos, agrônomos, farmacêuticos e químicos me tem sido muito enriquecedor. Estudar a vida é uma experiência única e participar deste estudo me é muito gratificante. Além destas razões, pessoalmente, tornar-me bioinformata me possibilitou alinhar com a área de trabalho de meu velho pai, agrônomo e microscopista de longa data. Ao me envolver com biologia molecular aplicada a plantas, consigo entendê-lo melhor, como profissional e mesmo como pai. Tenho trabalhado como bioinformata desde 1999, quando deixei para trás uma carreira promissora como professor adjunto no Departamento de Ciências da Computação da Universidade Federal de Minas Gerais. Cansado de empregar o dinheiro público em projetos sem retorno mais imediato para a sociedade, decidi mudar para uma área de trabalho mais aplicada. Cheguei mesmo a prestar vestibular para agronomia e economia, mas a necessidade de um salário falou mais alto e consegui uma bolsa de pósdoutorado da Fapesp no Laboratório de Bioinformática (LBI) do Instituto de Computação da Unicamp, então coordenado por João Carlos Setubal e João Meidanis. No início tudo era muito estranho, afinal de contas meu último contato com DNA havia sido no segundo grau. Mal me lembrava do que era o dogma central da biologia molecular, proteínas, enzimas e ribossomos. E, de repente, estava eu lá, fazendo informática para biólogos, envolto por uma atmosfera efervescente de desafio. Afinal de contas, a bioinformática do primeiro projeto genoma do Brasil estava acontecendo ali, ao meu lado. E depois do genoma da Xylella fastidiosa, outros vieram. Em meados de 2001, o destino me chamava para novos desafios. João Meidanis já havia mudado de rumo ao partir, alguns meses antes, para a iniciativa privada na área de software e serviços de bioinformática. João Setubal alçava vôos mais altos também, trabalhando e realizando contatos acadêmicos no exterior. A ironia do destino me fez bater, em dezembro de 2001, nas portas da Votorantim Ventures (VV). Naquela mesma época, a Votorantim havia decidido investir em empreendimentos de risco e, na área de biotecnologia, as propostas de investimento estavam sendo analisadas por um grupo chefiado por Fernando Reinach, um dos big boss da ciência no país. Na verdade, eu o havia procurado para me orientar sobre como submeter um projeto para a VV. Mas, nem tive tempo de expor os meus planos. Fernando já havia uma contraproposta: juntar-se, como sócio e diretor, a um novo grande empreendimento que estava surgindo na área de genômica aplicada a plantas - a Alellyx. Chamo isto tudo de “ironia do destino” porque, alguns meses antes desta proposta, ir para a iniciativa privada era para mim algo totalmente fora de cogitação. Eu sentia mesmo ojeriza a esta possibilidade. Mas eu estava vendo as pessoas a meu redor tomarem novos rumos e eu precisava encontrar o meu. Voltar para a universidade também estava fora de cogitação. O triângulo ensino-pesquisa-extensão era pesado demais para mim. Eu precisava de algo com mais foco. Confesso que o projeto Alellyx era tentador. Impus algumas condições e declarei minha completa inexeperiência em assuntos empresariais. Condições aceitas e inexeperiência tolerada, levamos, eu e mais 4 sócios, 3 meses para finalizar o projeto. Começamos a operar em março de 2002. Durante aquele ano, compartilhei a coordenação da bioinformática com João Setubal. Em 2003, com a volta de Setubal para a academia, assumi sozinho esta área na empresa. Atualmente, conto com uma equipe de primeira linha, entre bioinformatas, analistas de sistemas e de suporte e me considero privilegiado em estar onde estou. Vivi, nestes últimos 4 anos, então, diferentes experiências na área de bioinformática, do aprendizado à gerência, do público para o privado. Apesar do pouco tempo, esta heterogeneidade de situações me permitiu chegar a algumas conclusões que podem servir de orientação para quem pretende trabalhar na 15 área. Assim, como outras áreas emergentes de trabalho, a bioinformática se enquadra em um contexto onde o problema “não é hardware, nem software, mas peopleware”, como dizem por aí. Não considero a bioinformática a profissão do futuro, mas penso que ela exige profissionais do futuro. Um profissional de bioinformática bem colocado precisa: -Ter um bom conhecimento em ciência da computação: isto significa basicamente possuir uma boa capacidade de abstração algorítmica (isto é, saber traduzir a solução de um problema em um conjunto de programas eficazes e eficientes). Familiaridade e gosto por computação são condições obrigatórias para o sucesso na área; -Conhecer os príncipios da biologia molecular e as diferentes técnicas de bancada nos diferentes domínios onde a bioinformática se faz necessária (as “ômicas” principalmente: genoma, transcriptoma, proteoma e metaboloma). Gostar de biologia é essencial. Parece óbvio que um bioinformata qualificado precise preencher as duas condições acima. Mas existe uma condição adicional que, em várias situações, eu prezo muito mais do que a capacidade técnica e que muitas vezes não vem explicitada nos currículos dos profissionais: a capacidade de compreender o que os usuários ou clientes necessitam para resolver os problemas. Esta condição é vital para qualquer contexto de análise de sistemas, mas, em bioinformática, ela é mais dramática. Os bioinformatas atendem um público que está envolvido com um negócio bastante complexo: ciência. Biólogos são cientistas e muitas vezes a solução bioinformática que necessitam faz parte do processo da descoberta. Ou seja, nem mesmo os usuários sabem exatamente o que querem e onde querem chegar. O bom bioinformata deve estar ciente deste fato e sintonizado com o usuário. O bioinformata deve ser parte da definição da solução, antes mesmo da implementação dela. Visto de outro ângulo, um bioinformata deve, antes de tudo, saber se relacionar com pessoas e saber se colocar no lugar dos cientistas. Em geral, profissionais de computação que trabalham em ambientes de P&D devem possuir esta característica. Na formação da minha equipe na Alellyx, eu valorizo muito mais um profissional mediano entrosado e com foco do que um aluno nota 10 na faculdade, mas anti-social e disperso. Enfim, a bioinformática é uma área de trabalho que exige um profissional multidisciplinar. Isto implica obrigatoriamente na facilidade em se relacionar com diferentes tipos de profissionais (aqui, informatas se relacionando com outros informatas e com biólogos). Além do mais, deve possuir boa capacidade de abstração pois os problemas não são claros e as soluções menos ainda. João Paulo Kitajima é diretor de Bioinformática/Alellyx Applied Genomics. Texto tirado de http://www.comciencia.br/reportagens/bioinformatica/12.htm 16
Baixar