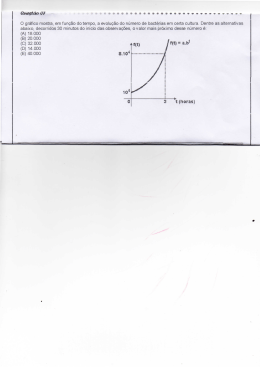
1 CENTRO DE SAÚDE E TECNOLOGIA RURAL CAMPUS DE PATOS-PB PROGRAMA DE PÓS-GRADUAÇÃO EM MEDICINA VETERINÁRIA BACTÉRIAS QUE DEGRADAM MONOFLUOROACETATO DE SÓDIO EXPEDITO KENNEDY ALVES CAMBOIM PATOS-PB 2012 2 EXPEDITO KENNEDY ALVES CAMBOIM BACTÉRIAS QUE DEGRADAM MONOFLUOROACETATO DE SÓDIO Tese apresentada ao Programa de Pós-Graduação em Medicina Veterinária da Universidade Federal de Campina Grande como requisito parcial para obtenção do título de Doutor em Medicina Veterinária. Prof. Dr. Franklin Riet-Correa Orientador Profa. Dra. Marcia Almeida Melo Co-Orientadora PATOS-PB 2012 3 FICHA CATALOGRÁFICA Dados de Acordo com AACR2, CDU E CUTTER Biblioteca Setorial - CSTR/UFCG – Campos de Patos/PB C176b 2012 Camboim, Expedito Kennedy Alves Bactérias que degradam monofluoroacetato de sódio / Expedito Kennedy Alves Camboim - Patos: CSTR/PPGMV, 2012. 56 f. Inclui bibliografia. Orientador (a): Franklin Riet-Correa Tese (Doutorado em Medicina Veterinária). Centro de Saúde e Tecnologia Rural, Universidade Federal de Campina Grande. 1 – Toxicologia veterinária. 2 – Biologia molecular. 3 – Bactérias. 4 – Fluoroacetato dehalogenase – Título. CDU: 615.9 4 Nome: CAMBOIM, Expedito Kennedy Alves Título: Bactérias que degradam monofluoroacetato de sódio Tese apresentada ao Programa de Pós-Graduação em Medicina Veterinária da Universidade Federal de Campina Grande como requisito parcial para obtenção do título de Doutor em Medicina Veterinária. Aprovada em 09/11/ 2012 BANCA EXAMINADORA ___________________________________________ Prof. Dr. Franklin Riet-Correa - Orientador Universidade Federal de Campina Grande – Campus de Patos/PB Unidade Acadêmica de Medicina Veterinária ___________________________________________ Prof. Dr. Luciano Nakazato Universidade Federal do Mato Grosso – Cuiabá/MT Laboratório de Biologia Molecular ___________________________________________ Prof. Dr. Edson Moleta Colodel Universidade Federal do Mato Grosso – Cuiabá/MT Departamento de Clínica Médica Veterinária ___________________________________________ Prof. Dr. Sergio Santos Azevedo Universidade Federal de Campina Grande – Campus de Patos/PB Unidade Acadêmica de Medicina Veterinária ___________________________________________ Prof. Dr. Sara Vilar Dantas Simões Universidade Federal de Campina Grande – Campus de Patos/PB Unidade Acadêmica de Medicina Veterinária 5 "Uma família feliz nada mais é do que o paraíso antecipado". Aos meus pais Davi e Preta, irmãos Kadmu e Kadna, pelo carinho, dedicação e compreensão, dedico. 6 AGRADECIMENTOS Ao Deus Pai, por guiar-me nesta caminhada, por me dar forças para enfrentar as dificuldades do dia a dia e por estar presente iluminando os caminhos de minha vida. Aos meus pais Davi e Maria de Fátima (Preta) e irmãos Kadmu e Kadna pelo amor, carinho e incentivos constantes em mais uma vitória em nossas vidas. Aos meus tios, primos, avós, por fazerem uma família unida e feliz. A minha Co-orientadora, que foi orientadora, Professora Marcia e ao Professor Paulo Andrade, pela confiança, atenção, amizade e pela minha formação. Ao meu Orientador Professor Riet, pelos ensinamentos e exemplo profissional. Aos professores Sara, Eldinê, Rosane, Flávio, Edisio, Sérgio, Moraes, Olaf, Pedro Isidro, Carlos Peña e Josemar Marinho pelos ensinamentos profissionais e pessoais. Aos colegas e amigos de Pós-Graduação: Adriana, André, Clarice, Adílio, Renault, Lizziane, Valéria, Tatyane, Albério, Giovana, Luciano e Rômulo pela amizade e por lutarem pelos seus ideais e darem continuidade ao aprendizado. A equipe do laboratório de Biologia molecular: Tereza, Eduardo Vaz, Vanessa, Arthur, Aline, Gilsane, pela convivência, trabalhos realizados, apoio que me ofereceram, pelos momentos de desabafo e amizade, que foram essenciais para esse sucesso. À UFCG e ao Programa de Pós-Graduação em Medicina Veterinária pela oportunidade de oferecer este curso de Pós-Graduação. À UFPR por ter colaborado para conclusão da pesquisa, especialmente aos professores Emanuel Maltempi e Fábio Pedrosa, a Michelle, Walter, Marcelo e Helisson. Ao INCT para o controle de plantas tóxicas por ter financiado toda esta pesquisa. À EMATER Paraíba e aos meus colegas e amigos Bruno, Branco, Bezerra, Jailson, Cícero, Chico Acácio, Bosco, Ronaldo, Socorro, Mariazinha, Luzimar, Geovanne Medeiros, Jeferson, Alexandre Alfredo, Romero, Marineide, Marconi, Alan Bergman, Franklyn, Marcigleudo, Madeline, Celiane, Esdras e Vespucci pelo profissionalismo, companheirismo e amizade. Aos membros da banca examinadora, por aceitarem o convite e por suas valiosas considerações. A todos os amigos que tenho. Todas estas pessoas foram muito importantes para mim durante esta caminhada. Muito obrigado! 7 SUMÁRIO Introdução ........................................................................................................... Referências ......................................................................................................... Pag. 8 9 CAPÍTULO I Defluoração de fluoroacetato de sódio por bactérias de solo e plantas no Brasil Resumo ............................................................................................................... Introdução............................................................................................................ Material e Métodos …......................................................................................... Coleta das amostras ............................................................................... Isolamento bacteriano .......................................................................... Identificação da sequência do gene 16S rRNA ..................................... Análise das sequências e filogenia ........................................................ 11 12 13 13 13 14 15 Resultados ........................................................................................................... Discussão ............................................................................................................ Referências ......................................................................................................... 15 19 21 CAPÍTULO II Isolamento de bactérias que degradam fluoroacetato de sódio de amostras de rúmen de caprino no Brasil Resumo ............................................................................................................... Introdução............................................................................................................ Material e Métodos …......................................................................................... Coleta e processamento das amostras .............................................. Isolamento bacteriano .......................................................................... Identificação da sequência do gene 16S rRNA ..................................... Análise das sequências e filogenia ........................................................ 25 26 27 27 27 28 28 Resultados e Discussão........................................................................................ Referências ......................................................................................................... 29 35 CAPÍTULO III Ubiquidade da fluoroacetato dehalogenase em bactérias: uma abordagem in silico Resumo ............................................................................................................... Abstract .................................................................................................. Introdução............................................................................................................ Material e Métodos …......................................................................................... Resultados e Discussão........................................................................................ Referências ......................................................................................................... Considerações finais ........................................................................................... 40 41 41 43 43 53 56 8 LISTA DE TABELAS Pag. CAPITULO I Defluoração de fluoroacetato de sódio por bactérias de solo e plantas no Brasil Tabela 1 Resultados do BLAST para as sequências do gene 16S rRNA obtidas de bactérias isoladas de amostras de solo e plantas ..................................... 16 CAPITULO II Isolamento de bactérias que degradam fluoroacetato de sódio de amostras de rúmen de caprino no Brasil Tabela 1 Resultados do alinhamento local das sequências bacterianas de 16S rRNA dos isolados que degradam fluoroacetato. As sequências foram comparadas àquelas depositadas no National Center for Biotechnology Information, utilizando o algoritmo regular BlastN disponível em http://www.ncbi.nlm.nih.gov/BLAST. Os organismos com sequências similares aos isolados ECPB08 (JQ345720) e ECPB09 (JQ345721) estão listadas na segunda coluna. Os valores dos parâmetros de escore total, cobertura e identidade máxima estão apresentados nas colunas 3 a 5. O código de acesso ao GenBank está apresentado em parênteses ................ 30 CAPITULO III Ubiquidade da fluoroacetato dehalogenase em bactérias: uma abordagem in silico Tabela 1 Alinhamento das sequências de fluoroacetato dehalogenases entre Burkholderia sp. (gi|95102016), Micromonospora lupini str. lupac 08 (gi|386690726), Moraxella sp. (gi|216773) e Rhodopseudomonas palustris (gi|81829712) e as sequências enzimáticas de bactérias que possuem a mesma tríade catalítica (seleção em preto), motivos estruturais (seleção em cinza claro) e confirmado/ provável resíduo de sítio ativo (seleção em cinza escuro) ............................................................................................... 45 Tabela 2 Alinhamento das sequências de fluoroacetato dehalogenases entre Burkholderia sp. (gi|95102016), Micromonospora lupini str. lupac 08 (gi|386690726), Moraxella sp. (gi|216773) e Rhodopseudomonas palustris (gi|81829712) e as 108 sequências enzimáticas de bactérias que possuem algumas alterações no sítio ativo. Tríade catalítica (seleção em preto), motivos estruturais (seleção em cinza claro) e confirmados/ prováveis resíduo de sítio ativo (seleção em cinza escuro) .................................... 47 Tabela 3 Percentual de substituição dos aminoácidos do sítio ativo das 108 sequências disponíveis no banco de dados de sequências proteicas do NCBI, em relação à fluoroacetato dehalogenase de Burkholderia sp. (gi|95102016) ..................................................................................... 51 9 LISTA DE FIGURAS Pag. CAPITULO I Defluoração de fluoroacetato de sódio por bactérias de solo e plantas no Brasil Figura 1 Árvore filogenética baseada em sequências 16S rRNA pelo método de máxima parcimônia. ECPB01 a ECPB07 representam os códigos dos isolados e Methanobacterium sp. MO-MB1 (gi|311141366|dbj|AB598270.1|) o grupo externo. A história evolucionária foi inferida utilizando o método de Máxima parcimônia. A árvore consenso do bootstrap foi inferida a partir de 1000 repetições e representa a história evolutiva do táxon examinado. O percentual de árvores de consenso nos quais os táxons se agruparam no bootstrap (1000 réplicas) é mostrado perto dos ramos. A árvore foi obtida utilizando o método Close-NeighborInterchange com algoritmo de busca nível 1, no qual as árvores iniciais foram obtidas mediante a adição aleatória de sequências (10 réplicas). A análise evolutiva foi realizada pelo método MEGA5 ..................................... 18 Figura 2 Taxa de degradação do fluoroacetato de sódio por bactérias isoladas de amostras de solo e plantas no Estado da Paraíba, Brasil. Os símbolos são como se segue. O círculo branco: ECPB01, o triângulo preto: ECPB02, o quadrado preto: ECPB03, o círculo cinza: ECPB04, o quadrado cinza: ECPB05, o triângulo cinza: ECPB06, o círculo preto: ECPB07 ................... 19 CAPITULO II Isolamento de bactérias que degradam fluoroacetato de sódio de amostras de rúmen caprino no Brasil Figura 1 Árvore filogenética baseada nas sequências do gene do 16S rRNA por análise de Máxima Parcimônia. ECPB08 (JQ345720) e ECPB09 (JQ345721) são os códigos dos isolados. Em parênteses estão os códigos do GenBank. É apresentada a relação entre ECPB08 (JQ345720) (a), ECPB09 (JQ345721) (b), os táxons relacionados e os grupos externos Achromobacter ruhlandii, A. denitrificans e Starkeya novella. A árvore de consenso bootstrap inferida a partir de 1000 repetições mostra a história evolutiva dos táxons analisados. Escala de 0,002: distância evolutiva ....................................................... 32 Figura 2 Taxa de degradação de fluoroacetato de sódio por bactérias isoladas de rúmen caprino. Isolado ECPB08 (quadrado cinza claro), isolado ECPB09 (losango preto), e P. fluorescens (triângulo cinza escuro, controle positivo). 33 8 INTRODUÇÃO As plantas e os animais são habitantes de nosso planeta em relação de mútuo benefício. Em casos específicos, esta relação ocasiona o aparecimento de intoxicações por plantas em animais de produção, relatados no Brasil desde a introdução dos primeiros bovinos. Isto ocorre porque ao sofrer predação pelos animais as plantas desenvolveram mecanismos físicos e químicos de defesa contra a herbivoria. Entre os mecanismos químicos, sintetizados a partir do seu metabolismo secundário, substâncias químicas podem promover quadros de intoxicação em animais que ingerirem estas plantas (BARBOSA et al., 2007). Entre estas substâncias, uma destaca-se por causar quadros de intoxicações que levam o animal ao óbito, é o monofluoroacetato de sódio (MFA). Composto altamente letal, responsável por causar anualmente milhares de mortes de ruminantes no Brasil, ocasionando perdas econômicas significativas. No Brasil, o grupo de plantas tóxicas mais importante é o das plantas que causam morte súbita associada ao exercício, em que o MFA está envolvido (TOKARNIA et al., 2000). Tentativas de controlar esta intoxicação pelos métodos tradicionais, incluindo a utilização de herbicidas, o uso de cercas para isolar as áreas onde as plantas ocorrem, ou eliminação das mesmas por métodos mecânicos não têm apresentado resultado satisfatório. Outra forma de controle desta intoxicação seria através da indução de resistência dos animais a essas plantas. Isto pode ser possível utilizando-se microrganismos que codificam a enzima fluoroacetato dehalogenase, capaz de metabolizar o MFA. Esta tentativa foi realizada na Austrália, através da inoculação no rúmen, de cepas da bactéria Butyrivibrio fibrisolvens modificadas geneticamente com a introdução de um gene, proveniente de uma espécie de Moraxella, que codifica uma dehalogenase, capaz de degradar este composto, reduzindo os sinais clínicos da intoxicação por este composto (GREEG et al. 1998). Esta tese teve o objetivo de isolar e identificar bactérias de solo e de rúmen que degradam fluoroacetato de sódio, bem como estudar a enzima envolvida neste processo. A mesma está dividida em três artigos, formatados de acordo com o que estabelece a NORMA Nº 01/2011 de 03 de junho de 2011 do Programa de Pós-Graduação em Medicina Veterinária da UFCG, Campus de Patos/PB (Anexo 1). O primeiro capítulo é um artigo de pesquisa, publicado na The Scientific World Journal, que aborda o isolamento de bactérias de solo e plantas capazes de degradar MFA. O segundo capítulo é uma pesquisa publicada na mesma revista, e descreve o isolamento de duas bactérias de rúmen caprino, capazes de 9 degradar MFA. O terceiro capítulo refere-se a uma análise computacional de sequências enzimáticas disponíveis no banco de dados do NCBI, para caracterização catalítica como fluoroacetato dehalogenase, ao apresentarem conservados tríade catalítica e sítio ativo. REFERÊNCIAS Barbosa, R. R.; Ribeiro Filho, M. R.; Silva, I. P. & Soto-Blanco, B. Plantas tóxicas de interesse pecuário: importância e formas de estudo. Acta Veterinaria Brasílica, v.1, n.1, p.1-7, 2007. Gregg K., hamdorf b., Henderson K., Kopency J., Wong C. Genetically modified ruminal bacteria protect sheep from fluoroacetato poisoning. Applied and Environmental Microbiology. v. 64, n. 9, p. 3496-3498, 1998. Tokarnia C.H., Döbereiner J. & Peixoto P.V. 2000. Plantas Tóxicas do Brasil. Helianthus, Rio de Janeiro. 310p. 10 CAPÍTULO I Defluoração de fluoroacetato de sódio por bactérias de solo e plantas no Brasil (Defluorination of sodium fluoroacetate by bacteria from soil and plants in Brazil) Publicado na ―The Scientific World Journal‖ (anexo 2), de acordo com o que estabelece a Norma nº 01/2011 de 03 de junho de 2011, do Programa de Pós-Graduação em Medicina Veterinária da UFCG, Campus de Patos/PB. 11 Defluoração de fluoroacetato de sódio por bactérias de solo e plantas no Brasil Expedito K. A. Camboim1, Michelle Z. Tadra-Sfeir2, Emanuel M. de Souza2, Fabio de O. Pedrosa2, Paulo P. Andrade3, Chris S. McSweeney4, Franklin Riet-Correa1 e Marcia A. Melo1 1 Unidade Acadêmica de Medicina Veterinária, Universidade Federal de Campina Grande, Patos, Paraíba, Brasil. 2 Laboratório de Fixação Biológica de Nitrogênio, Departamento de Bioquímica e Biologia Molecular, Universidade Federal do Paraná, Curitiba, Paraná, Brasil. 3 Universidade Federal de Pernambuco, Departamento de Genética, Recife, Pernambuco, Brasil. 4 CSIRO Livestock Industries, Queensland Bioscience Precinct, St Lucia, Qld, Austrália Autor para correspondência Marcia A. Melo, Unidade Acadêmica de Medicina Veterinária, Universidade Federal de Campina Grande. Av. Universitária, s/n, Bairro Sta. Cecília, Patos, Paraíba, Brasil. CP: 64, CEP: 58708-110. e-mail: [email protected] RESUMO O objetivo deste trabalho foi isolar e identificar bactérias capazes de degradar fluoroacetato de sódio (FS) em amostras de solo e plantas, coletadas em áreas onde são encontradas plantas que contêm fluoroacetato tais como Mascagnia rigida e Palicourea aenofusca. As amostras foram cultivadas em meio mineral acrescido de 20 mmol L −1 de fluoroacetato de sódio. Através do sequenciamento do gene 16S rRNA os sete isolados foram identificados como Paenibacillus sp. (ECPB01), Burkholderia sp. (ECPB02), Cupriavidus sp. (ECPB03), Staphylococcus sp. (ECPB04), Ancylobacter sp. (ECPB05), Ralstonia sp. (ECPB06), e Stenotrophomonas sp. (ECPB07). Todos os sete isolados degradaram o FS contido no meio, alcançando uma taxa de degradação de 20 mmol L −1 de íon flúor. Dos 12 isolados, seis são descritos pela primeira vez como capazes de degradar FS. No futuro, alguns destes microorganismos podem ser utilizados para estabelecer no rúmen uma população bacteriana capaz de degradar o fluoroacetato de sódio e proteger ruminantes de intoxicações por este composto. INTRODUÇÃO No Brasil, treze espécies de plantas que causam morte súbita associada a esforço físico são responsáveis por cerca de 500.000 mortes de bovinos por ano: Palicourea marcgravii, P. aeneofusca, P. juruana, P. grandiflora, Pseudocalymma elegans, Arrabidaea bilabiata, A. japurensis, Mascagnia rigida, M. elegans, M. pubiflora, M. aff. rigida, M. exotropia, e M. sepium [1, 2]. O Fluoroacetato de sódio (FS) foi identificado como o princípio ativo de P. maracgravii [3] e A. bilabiata [4] e provavelmente está presente em outras plantas destes gêneros. Ele atua interrompendo o ciclo do ácido tricarboxílico (TCA), sendo primeiro convertido a fluorcitrato que inibe a enzima aconitase e succinato desidrogenase, resultando em acúmulo de citrato nos tecidos e plasma e, finalmente, causando privação de energia e morte animal [5]. O uso de FS é proibido no Brasil, ocorrendo exclusivamente como produto natural em plantas. Na Austrália, o composto é usado em iscas impregnadas para o controle de coelhos, raposas, cachorro do mato e outras populações de mamíferos; contudo, quando introduzido no ambiente pode selecionar microorganismos capazes de degradar FS [6]. A degradação microbiológica do fluoroacetato de sódio é catalisada pela haloacetato halidohidrolase, a qual é capaz de quebrar a forte ligação do carbono-flúor [7]. Vinte e quatro microorganismos que degradam FS foram isolados de solo na Austrália Central, pertencentes a sete gêneros bacterianos (Acinetobacter, Arthrobacter, Aureobacterium, Bacillus, Pseudomonas, Weeksella, e Streptomyces) e quatro gêneros de fungos (Aspergillus, Fusarium, Cryptococcus e Penicillium) [8]. A possibilidade para prevenir intoxicações por fluoroacetato em ruminantes através da inoculação ruminal de bactérias geneticamente modificadas, contendo um gene que codifica uma fluoroacetato dehalogenase vem sendo investigada [9, 10]. Uma pesquisa aprofundada em amostras retiradas do ambiente, tais como solo, folhas e conteúdo do trato digestivo de herbívoros que estão em contato com o FS, seria importante para verificar a 13 diversidade e ocorrência de microorganismos capazes de degradar esta toxina. No futuro, genes que codificam enzimas provenientes destes microorganismos podem ser utilizados em engenharia genética para modificar bactérias que colonizam o rúmen para que se tornem capazes de degradar FS de plantas tóxicas, protegendo os animais da intoxicação por este composto. Este estudo objetivou isolar e identificar bactérias capazes de degradar o fluoroacetato de sódio proveniente de amostras de solo e plantas coletadas no Estado da Paraíba, Brasil. MATERIAL E MÉTODOS Coleta das amostras Amostras de solo e plantas foram coletadas no Estado da Paraíba, Brasil, em áreas onde Mascagnia rigida e Palicourea aenofusca estavam presentes. As amostras de solo foram coletadas na base das plantas, 1 a 8 cm de profundidade. Folhas e flores também foram coletadas. As amostras foram colocadas individualmente em tubos tipo Falcon de 50 mL e enviadas ao laboratório sob refrigeração para cultivo imediato das bactérias associadas. Isolamento bacteriano O isolamento bacteriano foi realizado em tubos tipo Falcon de 50 mL em meio mineral (Brunner), acrescido (http://www.dsmz.de/microorganisms/medium/pdf/DSMZ com Medium457.pdf) vitaminas e 20 milimoles (mmol) L−1 de fluoroacetato de sódio (Sigma-Fluka) como única fonte de carbono. Este meio será aqui designado de meio Brunner. As amostras foram incubadas a 28°C em um agitador orbital. Após 48 horas, um (1) mL do cultivo primário foi transferido para tubos de ensaio, contendo nove (9) mL de meio Brunner e incubados sob as mesmas condições descritas acima. A defluoração do FS foi mensurada com eletrodo seletivo para F− (Thermo Electron Corporation) em placas de 24 poços, contendo 500 μL de cultura e 500 μL de Total Ionic Strength Adjustment Buffer-TISAB (diaminociclohaxano, cloreto de sódio e ácido acético glacial, pH 5.5). A liberação de íon flúor proveniente da degradação 14 microbiana do fluoroacetato de sódio foi expressa em milimoles. A taxa de defluoração de 20 mmol L−1 de FS corresponde a liberação de 20 mmol L−1 F−. As amostras que apresentaram defluoração do FS foram cultivadas em diluições seriadas de 10−1 a 10−9. Para obter colônias puras, a maior diluição que apresentou defluoração do FS foi plaqueada em Ágar Brunner (meio Brunner acrescido com 1% de Ágar) e incubada a 28°C por 72 horas. Subsequentemente, cada colônia foi usada para inocular três tubos de ensaio contendo nove (9) mL de meio Brunner, que foram monitorados quanto à defluoração do FS. Pseudomonas fluorescens (DSM 8341) foi utilizada como controle positivo para atividade da fluoroacetato dehalogenase. Nove mL de meio Brunner foram incubados sem inóculo bacteriano, sob as mesmas condições, para avaliar a estabilidade da degradação do FS. A amostra padrão (Pseudomonas fluorescens DSM 8341) e as bactérias isoladas de solo e plantas foram cultivadas em meio Brunner em concentrações crescentes de FS (20 mmol L−1, 40 mmol L−1, 60 mmol L−1 e 80 mmol L−1 a 200 mmol L−1) para avaliar a maior taxa de defluoração. Adicionalmente, para avaliar a defluoração na presença de outras fontes de carbono, a amostra padrão e as bactérias isoladas de solo e plantas foram também cultivadas em meio Brunner enriquecido com extrato de levedura e glicose, sob as seguintes concentrações: (1) somente meio Brunner; (2) meio suplementado com extrato de levedura 0.01% e glicose 2%; (3) meio com extrato de levedura 0.01%; (4) meio com glicose 2%. Identificação da sequência do gene 16S rRNA As bactérias que apresentaram atividade de defluoração foram identificadas por amplificação do gene 16S rRNA pela Reação em Cadeia pela Polimerase (PCR) e posterior sequenciamento. A extração de DNA foi realizada com Brazol® (LGC Biotecnologia) de acordo com as especificações do fabricante. A amplificação do gene 16S rRNA foi efetuada em tampão contendo 0.5 µM dos primers 27F e 1492R [11], 2U de Taq DNA polimerase, 0.2 mM de dNTP, 100 ng de DNA e água ultra pura para volume final de 20 µL. No controle negativo, o volume de DNA foi substituído por água ultra-pura. Os produtos de amplificação foram aplicados em gel de agarose 1% e submetidos à eletroforese. O DNA foi corado com brometo de etídio e as bandas visualizadas em capturador de imagem (UVP- BioImaging Systems). 15 A reação de sequenciamento foi realizada utilizando-se kit BigDye (Applied Biosystems), de acordo com as recomendações do fabricante, e os produtos sequenciados no sequenciador da Genetic Analyzer 3500 XL (Applied Biosystems). Análise das sequências e filogenia As sequências do gene 16S rRNA foram montadas com o programa CAP3 (http://pbil.univ-lyon1.fr/cap3.php). As sequências de DNA foram analisadas através do Basic Local Alignment Search Tool (BLAST) disponível no website do National Center for Biotechnology Information (NCBI - www.ncbi.nlm.nih.gov / BLAST). A identificação das espécies foi baseada no escore máximo, na identidade e no valor de cobertura. O banco de dados do Greengenes e workbench (http://greengenes.lbl.gov) foi utilizado para corroborar com a identificação das espécies. A árvore filogenética foi gerada com o programa MEGA 5 usando os parâmetros padrões (http://www.megasoftware.net/mega.php) [12, 13]. RESULTADOS Após a análise das sequências dos genes 16S rRNA, sete isolados foram identificados como Paenibacillus sp. (ECPB01), Burkholderia sp. (ECPB02), Cupriavidus sp. (ECPB03), Staphylococcus sp. (ECPB04), Ancylobacter sp. (ECPB05), Ralstonia sp. (ECPB06) e Stenotrophomonas sp. (ECPB07) (Tabela 1). Cupriavidus sp. e Ralstonia sp. foram isolados em amostras de solo e plantas, enquanto Paenibacillus sp., Burkholderia sp. e Ancylobacter sp. foram encontrados em amostras de solo e, Staphylococcus sp. e Stenotrophomonas sp. foram isolados de amostras de plantas. 16 TABELA 1. Resultados do BLAST para as sequências do gene 16S rRNA obtidas de bactérias isoladas de amostras de solo e plantas. Código do isolado Espécie mais similar Origem do isolado Tamanho Cobertura Escore Valor de Identidade sequência (%) Máximo cobertura (%) 16S rRNA ECPB01 Paenibacillus sp. Solo 1425 99 2619 0.0 99 ECPB02 Burkholderia sp. Solo 1398 99 2553 0.0 99 ECPB03 Cupriavidus sp. Solo e Planta 1398 99 2540 0.0 99 ECPB04 Staphylococcus sp. Planta 1402 100 2569 0.0 99 ECPB05 Ancylobacter sp. Solo 1368 99 2435 0.0 99 ECPB06 Ralstonia sp. Solo e Planta 1407 99 2551 0.0 99 Stenotrophomonas Planta 1417 99 2606 0.0 sp. * Gêneros foram identificados baseados sobre o escore máximo, identidade e cobertura. ECPB07 Objetivando um suporte adicional à atribuição de gênero que foi baseada no escore máximo, na identidade e na cobertura do BLAST, as sequências foram previamente aparadas para ter o mesmo tamanho e foi construída uma árvore filogenética, identificando-se que cinco isolados eram filogeneticamente relacionados (Figura 1): Ancylobacter sp., três Burkholderiaceae (Cupriavidus sp., Ralstonia sp. e Burkholderia sp.) e Stenotrophomonas sp. Duas outras espécies, Paenibacillus sp. e Staphylococcus sp. formam um grupo heterogêneo. 99 17 FIGURA 1. Árvore filogenética baseada em sequências 16S rRNA pelo método de máxima parcimônia. ECPB01 a ECPB07 representam os códigos dos isolados e Methanobacterium sp. MO-MB1 (gi|311141366|dbj|AB598270.1|) o grupo externo. A história evolucionária foi inferida utilizando o método de Máxima parcimônia. A árvore consenso do bootstrap foi inferida a partir de 1000 repetições e representa a história evolutiva do táxon examinado. Ramos com valores de bootstrap abaixo de 50% foram recolhidos. O percentual de árvores de consenso nos quais os táxons se agruparam no bootstrap (1000 réplicas) é mostrado perto dos ramos. A árvore foi obtida utilizando o método Close-Neighbor-Interchange com algoritmo de busca nível 1, no qual as árvores iniciais foram obtidas mediante a adição aleatória de sequências (10 réplicas). A análise utilizou oito (8) sequências de nucleotídeos. As posições com espaços ou dados perdidos foram eliminadas. Foram consideradas um total de 1235 posições no conjunto de dados final. A análise evolutiva foi realizada pelo método MEGA5. Todos os isolados bacterianos apresentaram atividade de degradação do FS, alcançando um nível de liberação de 20 mmol L−1 de íon flúor, após 32 horas de incubação em meio Brunner, contendo 20 mmol L−1 de FS (Figura 2). O mesmo resultado foi observado com a amostra controle de Pseudomonas fluorescens (DSM 8341). Não houve liberação de íon flúor quando o meio Brunner foi incubado na ausência de bactérias. 18 FIGURA 2. Taxa de degradação do fluoroacetato de sódio por bactérias isoladas de amostras de solo e plantas no Estado da Paraíba, Brasil. Os símbolos são como se segue. O círculo branco: ECPB01, o triângulo preto: ECPB02, o quadrado preto: ECPB03, o círculo cinza: ECPB04, o quadrado cinza: ECPB05, o triângulo cinza: ECPB06, o círculo preto: ECPB07. Quando as amostras foram cultivadas em diferentes concentrações de FS, a taxa máxima de defluoração foi de 140 mmol L−1 F− em 140 mmol L−1 de concentração. Nas maiores concentrações de FS (160 mmol L−1, 180 mmol L−1 e 200 mmol L−1), o nível de degradação permaneceu em 140 mmol L−1. Quando os isolados foram cultivados em meio Brunner acrescido de outras fontes de carbono (extrato de levedura 0,01% e glicose 2%), um intenso crescimento bacteriano foi observado após 24 horas, porém a degradação de FS apenas alcançou 20 mmol L−1 F− entre 48 a 64 horas com 20 mmol L−1 de FS na concentração inicial. 19 DISCUSSÃO Ancylobacter sp., as Burkholderiaceaes (Cupriavidus sp., Ralstonia sp. e Burkholderia sp.) e Stenotrophomonas sp. pertencem às classes de Alfa, Beta e Gama proteobacteria, respectivamente. As duas outras espécies, Staphylococcus famílias sp., são das Paenibacillaceae Paenibacillus sp. e e Staphylococcaceae, respectivamente, formando um grupo heterogêneo; a atribuição do gênero baseada sobre os valores do BLAST foi coerente com a árvore filogenética, que está de acordo com a filogenia destas bactérias. A existência de uma atividade similar de haloácida dehalogenase entre microorganismos distantemente relacionados, isolados em uma área restrita, sugere uma pressão seletiva comum e efetiva, atuando sobre um amplo conjunto de microorganismos de solo. As sequências genômicas das espécies Burkholderia sp., Cupriavidus taiwanensis, Staphylococcus saprophyticus, Paenibacillus sp., Ralstonia sp. e Stenotrophomonas maltophilia estavam disponíveis no banco de dados de genoma do NCBI. O genoma de Ancylobacter dichloromethanicus ainda não foi sequenciado, porém, há uma sequência de 16S rRNA para esta espécie depositada no Genbank. Dos gêneros listados acima, a Burkholderia sp. era a única para qual já havia citação sobre a fluoroacetato dehalogenase (Fac-Dex FA1) [14]. Para Cupriavidus sp., Staphylococcus sp., Paenibacillus sp., Ralstonia sp., Stenotrophomonas sp. e Ancylobacter sp. as dehalogenases estavam anotadas como proteína com domínio de hidrolase dehalogenase haloácida [15, 16], hidrolase tipo dehalogenase haloácida [17, 18], 2-haloalcano dehalogenase [17, 18], haloacetato dehalogenase [18, 19], e diclorometane dehalogenase [20]. Contudo, quando os sete isolados foram cultivados na presença de FS o substrato foi degradado. Este achado pode ser explicado pela inespecificidade das dehalogenases, que podem usar como substrato outros compostos estruturalmente similares ao FS, processo conhecido como adaptação cruzada [21]. Liu et al. [22] verificaram que a fluoroacetato dehalogenase degrada outros compostos halogenados como cloroacetato, bromoacetato, iodoacetato e dicloroacetato. Resultados similares foram obtidos por Donnelly e Murphy [23], que encontraram atividade da fluoroacetato dehalogenase sob cloroacetato, bromoacetato e etil fluoroacetato. O FS também pode ser degradado pela L-2 haloácida delalogenase [24]. Outra possibilidade é a transferência lateral de genes que codificam fluoroacetato dehalogenase. 20 Embora outras bactérias ambientais como Moraxella sp. [25], Acinetobacter, Arthrobacter, Aureobacterium, Bacillus, Pseudomonas, Weeksella e Streptomyces [8] também apresentem atividade de fluoroacetato dehalogenase, não existem relatos anteriores sobre a referida atividade de microorganismos pertencentes a qualquer das seis espécies descritas aqui: Cupriavidus sp., Staphylococcus sp., Paenibacillus sp., Ralstonia sp., Stenotrophomonas sp. e Ancylobacter sp., para os quais os genes de haloácida dehalogenases foram anotados nos genomas das cinco primeiras espécies. Por isso, é sensato assumir que todos os sete isolados têm os genes de fluoroacetato em seus cromossomos e não em plasmídeos, o que é um suporte adicional da existência de uma pressão ambiental seletiva forte, durável e comum sobre estes organismos. Em todos os isolados ocorreu defluoração após 32 horas de cultivo, com a liberação de 20 mmol L−1 de íon flúor na presença de 20 mmol L −1 de FS. Estes resultados foram similares aos relatados por Davis et al. [26] com Burkholderia sp., que também degradou FS em 32 horas, liberando 20 mmol L−1 F− com 20 mmol L−1 de FS. Os cultivos dos isolados em diferentes concentrações de FS indicaram que a maior taxa de degradação foi 140 mmol L−1 F− com 140 mmol L−1 de substrato. Para as maiores concentrações de FS a taxa de defluoração não aumentou, o que pode ser devido à exaustão dos outros nutrientes. Quando as bactérias foram cultivadas em meio Brunner acrescido de extrato de levedura e glicose, a atividade da dehalogenase foi tardia, provavelmente devido à disponibilidade de outras fontes de energia e a difícil quebra da forte ligação carbono-flúor do fluoroacetato [7]. Em conclusão, sete bactérias capazes de degradar o fluoroacetato foram isoladas de solo e plantas, das quais seis não foram previamente descritas como capazes de degradar o FS. A possibilidade de usar algumas destas bactérias para estabelecer no rúmen, uma população bacteriana capaz de degradar o fluoroacetato e proteger ruminantes de intoxicações por este composto, deve ser explorada no futuro. AGRADECIMENTOS Este trabalho foi apoiado pelo INCT para o controle de plantas tóxicas. Projeto número: 573534/2008-0. 21 REFERÊNCIAS [1] C. H. Tokarnia, P. V. Paixoto, and J. Dobereiner, ―Toxic plants affecting heart function of cattle in Brazil,‖ Pesquisa Veterinária Brasileira, vol. 10, pp. 1–10, 1990. [2] S.V. Schons, T. L. Mello, F. Riet-Correa, and A. L. Schild, ―Poisoning by amorimia (Mascagnia) sepium in sheep in Northern Brazil,‖ Toxicon, vol. 57, pp. 781–786, 2011. [3] M. M. Oliveira, ―Chromatographic isolation of monofluoroacetic acid from Palicourea marcgravii St. Hil,‖ Experientia, vol. 19, no. 11, pp. 586–587, 1963. [4] H. C. Krebs, W. Kemmerling, and G. Habermehl, ―Qualitative and quantitative determination of fluoroacetic acid in Arrabidea bilabiata and Palicourea marcgravii by 19F-NMR spectroscopy,‖ Toxicon, vol. 32, no. 8, pp. 909–913, 1994. [5] P. A. Mayes, ―O ciclo do ácido cítrico: o catabolismo da acetilcoA,‖ in Harper: Bioquímica, K. R. Murray et al., Ed., pp. 182–189, Atheneu, São Paulo, Brazil, 9th edition, 2002. [6] L. E. Twigg and D. R. King, ―The impact of fluoroacetate-bearing vegetation on native Australian fauna: a review,‖ Oikos, vol. 61, no. 3, pp. 412–430, 1991. [7] S. Fetzner and F. Lingens, ―Bacterial dehalogenases: biochemistry, genetics, and biotechnological applications,‖ Microbiological Reviews, vol. 58, no. 4, pp. 641–685, 1994. [8] L. E. Twigg and L. V. Socha, ―Defluorination of sodium monofluoroacetate by soil microorganisms from central Australia,‖ Soil Biology and Biochemistry, vol. 33, no. 2, pp. 227–234, 2001. [9] K. Gregg, C. L. Cooper, D. J. Schafer et al., ―Detoxification of the plant toxin fluoroacetate by a genetically modified rumen bacterium,‖ Nature, vol. 12, no. 13, pp. 1361–1365, 1994. [10] K. Gregg, B. Hamdorf, K. Henderson, J. Kopecny, and C. Wong, ―Genetically modified ruminal bacteria protect sheep from fluoroacetate poisoning,‖ Applied and Environmental Microbiology, vol. 64, no. 9, pp. 3496–3498, 1998. 22 [11] W. G. Weisburg, S. M. Barns, D. A. Pelletier, and D. J. Lane, ―16S ribosomal DNA amplification for phylogenetic study,‖ Journal of Bacteriology, vol. 173, no. 2, pp. 697– 703, 1991. [12] J. Felsenstein, ―Confidence limits on phylogenies: an approach using the bootstrap,‖ Evolution, vol. 39, pp. 783–791, 1985. [13] K. Tamura, D. Peterson, N. Peterson, G. Stecher, M. Nei, and S. Kumar, ―MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods,‖ Molecular Biology and Evolution, vol. 28, no. 10, pp. 2731–2739, 2011. [14] T. Kurihara, T. Yamauchi, S. Ichiyama, H. Takahata, and N. Esaki, ―Purification, characterization, and gene cloning of a novel fluoroacetate dehalogenase from Burkholderia sp. FA1,‖ Journal of Molecular Catalysis B, vol. 23, pp. 347–355, 2003. [15] S. Lucas, A. Copeland, A. Lapidus et al., ―Paenibacillus sp. Y412MC10, complete genome,‖ 2011, http://www.ncbi.nlm.nih.gov/. [16] S. Lucas, A. Copeland, A. Lapidus et al., ―Stenotrophomonas maltophilia R551-3, complete genome,‖ 2011, http://www.ncbi.nlm.nih.gov/. [17] M. Kuroda, A. Yamashita, H. Hirakawa et al., ―Whole genome sequence of Staphylococcus saprophyticus reveals the pathogenesis of uncomplicated urinary tract infection,‖ Proceedings of the National Academy of Sciences of the United States of America, vol. 102, no. 37, pp. 13272–13277, 2005. [18] C. Amadou, G. Pascal, S. Mangenot et al., ―Genome sequence of the beta-rhizobium Cupriavidus taiwanensis and comparative genomics of rhizobia,‖ Genome Research, vol. 18, pp. 1472–1483, 2008. [19] D. Ward, A. Earl, M. Feldgarden et al., ―The genome sequence of Ralstonia sp. strain 5 7 47FAA,‖ 2011, http://www.ncbi.nlm.nih.gov/. [20] J. E. Firsova, N. V. Doronina, E. Lang, C. Spröer, S. Vuilleumier, and Y. A. Trotsenko, ―Ancylobacter dichloromethanicus sp. nov.—a new aerobic facultatively methylotrophic bacterium utilizing dichloromethane,‖ Microbiology, vol. 32, no. 4, pp. 227–232, 2009. Systematic and Applied 23 [21] I. S. Melo and J. L. Azevedo, ―Como isolar microrganismos degradadores de moléculas xenobióoticas,‖ in Microbiologia Ambiental, I. S. Melo and J. L. Azevedo, Eds., pp. 167–183, EMBRAPA-CNPMA, Jaguariuna, Brazil, 1997. [22] J. Q. Liu, T. Kurihara, S. Ichiyama et al., ―Reaction mechanism of fluoroacetate dehalogenase from Moraxella sp. B,‖ Journal of Biological Chemistry, vol. 273, no. 47, pp. 30897–30902, 1998. [23] C. Donnelly and C.D. Murphy, ―Purification and properties of fluoroacetate dehalogenase from Pseudomonas fluorescens DSM 8341,‖ Biotechnology Letters, vol. 31, pp. 245–250, 2009. [24] W. Y. Chan, M. Wong, J. Guthrie et al., ―Sequence- and activity-based screening of microbial genomes for novel dehalogenases,‖ Microbial Biotechnology, vol. 3, no. 1, pp. 107–120, 2010. [25] H. Kawasaki, K. Tsuda, I.Matsushita, and K. Tonomura, ―Lack of homology between two haloacetate dehalogenase genes encoded on a plasmid from Moraxella sp. strain B,‖ Journal of General Microbiology, vol. 138, no. 7, pp. 1317–1323, 1992. [26] C. K. Davis, S. E. Denman, L. I. Sly, and C. S. Mcsweeney, ―Development of a colorimetric colony-screening assay for detection of defluorination by micro-organisms,‖ Letters in Applied Microbiology, vol. 53, no. 4, pp. 417–423, 2011. 24 CAPÍTULO II Isolamento de bactérias que degradam fluoroacetato de sódio de amostras de rúmen caprino no Brasil (Isolation of sodium fluoroacetate degrading bacteria from caprine rumen in Brazil) Publicado na ―The Scientific World Journal‖ (anexo 3), de acordo com o que estabelece a Norma nº 01/2011 de 03 de junho de 2011, do Programa de Pós-Graduação em Medicina Veterinária da UFCG, Campus de Patos/PB. 25 Isolamento de bactérias que degradam fluoroacetato de sódio de amostras de rúmen caprino no Brasil Expedito K. A. Camboim1, Arthur P. Almeida1, Michelle Z. Tadra-Sfeir2, Felício Garino Jr1, Paulo P. Andrade3, Chris S. McSweeney4, Marcia A. Melo 1 e Franklin Riet-Correa1 1 Unidade Acadêmica de Medicina Veterinária, Universidade Federal de Campina Grande, Patos, Paraíba, Brasil. 2 Laboratório de Fixação Biológica de Nitrogênio, Departamento de Bioquímica e Biologia Molecular, Universidade Federal do Paraná. 3 Universidade Federal de Pernambuco, Departamento de Genética, Recife, Pernambuco, Brasil. 4 CSIRO Livestock Industries, Queensland Bioscience Precinct, St Lucia, Qld, Australia. Autor para Correspondência Marcia A. Melo, Unidade Acadêmica de Medicina Veterinária, Universidade Federal de Campina Grande. Av. Universitária, s/n, Bairro Sta. Cecília, Patos, Paraíba, Brasil. CP: 64, CEP: 58708-110. E-mail: [email protected] RESUMO O objetivo deste artigo foi descrever o isolamento de duas bactérias que degradam fluoroacetato de sódio de rúmen caprino. Os animais foram caprinos adultos, machos, mestiços, com fistula ruminal, alimentados com feno e pastagem nativa. O líquido ruminal foi obtido através das fístulas ruminais e 100 μL foi inoculado imediatamente em meio mineral acrescido de 20 mmol L−1 de fluoroacetato de sódio (FS), incubados a 39° C em agitador orbital. Pseudomonas fluorescens (DSM 8341) foi utilizada como controle positivo para a atividade da fluoroacetato dehalogenase. Dois isolados foram identificados por sequenciamento do gene 16S rRNA como sendo Pigmentiphaga kullae (ECPB08) e Ancylobacter dichloromethanicus (ECPB09). Estas bactérias degradaram FS, liberando 20 26 mmol L−1 de íon flúor, após 32 horas de incubação em meio Brunner, contendo 20 mmol L−1 de fluoroacetato de sódio. Não há relatos anteriores da atividade de fluoroacetato dehalogenase em P. kullae e A. dichloromethanicus. Medidas de controle para prevenir intoxicação por plantas, incluindo uso de cercas, herbicidas ou outros métodos de eliminação das plantas tóxicas, foram mal sucedidas para evitar intoxicações por plantas contendo fluoroacetato no Brasil. Desta forma, P. kullae e A. dichloromethanicus podem ser utilizadas para colonizar o rúmen de animais susceptíveis para evitar a intoxicação por plantas que contem fluoroacetato. INTRODUÇÃO No Brasil, o grupo de plantas tóxicas mais importante é o que causa ―morte súbita‖ durante esforço físico, responsável por cerca de 500.000 mortes de bovinos anualmente, e composto por 13 espécies de plantas, pertencentes a 3 famílias: Rubiaceae (Palicourea marcgravii, P. aeneofusca, P. juruana e P. grandiflora); Bignoniaceae (Pseudocalymma elegans, Arrabidaea bilabiata e A. japurensis); e Malpighiaceae (Mascagnia rigida, M. elegans, M. pubiflora, M. aff. rigida, M. exotropica e M. sepium) [1–10]. P. marcgravii é a planta tóxica mais importante do Brasil e M. rigida é a mais importante no Nordeste do Brasil [11]. O fluoroacetato de sódio foi identificado como princípio ativo em P. marcgravii, A. bilabiata [12, 13] e M. rigida [14]. Nas outras plantas o princípio ativo ainda não foi identificado, porém é provável que também seja fluoroacetato de sódio [15], que atua interrompendo o ciclo do ácido tricarboxílico, sendo primeiro convertido a fluorcitrato, que por sua vez inibe a enzima aconitase, resultando em acumulo de citrato nos tecidos e plasma e finalmente causando privação de energia e morte animal [16]. A degradação microbiológica do fluoroacetato de sódio é catalisada pela fluoroacetato dehalogenase, que cliva a forte ligação do carbono-flúor [17], mas também cliva, embora de forma menos eficiente, outras ligações tais como carbono-cloro, carbono-bromo e carbono-iodo [18]. Fluoroacetato de sódio também pode ser defluorado pela L-2 haloácida dehalogenases [19]. Várias bactérias de solo têm a capacidade de degradar o fluoroacetato de sódio. Na Austrália Central, foram isolados sete gêneros bacterianos que degradam o fluoroacetato, incluindo Acinetobacter, Arthrobacter, Aureobacterium, Bacillus, Pseudomonas, Weeksella e Streptomyces [20]. Recentemente, foi descrito o isolamento de sete estirpes 27 bacterianas isoladas de amostras de solo no Brasil, pertencentes aos gêneros Ancylobacter, Burkholderia, Cupriavidus, Paenibacillus, Staphylococcus, Stenotrophomonas e Ralstonia [21]. Contudo, um único isolado bacteriano ruminal, pertencente ao filo Synergistetes, foi relatado como sendo capaz de degradar o fluoroacetato de sódio [22, 23]. Como resultados de nossa tentativa para verificar a ocorrência natural de microorganismos que degradam fluoroacetato existentes no sistema digestivo de animais, relatamos aqui o isolamento de duas novas bactérias que degradam o fluoroacetato de amostras de rúmen caprino. MATERIAL E MÉTODOS Coleta e processamento das amostras Os animais foram caprinos adultos, machos, mestiços, com fistula ruminal, alimentados com feno e pastagem nativa e água ad libitum. Os animais foram criados no Hospital Veterinário da Universidade de Campina Grande, Nordeste do Brasil, e não tinham acesso a áreas com plantas tóxicas contendo fluoroacetato. O líquido ruminal foi obtido através das fístulas ruminais e inoculado imediatamente em tubos de ensaio. Isolamento bacteriano O isolamento bacteriano iniciou pela inoculação de 100 μL de fluido ruminal em tubos contendo 9 mL em meio mineral (Brunner) acrescido com vitaminas (http://www.dsmz.de/microorganisms/medium/pdf/DSMZ Medium457.pdf) e 20 mmol L−1 de fluoroacetato de sódio (FS) (Sigma-Fluka) como única fonte de carbono. Este meio será aqui designado de meio Brunner. As amostras foram incubadas a 39°C em um agitador orbital. Após 48 horas, 1 mL do cultivo primário foi transferido para tubos de ensaio, contendo 9 mL de meio Brunner e incubados sob as mesmas condições, descritas acima. A defluoração do FS foi mensurada com eletrodo seletivo para F− (Thermo Electron Corporation) em placas de 24 poços, contendo 500 μL de cultura e 500 μL de Total Ionic Strength Adjustment Buffer-TISAB (diaminociclohexano, cloreto de sódio e ácido acético glacial, pH 5.5). A liberação de íon flúor proveniente da degradação microbiana do fluoroacetato de sódio foi expressa em milimoles (mmol), a taxa de defluoração de 20 mmol L−1 de FS corresponde a liberação de 20 mmol L−1 F−. 28 As amostras que apresentaram defluoração do FS foram cultivadas em diluições seriadas de 10−1 a 10−9. Para obter colônias puras a maior diluição que apresentou defluoração do FS foi plaqueada em Agar Brunner (meio Brunner acrescido com 1% de Agar) e incubada a 39°C por 72 horas. Subsequentemente, cada colônia foi usada para inocular três tubos de ensaio contendo 9 (nove) mL de meio Brunner, que foram monitoradas quanto a defluoração do FS. Pseudomonas fluorescens (DSM 8341) foi utilizada como controle positivo para atividade da fluoroacetato dehalogenase, cultivada sob as mesmas condições, exceto a incubação que foi a 28°C [24]. Nove mL de meio Brunner sem inóculo bacteriano foram incubados sob as mesmas condições para avaliar a estabilidade da degradação do FS. Identificação da sequência do gene 16S rRNA A identificação do gênero bacteriano que apresentou atividade de defluoração foi realizada através da amplificação do gene 16S rRNA pela Reação em Cadeia da Polimerase (PCR) e posterior sequenciamento. A extração de DNA foi realizada com Brazol® (LGC Biotecnologia) de acordo com as especificações do fabricante. A amplificação do gene 16S rRNA foi realizada em tampão contendo 0.5 µM de 27F e 1492R dos primers universais [25], 2U de Taq DNA polimerase, 0.2 mM de dNTP e 100 ng de DNA e água ultra pura para volume final de 20 µL. No controle negativo, o volume de DNA foi substituído por água ultra pura. Os produtos de amplificação foram aplicados em gel de agarose 1% e submetidos a eletroforese. O DNA foi corado com brometo de etídio e as bandas visualizadas em capturador de imagem (UVP- BioImaging Systems). O sequenciamento foi realizado com kit BigDye de acordo com as recomendações do fabricante (Applied Biosystems) e os produtos foram sequenciados no sequenciador da Genetic Analyzer 3500 XL (Applied Biosystems). Análise das sequências e filogenia As sequências do gene 16S rRNA foram montadas com auxílio do programa CAP3 (http://pbil.univ-lyon1.fr/cap3.php). As sequências de DNA foram analisadas através do Basic Local Alignment Search Tool (BLAST) disponível no website do National Center for Biotechnology Information (NCBI - www.ncbi.nlm.nih.gov / BLAST). A identificação das espécies foi baseada no escore máximo, identidade e valor de cobertura. Os dados do Greengenes e workbench foram usados para corroborar com a identificação das espécies (http://greengenes.lbl.gov). A árvore filogenética foi gerada com o programa MEGA 5 29 usando o método estatístico neighbor-joining (http://www.megasoftware.net/mega.php) [26]. RESULTADOS E DISCUSSÃO As sequências dos genes 16S rRNA dos dois isolados que degradam fluoroacetato ECPB08 (JQ345720) e ECPB09 (JQ345721) foram similares aos genes 16S rRNA das espécies Pigmentiphaga e Ancylobacter, respectivamente (Tabela 1). Com base apenas no valor do escore, a sequência de P. kullae foi a mais similar (escore = 2237) a ECPB08, embora outras sequências de 16S rRNA tiveram os mesmos valores nos parâmetros cobertura e identidade. Similarmente, as sequências de Ancylobacter vacuolatus e Ancylobacter sp. WPCB135 foram mais similares (escore 2388) a ECPB09, mas as diferenças nos parâmetros de alinhamento para outras sequências de 16S rRNA de Ancylobacter foram apenas marginais. Consequentemente, não foi possível inferir a partir dos valores do escore, quais espécies seriam filogeneticamente mais próximas a ECPB09 (JQ345721). 30 Tabela 1: Resultados do alinhamento local das sequências bacterianas de 16S rRNA dos isolados que degradam fluoroacetato. As sequências foram comparadas aquelas depositadas no National Center for Biotechnology Information, utilizando o algoritmo regular BlastN disponível em http://www.ncbi.nlm.nih.gov/BLAST. Os organismos com sequências similares aos isolados ECPB08 (JQ345720) e ECPB09 (JQ345721) estão listadas na segunda coluna. Os valores dos parâmetros de escore total, cobertura e identidade máxima estão apresentados nas colunas 3 a 5. O código de acesso ao GenBank está apresentado em parênteses. ECPB09 (JQ345721) ECPB08 (JQ345720) Isolado (Acc.nr) Espécies/linhagens (nº de acesso) Escore Cobertura Identidade (%) máx. (%) total Pigmentiphaga kullae strain K24 (NR_025112.1) 2237 100 98 Pigmentiphaga daeguensis strain K110 (NR_044082.1) 2215 100 98 Pigmentiphaga daeguensis strain NML080357 (JN585327.1) 2197 100 97 Pigmentiphaga litoralis strain JSM 061001 (NR_044530.1) 2152 99 97 Ancylobacter sp. WPCB135 (FJ006900.1) 2388 99 99 Ancylobacter vacuolatus strain DSM 1277 (NR_042794.1) 2388 99 99 Ancylobacter dichloromethanicus strain DM16 (EU589386.1) 2387 99 99 Ancylobacter polymorphus strain DSM 2457 (NR_042795.1) 2372 99 99 Ancylobacter rudongensis strain AS1.1761 (NR_029047.1) 2370 99 99 Ancylobacter polymorphus T10AII (GQ921957.1) 2358 98 99 Ancylobacter aquaticus (NR_044737.1) 2351 99 98 Ancylobacter oerskovii strain NS05 (NR_042655.1) 2341 99 98 A análise comparativa das sequências usando o alinhamento global confirmou que os isolados ECPB08 (JQ345720) e ECPB09 (JQ345721) são filogeneticamente relacionados com os gêneros Pigmentiphaga e Ancylobacter, respectivamente (Figura 1). Na verdade, o alinhamento global corrobora com a inferência taxonômica com base nos resultados do alinhamento 16S rRNA para ECPB08 (JQ345720), indicando que o isolado está relacionado com a P. kullae K24 (Figura 1a), e está apoiado pelos resultados anteriores sobre a taxonomia deste gênero [27, 28]. Contudo, o alinhamento global permitiu uma melhor estimativa da posição taxonômica para ECPB09 (JQ345721), sugerindo uma estreita relação com A. polymorphus (Figura 1b). Embora a parte superior 31 da árvore na Figura 1b apresente baixos valores de bootstrap, a totalidade do dendrograma é consistente com os resultados apresentados por Firsova et al. [29] para o gênero Ancylobacter. Figura 1: Árvore filogenética baseada nas sequências do gene do 16S rRNA por análise de Máxima Parcimônia. ECPB08 (JQ345720) e ECPB09 (JQ345721) são os códigos dos isolados. Em parênteses estão os códigos do GenBank. É apresentada a relação entre ECPB08 (JQ345720) (a), ECPB09 (JQ345721) (b), os táxons relacionados e os grupos externos Achromobacter ruhlandii, A. denitrificans e Starkeya novella. A árvore de consenso bootstrap inferida a partir de 1000 repetições mostra a história evolutiva dos táxons analisados. Escala de 0,002: distância evolutiva. O gênero Pigmentiphaga foi inicialmente proposto por Blümel et al. [30] com Pigmentiphaga kullae (K24) como única espécie, que degrada compostos xenobióticos. Em 2007, Yoon et al. [31] descreveram um novo membro do gênero, Pigmentiphaga daeguensis ( K110T), isolado de águas residuais coletadas de tinturarias na Coréia. Mais tarde, Chen et al. [27] e Lee et al. [28] isolaram Pigmentiphaga litoralis ( JSM 061001) e P. soli de amostras de planície de maré no mar da China e de solo na Coréia do Sul, respectivamente. 32 Raj e Maloy [32] propuseram a substituição no nome do gênero Microcyclus por Ancylobacter, com uma única espécie, A. aquaticus [33]. Desde então, cinco novas espécies foram descritas, a última sendo Ancylobacter dichloromethanicus (DM16), isolada de solo contaminado [29]. Esta espécie usa os compostos diclorometano, metanol, formiato e policarbonato de formaldeído como fontes de carbono e energia. Análises enzimáticas mostraram que a presença de diclorometano dehalogenase dependente de GSH (glutationa). Ambos ECPB08 (JQ345720) e ECPB09 (JQ345721) cresceram em meio de cultura contendo FS como única fonte de carbono. Estas bactérias apresentaram atividade de degradação do FS, liberando 20 mmol L−1 de íon flúor após 32 horas de incubação em meio Brunner com 20 mmol L−1 de FS (Figura 2). Estes resultados são similares aos relatados por Davis et al. [34] para Burkholderia sp. A amostra controle Pseudomonas fluorescens (DSM 8341) alcançou o mesmo nível de defluoração e não houve liberação de íon flúor quando o meio Brunner foi incubado sem bactéria, devido à estabilidade da forte ligação carbono-flúor do fluoroacetato [17]. Não há relatos anteriores sobre a atividade de fluoroacetato dehalogenase para as espécies de Pigmentiphaga ou Ancylobacter. Concentração de flúor (mM) 20 15 10 5 0 0 8 16 24 Horas após incubação 32 Figura 2: Taxa de degradação de fluoroacetato de sódio por bactérias isoladas de rúmen caprino. Isolado ECPB08 (quadrado cinza claro), isolado ECPB09 (losango preto), e P. fluorescens (triângulo cinza escuro, controle positivo). 33 A capacidade de defluoração de ambos isolados pode ser devida à expressão de um gene que codifica uma fluoroacetato dehalogenase ou outra dehalogenase inespecífica. Embora não tenha sido descrito nenhum gene de dehalogenase para Pigmentiphaga, está depositada no GenBank uma sequência de diclorometano dehalogenase para A. dichloromethanicus [29]. Liu et al. [35] confirmaram que a fluoroacetato dehalogenase degrada outros compostos halogenados, tais como cloroacetato, bromoacetato, iodoacetato e dicloroacetato, por um mecanismo de adaptação cruzada [36]. Resultados semelhantes foram obtidos por Donnelly e Murphy [24] que observaram capacidade da fluoroacetato dehalogenase para degradar cloroacetato, bromoacetato e etil fluoroacetato. Alternativamente, o fluoroacetato de sódio foi defluorado pela L-2 haloácida dehalogenase [18]. Bactérias que degradam fluoroacetato de sódio, principalmente espécies de Bacillus e Flavobacterium, foram isoladas em amostras de solo na Austrália, mesmo na ausência de fluoroacetato no ambiente [37]. Neste estudo, duas espécies Pigmentiphaga e Ancylobacter foram isoladas do rúmen caprino, apesar dos animais não serem alimentados com plantas que produzam esse composto. Este achado sugere que o gene que codifica a fluoroacetato dehalogenase é ubíquo e que sua expressão pode representar vantagem seletiva para microorganismos, pela possibilidade da enzima degradar outros compostos, como mencionado antes. Por outro lado, tem sido relatado que animais que pastam em áreas onde Mascagnia rigida está presente são mais resistentes à intoxicação do que aqueles que não têm contato com a planta [6]. Resultados recentes de nosso grupo de pesquisa demonstram que a resistência a intoxicação por M. rigida pode ser induzida através de administrações sucessivas de doses não tóxicas da planta ou por transfaunação da fauna ruminal de animais resistentes para susceptíveis (dados não publicados). Estes resultados sugerem que tanto as alterações na microflora ruminal ou níveis sustentados de expressão de gene que codifique uma fluoroacetato dehalogenase da microbiota endógena do rúmen são responsáveis por esta tolerância. Medidas de controle para prevenir intoxicações por plantas, incluindo uso de cercas, herbicidas ou outros métodos de eliminação das plantas, foram mal sucedidas para evitar intoxicações por plantas que contem fluoroacetato no Brasil [38]. Uma forma alternativa pode ser através da detoxificação microbiana de plantas pela inoculação no rúmen de bactérias que degradam fluoroacetato. Esta estratégia foi utilizada por Gregg et al. [39] que inoculou no rúmen uma estirpe geneticamente modificada de Butyrivibrio 34 fibrisolvens com um gene que codifica uma fluoroacetato dehalogenase proveniente de uma espécie de Moraxella, que foi eficiente em prevenir a intoxicação por fluoroacetato em ovino. Numa abordagem alternativa, os dois isolados, Pigmentiphaga sp. e Ancylobacter sp. são candidatos para serem utilizados na colonização do rúmen para evitar intoxicação por plantas que contem fluoroacetato. AGRADECIMENTOS Este trabalho foi apoiado pelo INCT para o controle de plantas tóxicas. Projeto número: 573534/2008-0. 35 REFERÊNCIAS [1] A. Gava, J. Cristani, J. V. Branco, D. S. Neves, A. J.Mondadori, and R. S. Sousa, ―Sudden death in cattle by Mascagnia sp. (Malpighiaceae) in the State of Santa Catarina, Brazil,‖ Pesquisa Veterinaria Brasileira, vol. 18, no. 1, pp. 16–20, 1998. [2] M. A. Helayel, T. N. Franc¸a, J. N. Seixas, V. A. Nogueira, S. A. Caldas, and P. V. Peixoto, ―Sudden death in cattle caused by ingestion of Pseudocalymma elegans (Bignoniaceae) in the county of Rio Bonito, Rio de Janeiro,‖ Pesquisa Veterinaria Brasileira, vol. 29, no. 7, pp. 498–508, 2009. [3] R. M. T. Medeiros, S. A. Geraldo Neto, R. C. Barbosa, E. F. Lima, and F. Riet-Correa, ―Sudden bovine death from Mascagnia rigida in Northeastern Brazil,‖ Veterinary and Human Toxicology, vol. 44, no. 5, pp. 286–288, 2002. [4] S. P. Pavarini, M. P. Soares, P. M. Bandarra et al., ―Sudden death in cattle due to the consumption of Amorimia exotropica (Malpighiaceae) in Rio Grande do Sul, Brazil,‖ Pesquisa Veterinaria Brasileira, vol. 31, no. 4, pp. 291–296, 2011. [5] B. Soto-Blanco, M. Haraguchi, J. A. Silva, and Górniak, ―Intoxicação natural de caprinos e ovinos por Palicourea marcgravii St. Hil. (Rubiaceae),‖ Caatinga, no. 1, pp. 52–56, 2004. [6] I. P. Silva, R. A. Lira, R. R. Barbosa, J. S. Batista, and B. Soto-Blanco, ―Intoxicação natural pelas folhas de Mascagnia rigida (Malpighiaceae) em ovinos,‖ Arquivos do Instituto Biologico, vol. 75, no. 2, pp. 229–233, 2008. [7] S. V. Schons, T. L. deMello, F. Riet-Correa, and A. L. Schild, ―Poisoning by Amorimia (Mascagnia) sepium in sheep in Northern Brazil,‖ Toxicon, vol. 57, no. 5, pp. 781–786, 2011. [8] C. H. Tokarnia, J. D¨obereiner, and P. V. Peixoto, Plantas Tóxicas do Brasil, Editora Helianthus, Rio de Janeiro, Brazil, 2000. [9] J. S. Vasconcelos, F. Riet-Correa, A. F. M. Dantas et al., ―Poisoning by Mascagnia rigida (Malpighiaceae) in sheep and goats,‖ Pesquisa Veterinária Brasileira, vol. 28, no. 10, pp. 521–526, 2008. [10] J. S. Vasconcelos, F. Riet-Correa, A. F. M. Dantas, R. M. T. Medeiros, and A. J. A. Dantas, ―Sudden deaths caused by Palicourea aeneofusca (Rubiaceae) and Mascagnia rigida (Malpighiaceae) in cattle in the Zona da Mata of Paraíba,‖ Pesquisa Veterinária Brasileira, vol. 28, no. 10, pp. 457–460, 2008. [11] C. H. Tokarnia, P. V. Peixoto, and J. Dobereiner, ―Poisonous plants affecting heart funtions of cattle in Brazil,‖ Pesquisa Agropecuária Brasileira, vol. 10, pp. 1–10, 1990. [12] M. M. de Oliveira, ―Chromatographic isolation of monofluoroacetic acid from Palicourea marcgravii St. Hil,‖ Experientia, vol. 19, no. 11, pp. 586–587, 1963. [13] H. C. Krebs, W. Kemmerling, and G. Habermehl, ―Qualitative and quantitative determination of fluoroacetic acid in Arrabidea bilabiata and Palicourea marcgravii by 19F-NMR spectroscopy,‖ Toxicon, vol. 32, no. 8, pp. 909–913, 1994. 36 [14] L. C. Cunha, S. L. Gorniak, M. Haraguchi, F. Riet-Correa, F. G. Xavier, and J. C. Florio, ―Palicourea marcgravii e Mascagnia rigida: um estudo por cromatografia em camada delgada (CCD),‖ in Proceedings of the II Simpósio de Pós-Graduação and XV Semana Científica, Prof. Dr. Benjamin Eurico Malucelli, São Paulo, Brazil, 2006. [15] F. Riet-Correa and M. C. Méndez, ―Intoxicações por plantas e micotoxinas,‖ in Doenças de Ruminantes e Eqüídeos, F. Riet-Correa, A. L. Schild, R. A. A. Lemos, and J. R. J. Borges, Eds., vol. 2, Editora Pallotti, Santa Maria, RS, Brasil, 2007. [16] P. A. Mayes, ―O ciclo do ácido cítrico: o catabolismo da AcetilcoA,‖ in Harper: Bioquímica, K. R. Murray, Ed., pp. 182–189, Atheneu, São Paulo, Brazil, 9th edition, 2002. [17] S. Fetzner and F. Lingens, ―Bacterial dehalogenases: biochemistry, genetics, and biotechnological applications,‖ Microbiological Reviews, vol. 58, no. 4, pp. 641–685, 1994. [18] T. Kurihara, T. Yamauchi, S. Ichiyama, H. Takahata, and N. Esaki, ―Purification, characterization, and gene cloning of a novel fluoroacetate dehalogenase from Burkholderia sp. FA1,‖ Journal of Molecular Catalysis B, vol. 23, no. 2–6, pp. 347–355, 2003. [19] W. Y. Chan, M. Wong, J. Guthrie et al., ―Sequence- and activity-based screening of microbial genomes for novel dehalogenases,‖Microbial Biotechnology, vol. 3, no. 1, pp. 107–120, 2010. [20] L. E. Twigg and L. V. Socha, ―Defluorination of sodium monofluoroacetate by soil microorganisms from central Australia,‖ Soil Biology and Biochemistry, vol. 33, no. 2, pp. 227–234, 2001. [21] E. K. A. Camboim, M. Z. Tadra-Sfeir, E. M. Souza et al., ―Defluorination of sodium fluoroacetate by bacteria from soil and plants in Brazil,‖ The Scientific World Journal, vol. 2012, Article ID 149893, 5 pages, 2012. [22] C. Davis, Investigations on the microbial degradation of fluoroacetate [Ph.D. thesis], School of Chemistry and Molecular Bioscience. The University of Queensland, Australia, 2011. [23] L. E. X. Leong, S. E. Denman, C. K. Davis, T. Huber, and C. S. McSweeney, ―Peptide utilization of the novel fluoroacetate degrading ruminal bacterium,‖ Proceedings of the Australian Society of Animal Production, vol. 28, p. 64, 2010. [24] C. Donnelly and C. D. Murphy, ―Purification and properties of fluoroacetate dehalogenase from Pseudomonas fluorescens DSM 8341,‖ Biotechnology Letters, vol. 31, no. 2, pp. 245–250, 2009. [25] W. G. Weisburg, S. M. Barns, D. A. Pelletier, and D. J. Lane, ―16S ribosomal DNA amplification for phylogenetic study,‖ Journal of Bacteriology, vol. 173, no. 2, pp. 697– 703, 1991. [26] K. Tamura, D. Peterson, N. Peterson, G. Stecher, M. Nei, and S. Kumar, ―MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods,‖ Molecular Biology and Evolution, vol. 28, no. 10, pp. 2731–2739, 2011. 37 [27] Y. G. Chen, Y. Q. Zhang, K. Huang et al., ―Pigmentiphaga litoralis sp. nov., a facultatively anaerobic bacterium isolated from a tidal flat sediment,‖ International Journal of Systematic and Evolutionary Microbiology, vol. 59, no. 3, pp. 521–525, 2009. [28] J. J. Lee, S. Srinivasan, and M. K. Kim, ―Pigmentiphaga soli sp. nov., a bacterium isolated from soil,‖ J. Microbiol, vol. 49, no. 5, pp. 857–861, 2011. [29] J. Firsova, N. Doronina, E. Lang, C. Spröer, S. Vuilleumier, and Y. Trotsenko, ―Ancylobacter dichloromethanicus sp. nov.- a new aerobic facultatively methylotrophic bacterium utilizing dichloromethane,‖ Systematic and Applied Microbiology, vol. 32, no. 4, pp. 227–232, 2009. [30] S. Blümel, B. Mark, H. J. Busse, P. Kämpfer, and A. Stolz, ―Pigmentiphaga kullae gen. nov., sp. nov., a novel member of the family Alcaligenaceae with the ability to decolorize azo dyes aerobically,‖ International Journal of Systematic and EvolutionaryMicrobiology, vol. 51, no. 5, pp. 1867–1871, 2001. [31] J. H. Yoon, S. J. Kang,W. Kim, and T. K. Oh, ―Pigmentiphaga daeguensis sp. nov., isolated from wastewater of a dye works, and emended description of the genus Pigmentiphaga,‖ International Journal of Systematic and Evolutionary Microbiology, vol. 57, no. 6, pp. 1188–1191, 2007. [32] H. D. Raj and S. R. Maloy, ―Family Spirosomaceae: gramnegative ring-forming aerobic bacteria,‖ Critical Reviews in Microbiology, vol. 17, no. 5, pp. 329–364, 1990. [33] J. Orskov, ―Beschreibung eines neuen Microben, Microcyclus aquaticus, mit eigentiimlicher Morphologie,‖ Zentralblatt fur Bakteriologie, Parasitenkunde, Infektionskrankheiten und Hygiene, vol. 107, pp. 180–184, 1923. [34] C. K. Davis, S. E. Denman, L. I. Sly, and C. S. McSweeney, ―Development of a colorimetric colony-screening assay for detection of defluorination by micro-organisms,‖ Applied Microbiology, vol. 53, no. 4, pp. 417–423, 2011. [35] J. Q. Liu, T. Kurihara, S. Ichiyama et al., ―Reaction mechanism of fluoroacetate dehalogenase from Moraxella sp. B,‖ Journal of Biological Chemistry, vol. 273, no. 47, pp. 30897–30902, 1998. [36] I. S. Melo and J. L. Azevedo, ―Como isolar microrganismos degradadores de moléculas xenobióticas,‖ in Microbiologia Ambiental, I. S. Melo and L. J. Azevedo, Eds., pp. 167–183, EMBRAPA-CNPMA, Jaguariuna, Brazil, 1997. [37] D. H. Wong, W. E. Kirkpatrick, D. R. King, and J. E. Kinnear, ―Defluorination of sodium monofluoroacetate (1080) by microorganisms isolated from western Australian soils,‖ Soil Biology and Biochemistry, vol. 24, no. 9, pp. 833–838, 1992. [38] F. Riet-Correa and R. M. T. Medeiros, ―Intoxicações por plantas em ruminantes no Brasil e no Uruguai: importância econômica, controle e riscos para a saúde pública,‖ Pesquisa Veterinária Brasileira, vol. 21, no. 1, pp. 38–42, 2001. [39] K. Gregg, B. Hamdorf, K. Henderson, J. Kopecny, and C. Wong, ―Genetically modified ruminal bacteria protect sheep from fluoroacetate poisoning,‖ Applied and Environmental Microbiology, vol. 64, no. 9, pp. 3496–3498, 1998. 38 CAPÍTULO III Ubiquidade da fluoroacetato dehalogenase em bactérias: uma abordagem in silico (Ubiquity of fluoroacetate dehalogenase in bacteria: an in silico approach) Artigo submetido para publicação na revista PLOS ONE, de acordo com o que estabelece a Norma nº 01/2011 de 03 de junho de 2011, do Programa de Pós-Graduação em Medicina Veterinária da UFCG, Campus de Patos/PB. 39 Ubiquidade da fluoroacetato dehalogenase em bactérias: uma abordagem in silico Expedito K. A. Camboim1, Antonio F. M. Vaz1, Tereza E. F. Rotondano1, Paulo P. Andrade2, Franklin Riet-Correa1 e Marcia A. Melo1 1 Unidade Acadêmica de Medicina Veterinária, Universidade Federal de Campina Grande, Patos, Paraíba, Brasil. 2 Universidade Federal de Pernambuco, Departamento de Genética, Recife, Pernambuco, Brasil. Autor para Correspondência: Marcia A. Melo, Unidade Acadêmica de Medicina Veterinária, Universidade Federal de Campina Grande. Av. Universitária, s/n, Bairro Sta. Cecília, Patos, Paraíba, Brasil. CP: 64, CEP: 58708-110. E-mail: [email protected] 40 RESUMO Monofluoroacetato de sódio (MFA) é detectado em plantas tóxicas que causam morte súbita em animais. Este composto é catalisado pela fluoroacetato dehalogenase, que cliva a ligação carbono-flúor. Até o momento, existem apenas cinco descrições destas enzimas disponíveis no banco de dados do NCBI. Por isso, realizamos uma análise comparativa de sequências enzimáticas, utilizando como critério de seleção a conservação da tríade catalítica, sítio ativo e motivos estruturais da fluoroacetato dehalogenase. De um total de 549 sequências proteicas obtidas do NCBI, 128 continham a tríade catalítica, o sítio ativo e motivo estrutural encontrados na fluoroacetato dehalogenase de Burkholderia sp. (gi|95102016), Micromonospora lupini str. lupac 08 (gi|386690726), Rhodopseudomonas palustris (gi|81829712) e Moraxella sp. (gi|216773), sugerindo que todas são fluoroacetato dehalogenase pela presença do mecanismo catalítico e estrutural único para defluoração do monofluoroacetato de sódio. A identificação de 128 sequências representa um aumento de 26 vezes no número de fluoroacetato dehalogenase previamente descritas, refletindo um mecanismo evolucionário comum; as plantas sintetizam monofluoroacetato de sódio como mecanismo químico de defesa, o que pode favorecer a ocorrência de um mecanismo coevolutivo entre plantas e a flora bacteriana associada. 41 ABSTRACT Sodium monofluoroacetate (MFA) is present in some poisonous plants that cause sudden death in animals. This compound is catalyzed by a fluoroacetate dehalogenase, which cleaves the carbon-fluorine bond. To date, there are only five descriptions of this enzyme available at the National Center for Biotechnology Information (NCBI) database. Therefore, we performed a comparative analysis of enzymatic sequences, using as criterion for sequence selection the fluoroacetate dehalogenase catalytic triad conservation, active site and structural motifs. From a total of 549 protein sequences selected from NCBI, 128 contained the catalytic triad, the active site and the structural motif found in the fluoroacetate dehalogenases of Burkholderia sp. (gi|95102016), Micromonospora lupini str. lupac 08 (gi|386690726), Rhodopseudomonas palustris (gi|81829712) and Moraxella sp. (gi|216773), suggesting that all are fluoroacetate dehalogenases, presenting a single structural and catalytic mechanism for sodium monofluoroacetate defluorination. The identification of 128 sequences represents a 26-fold increase in the amount of fluoroacetate dehalogenase sequences previously described, suggesting the existence of a common evolutionary mechanism; moreover, when plants synthesize MFA as a chemical defense mechanism, they may also elicit a co-evolutionary mechanism involving plants and associated bacterial flora. INTRODUÇÃO Os compostos orgânicos halogenados constituem um dos maiores grupos de poluentes ambientais, como resultado do uso disseminado de herbicidas, inseticidas, fungicidas, solventes, fluídos hidráulicos e de transferência de calor, plastificantes e intermediários para síntese de químicos [1]. Estes compostos também podem ser detectados em plantas tóxicas, sob a forma de monofluoroacetato de sódio (MFA) [2-5], que causa morte súbita durante esforço físico em animais após ingestão [6-9]. Anualmente, aproximadamente 500.000 mortes de bovinos ocorrem no Brasil consequente da ingestão de plantas que contem MFA [10]. A produção de MFA por plantas como proteção contra pressão de pastejo dos herbívoros e o desenvolvimento de resistência animal a toxina são 42 provavelmente eventos co-evolutivos [11] e a resistência animal vem sendo comumente associada a bactérias ruminais que degradam MFA [12]. Muitos compostos halogenados de plantas tóxicas são degradados por microrganismos, os quais usam as enzimas dehalogenases para catalisar compostos orgânicos halogenados, por clivagem da ligação do carbono-halogênio, sendo fundamentais para a detoxificação e biodegradação destes compostos [13]. Estas enzimas são proteínas citosólicas que usam água como único co-substrato e geralmente não requerem cofatores [1,13]. A degradação microbiana de MFA é catalisada pela fluoroacetato dehalogenase, que cliva a ligação carbono-flúor [1], mas também cliva, embora com menor eficiência, outras ligações tais como carbono-cloro, carbono-bromo e carbono-iodo [14]. O mecanismo de reação de Burkholderia sp. FAc-DEX FA1 foi descrito por Jitsumori et al. [15] baseado em experimentos de mutagêneses, que propôs que a tríade catalítica é constituída por Asp104, His271 e Asp128. Inicialmente, o grupo carboxilato nucleofílico de Asp104 ataca o átomo de carbono de MFA para deslocar o átomo de flúor, levando à formação de um éster intermediário. No segundo passo, o éster intermediário é hidrolisado por uma molécula ativada pela His272, que produz glicolato e regenera o grupo carboxilato de Asp104 [16]. Adicionalmente, Chan et al. [13] propôs que poucos resíduos do sítio ativo são considerados para caracterizar a atividade da fluoroacetato dehalogenase. Estes resíduos compreendem um aspartato (Asp-104) atuando como nucleófilo e duas argininas (Arg) na posição nucleófilo + 1 e nucleófilo + 4. Até o momento existem apenas cinco descrições de bactérias que tem fluoroacetato dehalogenase: Moraxella sp. [16], Burkholderia sp. [14], Pseudomonas fluorescens DSM 8341 [17], Micromonospora lupini str. lupac 08 [18] e Rhodopseudomonas palustris [19]. Até o momento, uma busca exaustiva por genomas de bactérias com genes que codificam fluoroacetato dehalogenase ainda não foi realizada. Como novas bactérias isoladas de solo e de rúmen caprino apresentando capacidade de degradar MFA foram recentemente descritas [20,21], faz-se necessário realizar uma análise comparativa de sequências enzimáticas disponíveis no banco de dados do NCBI, explorando a conservação da tríade catalítica, sítio ativo e motivo estrutural da fluoroacetato dehalogenase. Nossos resultados mostraram que os genes para fluoroacetato dehalogenase estão presentes de forma muito mais ampla em bactérias do que previamente suspeitava-se e possuem uma alta conservação. 43 METERIAL E MÉTODOS Identificação e Analise das Sequências de Fluoroacetato Dehalogenase As sequências de proteínas foram inicialmente selecionadas no site do National Center for Biotechnology Information (NCBI - http://www.ncbi.nlm.nih.gov), usando como palavra-chave haloacetato dehalogenase. As sequências de fluoroacetato dehalogenases de Burkholderia sp. (gi|95102016), Micromonospora lupini str. lupac 08 (gi|386690726), Moraxella sp. (gi|216773) e Rhodopseudomonas palustris (gi|81829712) também foram utilizadas para pesquisa no banco de dados do NCBI através do Blastp (Basic Local Alignment Search Tool - Protein). As sequências encontradas foram armazenadas em formato FASTA para posterior alinhamento no programa MEGA 5.0 (http://www.megasoftware.net/mega.php) [22] pelo ClustalW. A presença dos resíduos Asp104, Hist271 e Asp128 da tríade catalítica, o sítio ativo e o aminoácido triptofano da posição 150 (Trp150) foram os critérios para seleção das sequências, uma vez que estes resíduos determinam a atividade específica da fluoroacetato dehalogenase, de acordo com Liu et al. [16], Jitsumori et al. [15] e Chan et al. [13]. RESULTADOS E DISCUSSÃO Um total de 549 sequências proteicas foi selecionado através da palavra-chave e pela pesquisa do BLAST. Seguindo o alinhamento global e o uso dos critérios estruturais estabelecidos para as prováveis atividades de fluoroacetato dehalogenases, 128 sequências continham a tríade catalítica, o sítio ativo e o motivo estrutural encontrados na fluoroacetato dehalogenase de Burkholderia sp. (gi|95102016), Micromonospora lupini str. lupac 08 (gi|386690726), Rhodopseudomonas palustris (gi|81829712) e Moraxella sp. (gi|216773), sugerindo que todas são fluoroacetato dehalogenase. A maior e menor sequência enzimática contem 354 aminoácidos (Burkholderia dolosa AUO158, gi|254254471) e 262 aminoácidos (Crocosphaera watsonii WH 0003, gi|357266014), respectivamente. Todas as 128 sequências proteicas mantinham conservados os resíduos do sítio ativo, tais como Phe34, Asp104, Arg105, Arg108, Asp128, His271 e Phe272 constituindo o domínio principal e Tyr147, His149, Trp150 e Tyr212 no domínio superficial, de acordo como proposto por Jitsumori et al. [15], baseado na análise por mutagênese da 44 fluoroacetato dehalogenase de Burkholderia sp. Outra importante observação é a presença do Trp150, um importante resíduo envolvido na interação da enzima com o átomo de flúor do MFA, sendo provavelmente um requisito essencial para a redução da energia de ativação e clivagem da ligação carbono-flúor. Esta interação pode também ser necessária para a orientação adequada deste substrato no sítio ativo da fluoroacetato dehalogenase [15]. Alem disso, foi observado uma evidente conservação dos aminoácidos His32, Gly33, Pro35 e Gly106, estes quatro resíduos sendo previamente descritos como motivos estruturais das famílias de haloalcano dehalogenase tipo I e II, esterases e fluoroacetato dehalogenase como proposto por Chan et al. [13]. Das 128 sequências, 20 apresentaram conservação da tríade catalítica e do sítio ativo da fluoroacetato dehalogenase (Tabela 1), como proposto por Liu et al. [16], Jitsumori et al. [15] e Chan et al. [13]. 45 Tabela 1. Alinhamento das sequências de fluoroacetato dehalogenases entre Burkholderia sp. (gi|95102016), Micromonospora lupini str. lupac 08 (gi|386690726), Moraxella sp. (gi|216773) e Rhodopseudomonas palustris (gi|81829712) e as sequências enzimáticas de bactérias que possuem a mesma tríade catalítica (seleção em preto), motivos estruturais (seleção em cinza claro) e confirmado/ provável resíduo de sítio ativo (seleção em cinza escuro). 46 Na base de dados do NCBI, as prováveis funções atribuídas as 20 enzimas que apresentaram sítio ativo conservado foram: alfa/beta hidrolase, proteína do tipo alfa/beta hidrolase, produto do gene DehH1, haloacetato dehalogenase, provável haloacetato dehalogenase h-1, proteína hipotética, provável alfa/beta hidrolase provável haloacetato dehalogenase e provável hidrolase. Essas sequências pertencem a dois filos bacterianos: Actinobacteria e Proteobacteria. O primeiro filo é representado somente pelo gênero Pseudonocardia. Todos os demais gêneros (Acidovorax, Azorhizobium, Azospirillum, Curvibacter, Leptothrix, Maritimibacter, Methylobacterium, Oceanicola, Ralstonia, Ramlibacter, Roseobacter, Sagittula e Xanthobacter) pertencem ao filo Proteobacteria. No caso de Methylobacterium, duas sequências estavam disponíveis, sendo as estirpes M. extorquens DM4 e DSM 13060, ambas com 305 aminoácidos em sua composição, porém divergindo em seis deles, DM4: Ala32, Ser80, Ala208, Val235, Leu256 e Gln258; DSM 13060: Ser32, Arg80, Gly208, Ala235, Phe256 e Arg258. Além disso, duas sequências de organismos não foram identificadas até o nível de gênero: um organismo pertence à família Oxalobacteraceae e outro a ordem Rhodobacterales. Para as demais 108 sequências, listadas na tabela 2, a tríade catalítica também está conservada, entretanto, algumas delas apresentam substituições em aminoácidos do sítio ativo, explicitamente: Phe34, Tyr147, His149 e Phe272. Esses aminoácidos não são parte do consenso para a fluoroacetato dehalogenase proposta por Jitsumori et al. [15]. Além disso, em relação aos aminoácidos propostos por Chan et al. [13] como constituintes do motivo estrutural, a Gly102 por vezes também é substituída. 47 Tabela 2. Alinhamento das sequências de fluoroacetato dehalogenases entre Burkholderia sp. (gi|95102016), Micromonospora lupini str. lupac 08 (gi|386690726), Moraxella sp. (gi|216773) e Rhodopseudomonas palustris (gi|81829712) e as 108 sequências enzimáticas de bactérias que possuem algumas alterações no sítio ativo. Tríade catalítica (seleção em preto), motivos estruturais (seleção em cinza claro) e confirmados/ prováveis resíduo de sítio ativo (seleção em cinza escuro); * indica substituição dos aminoácidos. 48 Tabela 2. Continuação. 49 Tabela 2. Continuação. 50 A tabela 3 apresenta os aminoácidos do sítio ativo que não estavam totalmente conservados entre as 108 sequências enzimáticas, bem como seus respectivos percentuais de substituição. Não foi possível estabelecer um padrão que pudesse sugerir que estas sequências pertencessem a diferentes famílias de dehalogenases, como por exemplo, haloalcano dehalogenase tipo I e II ou esterases. Adicionalmente, o fato que Jitsumori et al. [15] não perceberem que poderia haver substituições de aminoácidos nos resíduos do sítio ativo e nos motivos estruturais pode ser explicado pelo fato de que estes autores usaram apenas uma sequência enzimática para desenvolver seu modelo experimental. 51 TABELA 3. Percentual de substituição dos aminoácidos do sítio ativo das 108 sequências disponíveis no banco de dados de sequências proteicas do NCBI, em relação à fluoroacetato dehalogenase de Burkholderia sp. (gi|95102016). Aminoácidos da enzima de Burkholderia sp. (gi|95102016) Aminoácidos das enzimas analisadas e percentual de substituição Phe34 H (62,04%) Y (28,70%) F (3,70%) N (2,78%) M (1,85%) I (0,93%) Tyr147 Y (92,58%) V (1,84%) A (0,93%) F (0,93%) I (0,93%) K (0,93%) His149 H (92,59%) W (6,48%) V (0,93%) Phe272 Y (69,44%) F (24,07%) S (5,56%) T (0,93%) N (0,93%) T (0,93%) 52 A disponibilidade de 133 sequências de fluoroacetato dehalogenase em bactérias permitirá um estudo mais amplo sobre sua característica catalítica. A identificação de 128 novas prováveis sequências representa um aumento de 26 vezes no número de sequências de fluoroacetato dehalogenase disponíveis no banco de dados do NCBI. Embora não seja suficiente prever completamente a especificidade das prováveis enzimas, cujas sequências estão listadas nas tabelas 1 e 2, baseadas apenas em sua homologia dos aminoácidos com as outras fluoroacetato dehalogenase previamente caracterizadas, nossos dados fortemente sugerem a existência de um mecanismo catalítico e estrutural único para defluoração do MFA. Isso pode refletir um mecanismo evolucionário comum, previamente não descrito. A presença de uma fluoroacetato dehalogenase conservada entre diferentes filos bacterianos, provavelmente está associada à síntese disseminada de metabolitos secundários contendo flúor, objetivando a proteção do hospedeiro contra herbivoria e patógenos [23,24]. De fato, Eason et al. [25] sugere que a capacidade de sintetizar MFA pode ser muito mais frequente entre as plantas do que previamente reconhecida. Isto pode ser uma tendência em plantas altamente tóxicas, enquanto que, os mecanismos de seleção natural podem ocorrer sob uma pressão química muito menor. Por outro lado, as plantas estão sujeitas a uma seleção inversa, o que pode levar a ocorrência de uma co-evolução das plantas e da flora bacteriana associada. 53 REFERÊNCIAS 1. Fetzner S, Lingens F (1994) Bacterial dehalogenases: biochemistry, genetics, and biotechnological applications. Microbiol. Reviews 58: 641-685. 2. Oliveira MM (1963) Chromatographic isolation of monofluoroacetate acid from Palicourea marcgravii St. Hil. Experientia 19: 586. 3. Krebs HC, Kemmerling W, Habermehl G (1994) Qualitative and quantitative determination of fluoroacetic acid in Arrabidaea bilabiata and Palicourea marcgravii by F-NMR spectroscopy. Toxicon 32: 909–913. 4. Cunha LC, Pípole F, Carvalho LR, Lago JHG, Górniak SL (2012) Isolation and characterization of sodium 2-fluoroacetate from Mascagnia rigida using chromatography and infrared spectroscopy. Toxicon 60: 329–332. 5. Lee ST, Cook D, Riet-Correa F, Pfister JA, Anderson WR, Lima FG, Gardner D (2012) Detection of monofluoroacetate in Palicourea and Amorimia species. Toxicon 60: 791-796. 6. Medeiros RMT, Geraldo Neto SA, Barbosa RC, Lima EF, Riet-Correa F 2002 Sudden death caused by Mascagnia rigida in Cattle in Paraíba, Northeastern Brazil. Vet Human Toxicol 44: 286-288. 7. Schons SV, Mello TL, Riet-Correa F, Schild AL (2011) Poisoning by Amorimia (Mascagnia) sepium in sheep in Northern Brazil. Toxicon 57: 781-786. 8. Vasconcelos JS, Riet-Correa F, Dantas AFM, Medeiros RMT, Galiza GJ, Oliveira DM, Pessoa AFA (2008) Intoxicação por Mascagnia rigida (Malpighiaceae) em ovinos e caprinos. Pesq. Vet. Bras. 28: 521-526. 9. Vasconcelos JS, Riet-Correa F, Dantas AFM, Medeiros RMT, Dantas AJ (2008) Mortes súbitas em bovinos causadas por Palicourea aeneofusca (Rubiaceae) e Mascagnia rigida (Malpighiaceae) na Zona da Mata Paraibana. Pesq. Vet. Bras. 28: 457-460. 10. Tokarnia CH, Brito MF, Barbosa JD, Döbereiner J, Peixoto PV (2012) Plantas Tóxicas do Brasil. 2nd ed, Helianthus, Rio de Janeiro. 566p. 11. Mead RJ, Oliver AJ, King DR, Hubach PH (1985) The co-evolutionary role of fluoroacetate in plant-animal interactions in Australia. OIKOS 44: 55-60. 12. Gregg K, Hamdorf B, Henderson K, Kopecny J, Wong C (1998) Genetically modified ruminal bacteria protect sheep from fluoroacetate poisoning. Appl Environ Microbiol 64: 3496-3498. 54 13. Chan WY, Wong M, Guthrie J, Savchenko AV, Yakunin AF, Pai EF, Edwards EA (2010) Sequence and activity based screening of microbial genomes for novel dehalogenases. J Microb Biotechnol 3: 107-120. 14. Kurihara T, Yamauchi T, Ichiyama S, Takahata H, Esaki N (2003) Purification, characterization, and gene cloning of a novel fluoroacetate dehalogenase from Burkholderia sp. FA1. J Mol Catal B: Enzym 23: 347-355. 15. Jitsumori K, Omi R, Kurihara T, Kurata A, Mihara H, Miyahara I, Hirotsu K, Esaki N (2009) X-Ray Crystallographic and Mutational Studies of Fluoroacetate Dehalogenase from Burkholderia sp. Strain FA1. J Bacteriol 191: 2630-2637. 16. Liu JQ, Kurihara T, Ichiyama S, Miyagi M, Tsunasawa S, Kawasaki H, Soda K, Esaki N (1998) Reaction mechanism of fluoroacetate dehalogenase from Moraxella sp.. J Biol Chem 273: 30897-30902. 17. Donnelly C, Murphy CD (2009) Purification and properties of fluoroacetate dehalogenase from Pseudomonas fluorescens DSM 8341. Biotechnol Lett 31: 245– 250. 18. Alonso-Vega NP, Bacigalupe R, Pujic P, Lajus A, Vallenet D, Carro L, Coll P, Trujillo ME (2012) Genome sequence of Micromonospora lupini Lupac 08, isolated from root nodules of Lupinus angustifolius. J Bacteriol 194: 4135. 19. Larimer FW, Chain P, Hauser L, Lamerdin J, Malfatti S, Do L, Land ML, Pelletier DA, Beatty JT, Lang AS, Tabita FR, Gibson JL, Hanson TE, Bobst C, Torres JL, Peres C, Harrison FH, Gibson J, Harwood CSI (2004) Complete genome sequence of the metabolically versatile photosynthetic bacterium Rhodopseudomonas palustris. J Nat Biotechnol 22: 55-61. 20. Camboim EKA, Tadra-Sfeir MZ, Souza EM, Pedrosa FO, Andrade PP, McSweeney CS, Riet-Correa F, Melo MA (2012). Defluorination of sodium fluoroacetate by bacteria from soil and plants in Brazil. Scientific World Journal doi:10.1100/2012/149893. 21. Camboim EKA, Almeida AP, Tadra-Sfeir MZ, Junior, GF, Andrade PP, McSweeney CS, Riet-Correa F, Melo MA (2012). Isolation and Identification of Sodium Fluoroacetate Degrading Bacteria from Caprine Rumen in Brazil. Scientific World Journal doi:10.1100/2012/178254. 22. Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S (2011) MEGA5: Molecular Evolutionary Genetics Analysis using Maximum Likelihood, 55 Evolutionary Distance, and Maximum Parsimony Methods. Mol Biol Evol 28: 2731-2739. 23. Iriti M, Faoro F (2009) Chemical Diversity and Defence Metabolism: How Plants Cope with Pathogens and Ozone Pollution. Int J Mol Sci 10: 3371-3399. 24. King DR, Oliver AJ, Mead RJ (1981) Bettongia and fluoroacetate: a role for 1080 in fauna management. Aust Wildlife Res 8: 529–536. 25. Eason C, Miller A, Ogilvie S, Fairweather A (2011) An updated review of the toxicology and ecotoxicology of sodium fluoroacetate (1080) in relation to its use as a pest control tool in New Zealand. New Zeal J Ecol 35: 1-20. 56 CONSIDERAÇÕES FINAIS Este trabalho foi iniciado em 2008, com o objetivo de obter bactérias com atividade de fluoroacetato dehalogenase para, futuramente, serem utilizadas na produção de inóculos que, uma vez introduzidos no rúmen, hidrolisem o monofluoroacetato de sódio (MFA) contido em plantas tóxicas, protegendo os animais da intoxicação. Nessa época havia duas correntes: a do pesquisador Greeg, que tinha desenvolvido uma bactéria modificada geneticamente, inserindo um gene proveniente de uma espécie de Moraxella, que codifica fluoroacetato dehalogenase na bactéria Butirivibrio fibrisolvins. Experimentalmente, a inoculação desta bactéria modificada foi eficiente na prevenção da intoxicação por MFA em ovinos. A segunda hipótese, do Dr Chris McSweeney (CSIRO, Austrália), colaborador deste projeto, propunha que as bactérias utilizadas deveriam ser bactérias anaeróbicas encontradas no rúmen, que uma vez isoladas poderiam ser multiplicadas para produzir inóculos a serem introduzidos intraruminalmente. Neste trabalho, foram identificadas diversas bactérias aeróbicas capazes de hidrolisar MFA, provenientes de solo, plantas e rúmen caprino. Simultaneamente, trabalhos de nosso grupo de pesquisa, determinaram que a administração de doses não tóxicas de Amorimia (Mascagnia) septentrionalis (tinguí), que contêm MFA, induzem resistência à intoxicação por essa substância. Esses resultados e os resultados dos dois primeiros trabalhos desta tese, sugerem que há normalmente no rúmen, bactérias que hidrolisam MFA, que ao serem expostas ao substrato são induzidas a expressarem genes que codificam uma fluoroacetato dehalogenases, protegendo o animal da intoxicação. Esta hipótese foi verificada no terceiro capítulo desta tese, no qual se observou que a presença destas enzimas em bactérias é bem mais disseminada do que se previa anteriormente. Esses achados abrem caminho para novas hipóteses como, por exemplo, a utilização de MFA em doses não tóxicas e/ou substratos químicos não tóxicos, que induzam a produção destas enzimas pelas bactérias ruminais.
Download