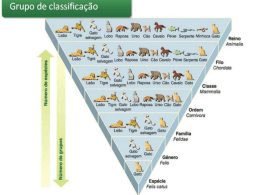
Bioinformática CURSOS EM BIOLOGIA, BIOQUÍMICA, BIOTECNOLOGIA, CIÊNCIAS BIOMÉDICAS E ENGENHARIA BIOLÓGICA João Varela [email protected] Aula T7 Temas da Aula T7 Taxonomia (Annotathon) Ontologia Génica (GO) Conclusões (Annotathon) Taxonomia Uma das funções das anotações (meta)genómicas é a definição do táxon da fonte de material genético Tipos de Classificações Classificação hierárquica (de Lineu) Classificação cladística Classificação Hierárquica de Sistemas Biológicos Domínio Superreino, Reino, Subreino Superfilo, Filo (≈ divisão), Subfilo (Superclasse,) Classe, Subclasse Superordem, Ordem, Subordem (, Infraordem) (Superfamília, Epifamília,) Família, Subfamília (, Tribo, Subtribo, Infratribo) Género, Subgénero Espécie, Subespécie Estirpe (≈ variedade) Clades Clades – ramos monofiléticos de uma árvore filogenética Monofilia, Parafilia e Polifilia Grupo monofilético – grupo taxonómico que provém de um ancestral comum cujos descendentes se encontram incluídos neste grupo (≈ grupo holofilético) Grupo parafilético – grupo taxonómico que provém de um ascendente comum cujos descendentes se encontram parcialmente incluídos neste grupo Grupo polifilético – grupo taxonómico que provém de mais que um ascendente Monofilia, Parafilia e Polifilia Clades NCBI Taxonomy Database Cada táxon tem um nº de identificação - NCBI numerical identifier ou Taxonomy ID Taxonomia: Annotathon http://www.ncbi.nlm.nih.gov/Taxonomy/ Pesquisa por táxon ou identificador numérico Colocar o identificador numérico no campo Taxonomy para o táxon menos abrangente ao qual a sequência deverá pertencer com uma elevada probabilidade (analisar valores E do BLAST e o nó imediatamente superior do ramo à qual pertence a sequência em estudo das árvores filogenéticas obtidas) Raramente se consegue identificar até à espécie a fonte biológica de sequências metagenómicas Temas da Aula T7 Taxonomia (Annotathon) Ontologia Génica (GO) Conclusões (Annotathon) Ontologia Génica (GO) Iniciativa bioinformática (geneontology.org) para definir termos GO em três domínios diferentes: Componente celular (biologia celular) Função molecular / bioquímica (biologia molecular) Processo Biológico (biologia de sistemas) Componente Celular Localização do produto génico dentro e / ou fora da célula A localização é uma pista onde a proteína / RNA actua na célula, que por sua vez é uma pista para a sua função Bioinformática da previsão do tráfico intracelular Tráfego Biossintético Tráfego Retentivo Tráfego Endocítico http://www.ncbi.nlm.nih.gov/books/bv.fcgi?rid=mboc4.figgrp.2303 Vias de tráfego biossintético, endocítico e retentivo Retículo Endoplasmático Rugoso http://www.ncbi.nlm.nih.gov/books/bv.fcgi?rid=mboc4.figgrp.2205 A via biossintético-secretora inicia-se no RER http://www.ncbi.nlm.nih.gov/books/bv.fcgi?rid=mboc4.figgrp.2215 Existência de péptido sinal numa sequência é indicadora de que a proteína é secretada ou está no sistema endomembranar http://www.ncbi.nlm.nih.gov/books/bv.fcgi?rid=mboc4.figgrp.2227 Existência de péptido sinal numa sequência é indicadora de que a proteína é secretada ou está no sistema endomembranar Tráfego Biossintético-Secretor http://www.ncbi.nlm.nih.gov/books/bv.fcgi?rid=mboc4.figgrp.2303 Existência de um sinal de retenção ou domínios transmembranares numa sequência é indicadora de que a proteína é membranar Tráfico intracelular para mitocôndrias e cloroplastos http://www.ncbi.nlm.nih.gov/books/bv.fcgi?highlight=Transport,Proteins,Mitochondria,Chloroplasts&rid=mboc4.section.2176 Proteínas com assinaturas de importação para mitocôndrias deverão ter localização mitocondrial http://www.ncbi.nlm.nih.gov/books/bv.fcgi?rid=mboc4.figgrp.2183 Proteínas com assinaturas de importação para cloroplastos deverão ter localização plastidial http://www.ncbi.nlm.nih.gov/books/bv.fcgi?rid=mboc4.figgrp.2192 Ferramentas bioinformáticas para prever a localização intracelular de proteínas PSORT http://psort.nibb.ac.jp/ SignalP Web Server http://www.cbs.dtu.dk/services/SignalP/ TargetDB http://targetdb.pdb.org/ Ontologia Génica Iniciativa bioinformática (geneontology.org) para definir termos em três domínios diferentes: Componente celular (biologia celular) Função molecular / bioquímica (biologia molecular) Processo Biológico (biologia de sistemas) Biologia molecular de um gene Função bioquímica / molecular de um gene e respectivo produto (RNA e / ou proteína) (por ex., catálise enzimática) Ligação a outras moléculas Exs: lactase, acetil-CoA carboxilase, ligação a DNA Ontologia Génica Iniciativa bioinformática (geneontology.org) para definir termos em três domínios diferentes: Componente celular (biologia celular) Função molecular / bioquímica (biologia molecular) Processo Biológico (biologia de sistemas) Processos biológicos Conjunto de eventos moleculares com princípio e fim bem definidos: Metabolismo das purinas Metabolismo de glícidos Locomoção Fototactismo / Fototaxia Fotossíntese Respiração Etc. Exemplo de termo GO Gene product: Actin, alpha cardiac muscle 1, UniProtKB:P68032 GO term: heart contraction ; GO:0060047 Utilização de termos GO no Annotathon Escolher a função molecular e / ou o processo biológico em que a proteína / RNA participa Para realizar esta anotação verificar quais os termos obtidos no BLAST e InterPro Caso não existem termos GO nas anotações do GenBank, analisar os valores E de sequências homólogas ou domínios funcionais homólogos e atribuir termos GO à sequência e explicar a vossa decisão em RESULT ANALYSIS Annotathon: Análise de Resultados Critérios de Avaliação ORFs Massa Molecular Domínios BLAST e Lineage Report MSA Filogenia e Taxonomia Ontologia Conclusões Critérios de Avaliação: ORFs e Análise de Resultados respectiva Codão START existe? Está correcto? Codão STOP existe? Está correcto? ORF contém codões STOP internos? Discute qual o quadro de leitura e cadeia em que ela se encontra? Existem ORFs maiores? Se sim, discute porque não escolheu essa para ser analisada? Existem ORFs adicionais com significado biológico? Determinou se a sequência é codificante ou não? Disse que era não codificante quando o tamanho da sequência não aponta para isso? Discute quantas ORFs encontrou nas duas cadeias? O protocolo está correcto e completo? Annotathon: Análise de Resultados Critérios de Avaliação ORFs Massa Molecular Domínios BLAST e Lineage Report MSA Filogenia e Taxonomia Ontologia Conclusões Critérios de Avaliação: Massa Molecular Não calculou a massa molecular da proteína quando a proteína está claramente completa? Calculou a massa molecular da proteína quando a proteína está claramente incompleta? Annotathon: Análise de Resultados Critérios de Avaliação ORFs Massa Molecular Domínios BLAST e Lineage Report MSA Filogenia e Taxonomia Ontologia Conclusões Domínios Proteicos e Análise de Resultados Discute a lista de domínios presente em RAW results e respectivas funções? Discute os valores E respectivos? Discute quais os domínios correctos e os domínios redundantes (sobrepostos)? Discute a função da proteína à luz dos domínios encontrados? Os domínios estão correctamente apresentados no esquema gráfico? Colocou os resultados todos em RAW RESULTS? Annotathon: Análise de Resultados Critérios de Avaliação ORFs Massa Molecular Domínios BLAST e Lineage Report MSA Filogenia e Taxonomia Ontologia Conclusões BLAST: Análise de Resultados A lista de resultados (hits) está completa? A lista dos 12 melhores alinhamentos está completa? Tem sequências a mais? Analisa os valores E, o nº de hits e a localização das homologias nos alinhamentos? Analisa a função possível da proteína? Analisa se a proteína tem proteínas homólogas conhecidas? O protocolo está correcto? Desistiu logo com resultados do BLASTp sem ter tentado análises alternativas (por ex., BLASTx)? Discute os resultados do Lineage Report? Discute a escolha dos ingroups e outgroups? Discute a escolha das sequências para fazer os alinhamentos múltiplos? Escolheu o nº máximo de hits correcto? Annotathon: Análise de Resultados Critérios de Avaliação ORFs Massa Molecular Domínios BLAST e Lineage Report MSA Filogenia e Taxonomia Ontologia Conclusões MSA: Análise de Resultados Determinou o início da ORF olhando para os resultados do MSA? Discutiu se as sequências no MSA têm o mesmo tamanho? Os resultados do MSA batem certo com os resultados dos domínios? Isso é discutido? O alinhamento MSA está correcto e bem apresentado (contém nomes fáceis de identificar, por ex.)? O alinhamento MSA contém sequências repetidas? Adicionou a ORF desconhecida ao MSA? (erro frequente!) Annotathon: Análise de Resultados Critérios de Avaliação ORFs Massa Molecular Domínios BLAST e Lineage Report MSA Filogenia e Taxonomia Ontologia Conclusões Filogenia e Taxonomia: Análise de Resultados O protocolo está correcto? Apresentação da árvore está correcta? Colocaram os nomes dos taxa (género, classe, por ex.) nas folhas da árvore? Discutiu a topologia das árvores? Realizou a construção de árvores com os 2 métodos (NJ e ML)? Discutiu se as árvores são concordantes? Seleccionou qual o grupo taxonómico mais provável? Annotathon: Análise de Resultados Critérios de Avaliação ORFs Massa Molecular Domínios BLAST e Lineage Report MSA Filogenia e Taxonomia Ontologia Conclusões Ontologia Génica Escolheu o processo biológico correcto? Escolheu a função molecular correcta? Annotathon: Análise de Resultados Critérios de Avaliação ORFs Massa Molecular Domínios BLAST e Lineage Report MSA Filogenia e Taxonomia Ontologia Conclusões Conclusões Discutir argumentos a favor / contra a hipótese da sequência ser codificante ou não (usar números) Discutir a sua função bioquímica e a sua participação num dado processo biológico Discutir a classificação taxonómica O que se deve EVITAR nas Conclusões A descrição em que botões se clicou A descrição do método utilizado Escrever conclusões mal estruturadas (escrita telegráfica) Pôr “palha” na esperança de obter melhor nota Fazer plágio, copiando e colando descrições de funções de páginas da Internet (implica anulação da anotação!) Não relacionar conclusões dos vários campos da anotação (por ex. Não relacionar os resultados do MSA com os resultados do InterPro) Pôr hipóteses sem as fundamentar (referências bibliográficas e / ou números)
Download