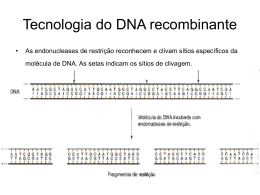
Estrutura dos Ácidos Nucléicos DNA e RNA Replicação – Transcrição – Tradução Introdução à Biologia Molecular Alexandre Havt Moléculas com informações para a síntese de proteínas DNA – armazém ou biblioteca celular Contém informações para a construção das células e tecidos Duplicação dos genes garante a perpetuação das espécies Transcrição – síntese de RNA RNA mensageiro (mRNA) RNA transportador (tRNA) RNA ribossômico (rRNA) Tradução – síntese de proteínas a partir das informações dos RNAs Descoberta da estrutura do DNA em 1953 Watson e Crick Estrutura dos Ácidos Nucléicos – DNA e RNA Estrutura dos Ácidos Nucléicos – DNA e RNA Estrutura dos Ácidos Nucléicos – DNA e RNA Estrutura dos Ácidos Nucléicos - DNA 1 Estrutura dos Ácidos Nucléicos - RNA RNA mensageiro - mRNA RNA ribossômico - rRNA RNA transportador - tRNA Ribossomos Procariotos Citoplasma Mitocôndrias Eucariotos Procariotos 3 tipos de moléculas de rRNA • 16S + 21 proteínas – 30S • 23S +34 proteínas – 50S • 5S Função 70S Citoplasma de eucariotos 4 tipos de moléculas de rRNA • 18S + 33 proteínas – 40S • 5S • 28S +50 proteínas – 60S • 5,8S Mitocondriais 80S Transportar aminoácidos para a síntese protéica Assegurar a incorporação dos mesmos na posição correta • anticódon Células com pelo menos 20 tRNA Pequenas moléculas 80 nucleotídeos 4S 55S • Propriedades similares ao das bactérias Regras da Síntese dos Ácidos Nucléicos e Proteínas Regras da Síntese dos Ácidos Nucléicos e Proteínas Formados por blocos monoméricos RNA e DNA • 5 Nucleotídeos Proteínas • 20 aminoácidos Monômeros adicionados 1 por vez Cadeia de formação tem ponto de partida específico com crescimento unidirecional para um terminal fixo Direção 5´ - 3´ Produto primário é frequentemente modificado 2 Replicação do DNA Duplicação do material genético para a divisão celular Replicação é semiconservativa • Uma fita parental e uma nova fita sintetizada Replicação do DNA Forquilha de replicação Helicases - Separação das duplas fitas Proteínas protegem DNA de fita simples e evitam reaproximação Primase – síntese de iniciador Topoisomerase • Desenrolam supertorção Ligases – união dos fragmentos de Okasaki DNA polimerases • • • • α - associação com primase para formação do iniciador δ - polimerização das fitas líder e lenta γ - polimerização DNA mitocondrial β ε κ η ζ ι - polimerases de reparo Replicação do DNA Eucariótico Síntese de RNA Transcrição Síntese de RNA Transcrição – Síntese de RNA RNA polimerases devem reconhecer O ponto de início da transcrição Qual das fitas deve ser usada como molde – transcrita Região promotora do DNA Controla a ligação da RNA polimerase Identifica o ponto de início da transcrição Controla a frequência da transcrição • Sequências regulatórias no promotor • Sequências perto do promotor • Reforçadores Interagem com proteínas que estabilizam a ligação RNA-polimerase ao promotor 3 Transcrição Processamento do mRNA Eucariótico Síntese dos rRNAs Eucarióticos Síntese dos tRNAs Eucarióticos Síntese Protéica - Tradução Ocorre nos ribossomos – rRNA Síntese de proteínas Tradução Direcionado pela sequência dos códons de mRNA Cada tRNA traz um aminoácido específico ao seu anticódon Aminoacil-tRNA 4 Código Genético Primeira base Segunda base Terceira base (5’) U C A G (3’) U Phe Ser Tyr Cys U Phe Ser Try Cys C Leu Ser Parada Parada A Leu Ser Parada Trp G Leu Pro His Arg U Leu Pro His Arg C Leu Pro Gln Arg A Leu Pro Gln Arg G C A Ile Thr Asn Ser U Ile Thr Asn Ser C Formação do aminoacil-tRNA Aminoacil-tRNA tRNA ligado covalentemente a um aminoácido na terminação 3’ Cada aminoácido com seu tRNA • Alanil-tRNAala • Metionil-tRNAi met 20 Aminoacil-tRNA-sintetases Processo dependente de ATP Ocorre em 2 passos Ile Thr Lys Arg A Met Thr Lys Arg G Val Ala Asp Gly U Val Ala Asp Gly C Val Ala Glu Gly A Hidroxila do carbono 2 ou 3 da ribose Val Ala Glu Gly G Nucleotídeo de adenina (CCA) • Formação do complexo: enzima/aminoacil-AMP e pirofosfato G • Transferência do aminoácido ativo Iniciação Processo da Tradução Envolvimento de 3 passos Iniciação Alongamento Terminação Alongamento – Ligação Peptídica – Translocação Terminação Códons de parada Inexistência de tRNA com anticódons que possam parear com • UAA • UGA • UAG Fatores de liberação unem-se ao ribossomo Hidrólise da ligação peptidiltransferase • Cadeia peptídica • tRNA Peptídeo sintetizado é liberado Dissociação das subunidades do ribossomo • Liberação do mRNA 5 Polissomos Ligação de vários ribossomos ao mRNA Cada ribossomo cobre 80 nucleotídeos de mRNA Aproximadamente um ribossomo a cada 100 nucleotídeos Técnicas de Biologia Molecular Reação de Polimerase em Cadeia – PCR Função - Multiplicação de trecho específico de DNA • Problema – análise de ácidos nucleicos Karis Mulis Baixa quantidade de ácidos nucléicos nos tecidos Primers Taq – DNA polimerase (Thermus aquaticus) • Enzima termoestável em temperatura até 117 °C • Temperatura ótima 72 °C Início – vários banhos maria com diferentes temperaturas 1987 – publicação do artigo de Mulis na Methods in Enzymology 1989 – primeiro termociclador 1994 – Nobel em química para Karis Mulis Técnicas de Biologia Molecular Técnicas de Biologia Molecular Fundamentos do PCR Desnaturação das cadeias de DNA à 94 °C Anelamento dos primers (de acordo com a TM de cada primer) Extensão – incorporação dos nucleotídeos pela enzima Taq á 72 °C Vídeo da reação de PCR Reagentes essenciais Taq – em solução de Tris-HCL (pH 8,0), 0,1 mM EDTA, 1mM DTT, 50% glicerol Tampão – 200 mM Tris-HCL (pH 8,4), 500 mM KCl Cloreto de magnésio – cofator para ação da enzima Desoxinucleotídios (ATP, TTP, CTP, GTP) – mistura de [ ]’s iguais Primers específicos à sequência que se deseja amplificar Técnicas de Biologia Molecular Fundamentos da Transcriptase Reversa Vírus de RNA tipo VI • Enzima transcriptase reversa CGTCATGCTTAGCCGTTCCGGCTGCAGCTTAAAAAAAAAAAAA mRNA ACGTCGAATTTTTTTTTTTTT cDNA CGTCATGCTTAGCCGTTCCGGCTGCAGCTTAAAAAAAAAAAAA mRNA GCAAGGCCGACGTCGAATTTTTTTTTTTTT cDNA CGTCATGCTTAGCCGTTCCGGCTGCAGCTTAAAAAAAAAAAAA mRNA GCAGTACGAATCGGCAAGGCCGACGTCGAATTTTTTTTTTTTT cDNA • Genoma de RNA orienta a formação de uma molécula de DNA AMV - Vírus da Mieloblastose aviária RNAse I M-MuL-V (Moloney Murine Leukemia Virus) Importante ferramenta para o estudo da expressão gênica a partir do mRNA CGTCATGCTTAGCCGTTCCGGCTGCAGCTTAAAAAAAAAAAAA mRNA GCAGTACGAATCGGCAAGGCCGACGTCGAATTTTTTTTTTTTT cDNA Síntese de uma fita de DNA complementar de fita simples (cDNA) Sequências de cDNA específico podem ser amplificados por PCR PCR 6 Isolamento de RNA por TRIZOL Técnicas de Biologia Molecular Fenol + Isotiocianato de guanidine Isolamento de RNA Imprescindível uso de luvas e amostras no gelo quando possível – evitar RNAse Membranas celulares e nucleares destruídas Manutenção da integridade do RNA Maceração da amostra com TRIZOL Clorofórmio – separação em 3 fases Qualquer amostra tecidual, cultura de células - (100mg) RNA – sobrenadante DNA – camada intermediária Protéinas – camada inferior Método muito utilizado Reagente TRIZOL Precipitação com Isopropanol Lavagem com Etanol 75% Leitura em espectrofotômetro 260nm – quantidade ( A260 x fator de diluição x 0,04 = [ ug/uL]) Razão 260nm/280nm – qualidade do RNA ( ≅ 2,00) Análise por Eletroforese Amostra 100 mg + 1 mL de TRIZOL Homogeneizar • Centrifugar 12.000 g 10 min. 2- 8 °C Principio básico O DNA tem carga negativa Aplicando uma voltagem as moléculas de DNA migrariam para o Vórtex por 15 sec. • Remover sobrenadante transparente para novo tubo • Acrescentar 500 uL de isopropanol Incubar RT °C p/ 2-3 min Centrifugar 12.000 g p/ 15 min catodo • Remover sobrenadante p/ novo tubo Malha de gel de agarose permite o deslocamento do DNA separando- • Acrescentar 200 uL de clorofórmio o por tamanho • Moléculas menores migram mais rápido • Incubar RT °C 10 min • Moléculas maiores migram mais lento • Descartar 3° sobrenadante • Centrifugar 12.000 g p/ 15 min à 2 – 8 °C • Acrescentar 3x 1mL de etanol 75% • Descartar sobrenadante • Centrifugar 3x a 7.500 g por 5 minutos à 2 – 8 °C • Evaporar etanol •Solubilizar pellet ddH2O autoclavada com A distância percorrida é comparada à distância de uma molécula de tamanho conhecido •Leitura espectrofotômetro Análise por Eletroforese Principio básico Agarose – polissacarídeo é aquecido para dissolver em tampão eletrolítico • Tris-Acetato-EDTA – TAE • Tris- Borato-EDTA • Solubilização do gel por esfriamento Aumento da viscosidade da amostra com glicerol Visualizaçãoda corrida da amostra com corante – bromofenol blue Visualização do DNA no gel por brometo de etídeo Cuba de Eletroforese Horizontal 7 8
Download