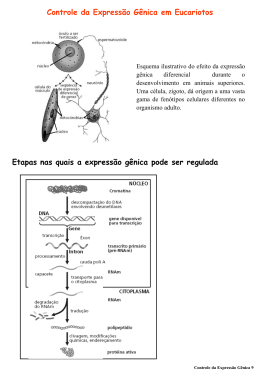
RNAs não codificadores Definição, funções e identificação através da bioinformática Gabriel Zaniboni RNAs Não Codificadores • Molécula de RNA funcional, ou seja, não precisa ser traduzida em proteína para que a informação contida em sua sequência exerça sua função; • Produtos de genes de RNA; RNAs Não Codificadores • RNA ribossômico (rRNA) – principal componente (60 %) dos ribossomos: – 3 rRNAs em procariotos (5 S, 16 S e 23 S), – 4 rRNAs em eucariotos (5 S, 5.8 S, 18 S, 28 S); RNAs Não Codificadores • RNA transportador (tRNA) – molécula adaptadora que atua como elo entre a sequência de nucleotídeos do mRNA e a sequência de aminoácidos das proteínas; RNAs Não Codificadores • Pequeno RNA nucleolar (snoRNA) – pequenas moléculas responsáveis por guiar modificações em outros RNAs durante a maturação destes (rRNAs, tRNAs, snRNAs): – C/D box snoRNAs: associados com metilação, – H/ACA box snoRNAs: associados com pseudouridilação; RNAs Não Codificadores • Pequeno RNA nuclear (snRNA) – presente no núcleo de eucariotos, envolvidos em vários processos, como: – Splice de RNAs, – Regulação de fatores de transcrição e da RNA polimerase II, – Manutenação dos telômeros; RNAs Não Codificadores • Micro RNA (miRNA) – atua na regulação da expressão gênica de eucariotos superiores, um único microRNA pode ter centenas de alvos diferentes: – Mecanismo de complementaridade parcial com mRNAs, geralmente na região 3’ UTR, – Atua diminuindo a expressão de seus alvos; RNAs longos não codificadores • Projetos de sequenciamento genômico evidenciaram que o número de genes codificadores de proteínas não muda significativamente entre os vertebrados, nem mesmo os metazoa; • O que explica a diferença na complexidade biológica entre esses organismos? RNAs longos não codificadores • Sabe-se hoje em dia que a maior parte do genoma dos metazoários é transcrita; • Porção do transcriptoma que não é codificadora chega a ser até 4 vezes maior que a porção codificadora; • Maior parte do genoma, que não codifica proteínas, parece estar envolvido na regulação da expressão gênica durante o desenvolvimento e a diferenciação dos organismos mais complexos; RNAs longos não codificadores • Arbitrariamente considerados como sendo transcritos mais longos do que 200 nucleotídeos; • lncRNAs se sobrepõem com ou intercalam-se entre outros transcritos codificadores ou não-codificadores; RNAs longos não codificadores • Descritos pela primeira vez durante o sequenciamento em larga escala de bibliotecas de cDNA de camundongo (2002); • Dado sua abundância, imaginou-se inicialmente que os lncRNAs eram apenas ruído transcricional; RNAs longos não codificadores • A expressão de muitos lncRNAs é restrita a determinadas fases de desenvolvimento; • Grande parte dos lncRNAs de camundongo são expressos especificamente durante a diferenciação celular de células-tronco embrionárias e no cérebro; RNAs longos não codificadores • A baixa conservação das sequências dos lncRNAs levou à afirmação de que esses transcritos não são funcionais; • A ligação de fatores de transcrição a loci não codificadores e a evidência de seleção purificadora atuando em promotores de lncRNAs sugerem que esse tipo de expressão é regulada; RNAs longos não codificadores • Porém: – Na realidade, vários lncRNAs conhecidos detêm um grau de conservação, sendo a função de vários deles conhecida; – lncRNAs podem exibir trechos de sequências conservadas mais curtas do que aquelas presentes em transcritos codificadores de proteínas (Xist); – A ausência de conservação pode ser indício de altas taxas de evolução das sequências primárias dos lncRNAs, caso essas sequências apresentem maior plasticidade em relação à sua estrutura/função (HAR1); RNAs longos não codificadores • A pouca conservação dos lncRNAs pode indicar que o processo da transcrição, ao invés do produto, é que é funcional (como por exemplo, a abertura de longos trechos de cromatina expõe sítios de ativação transcricional e propicia a atuação da RNA polimerase); • Demais evidências (conservação de estrutura secundária, padrões de splice e localização subcelular) sugerem funções além da remodelação transcricional; Funções • As funções dos lncRNAs não podem ser inferidas sempre com base na sua sequência ou estrutura; • Atuam regulando a expressão gênica em processos de dinâmica cromossomal, biologia de telômeros e organização estrutural subcelular; Modificação da Cromatina • lncRNAs podem mediar alterações epigenéticas recrutando complexos remodeladores de cromatina em loci específicos; • O ncRNA HOTAIR (Hox transcript antisense RNA) se origina no locus HOXC e silencia a transcrição através de 40 kb do locus HOXD (efeito trans); Modificação da Cromatina • O ncRNA HOTAIR seria responsável pelo recrutamento do complexo de remodelamento de cromatina PRC2, que atua através de metilação do DNA; Regulação Transcricional • Transcrição de acentuadores e promotores leva à formação de ncRNAs; • Promotores proximais podem ser transcritos e recrutar proteínas que se ligam a RNA, integrando suas funções na maquinaria transcricional; Regulação Transcricional • Danos no DNA induzem a transcrição do promotor da ciclina D1 (ciclo celular), que recruta a proteína TLS, um inibidor da CBP e p300, silenciando a expressão da ciclina D1; Regulação Transcricional • lncRNAs também podem atuar como cofatores modulação da atividade de fatores de transcrição; na Regulação Transcricional • O lncRNA Evf2 é transcrito a partir de um acentuador, recrutando o fator de transcrição DLX2 para induzir a expressão de genes codificadores de proteínas adjacentes; Regulação Transcricional • lncRNAs podem atuar regulando a atividade da RNA polimerase II; Regulação Transcricional • Um transcrito formado em uma região upstream do locus DHFR forma um triplex na região promotora e impede a ligação do cofator TFIID; Regulação Pós-Transcricional • A capacidade dos lncRNAs de reconhecer sequências complementares permite interações específicas durante o processamento de mRNAs, como splice, edição, transporte, tradução e degradação; • O lncRNA antisenso Zeb2 complementa o sítio de splice 5’ UTR do “dedo de zinco” (zinc finger) Hox mRNA Zeb2, prevenindo o splice de um intron necessário à tradução desse mRNA; Regulação Pós-Transcricional Regulação Pós-Transcricional • A ligação de ncRNAs a mRNAs, ou mesmo a ocorrência de ncRNAs que possuem longos grampos, pode levar à formação de pequenos RNAs de interferência (siRNAs) endógenos e ao silenciamento da expressão gênica; • Essas são as funções conhecidas, mas acredita-se que existam vários outros mecanismos a serem descobertos; Significância Médica • Potencial envolvimento dos lncRNAs em doenças que tenham relacionamento com processos de diferenciação e desenvolvimento, principalmente o câncer; • ncRNA antisenso transcrito a partir do locus p15 (supressor de tumor) tem perfil de expressão alterado em leucemia; Identificação de lncRNAs • A detecção de genes de RNAs não codificadores em sequências genômicas é um problema ainda não resolvido na bioinformática; • Principais abordagens: – Análise composicional, predição de estrutura secundária, homologia estrutural ou de sequências, sinais de promotores e terminadores; Identificação de lncRNAs • Duas ferramentas, QRNA e RNAz, combinam predição de estrutura secundária e análise de homologia entre sequências; • O algoritmo Dynalign realiza um alinhamento de estrutura secundária entre duas sequências de RNA; • Para maior precisão, algumas ferramentas limitam o escopo da análise a famílias de ncRNAs específicas, como tRNA, miRNA e snoRNA; Identificação de lncRNAs • Entretetanto, nenhuma ferramenta de detecção de ncRNAs gerais, disponíveis hoje em dia, é comparável em confiança aos softwares de detecção de genes codificadores de proteínas; • Como os diversos métodos disponíveis para a detecção de ncRNAs são complementares, uma abordagem prática consiste na combinação deles; Identificação de lncRNAs • Moses (modular sequence suite): combina o resultado de diversos métodos diferentes para encontrar regiões no genoma que contenham candidatos a serem ncRNAs; • Inclui: – BLAST, – RNAz, – Dynalign, – RNAfold.
Download