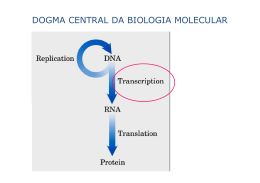
Ácido Ribonucleico RNA AAAAAA cap 5’UTR “open reading frame” coding region 3’UTR Definição de gene: um conceito indefinido Gene: unidade de transcrição, que consiste de um segmento de DNA que se estende do sítio de início de transcrição ao sítio de término de transcrição. O promotor deveria ser incluído? Nem todos os genes codificam proteínas. Muitos genes têm RNA como produto final. Organismos simples como bactérias têm cerca de 500 genes Humanos: cerca de 25.000 genes Sítios de início de transcrição alternativos, splicing alternativo, poliadenilação alterantiva e edição de mRNAs aumentam muito o número de diferentes proteínas originadas desses genes. Ácido Ribonucleico • Para que os genes se expressem, eles devem ser transcritos em moléculas de RNA • O RNA é uma réplica (com poucas diferenças) da fita de DNA complementar à fita que serve de molde • O processo de síntese de RNA é denominado TRANSCRIÇÃO do DNA • A escolha dos genes a serem transcritos e como são transcritos e processados é o que determina todo o processo vital ESTRUTURA DO RNA • Fita única de Ácido Ribonucleico • As unidades monoméricas são constituídas de: • Uma base: Adenina, Citosina, Guanina e URACILA • Um açúcar: RIBOSE • Um grupo fosfato Pirimidinas do RNA Pirimidinas do DNA Nucleosídeos do RNA Estrutura secundária do RNA RNA-POLIMERASES POLIMERIZAÇÃO: 5’ 3’ • Para a atividade de síntese de RNA: – Precisam de uma região promotora no DNA – Requerem uma fita de DNA molde – Precisam de rNTPs – Não requerem “primer” – Não têm atividade de exonuclease ( não fazem “proofreading”) RNA polimerase começa a síntese de um RNA a partir de nucleotídeos livres do meio. Não precisa de “primer” Promotor: Essencial para a montagem do complexo transcricional em procarioto Promotor é o local do DNA onde se monta o complexo que irá iniciar a transcrição Figure 7-35 Molecular Biology of the Cell (© Garland Science 2008) No promotor existem sequências consensuais que são sítios de reconhecimento para fatores de transcrição (elementos cis) Promotor em procarioto: No operador pode se ligar um regulador σ RNA Polimerase de procarioto requer o fator sigma para começar a transcrição Figure 6-11 Molecular Biology of the Cell (© Garland Science 2008) Em bactérias, o RNA pode se estender por 105 pb. A RNA polimerase tem que sintetizá-lo de uma só vez. Múltiplos RNAs sendo transcritos em um gene de bactéria Os ribossomos já se juntam ao RNA para síntese da proteína mesmo antes do término da transcrição O que define qual fita do DNA será molde para síntese do RNA é a localização do promotor O término de transcrição em procarioto requer a formação de uma estrutura secundária na extremidade 3’ do RNA que desestabiliza a RNA polimerase Término de transcrição RNA de procariotos não têm intros e são policistrônicos Cistron: a sequência de nucleotídeos que codifica um único peptídeo Em eucariotos três tipos de RNA-polimerases • RNA - Polimerase I: rRNA (ribossômico) • RNA - Polimerase II: mRNA (mensageiro) • RNA - Polimerase III: tRNA e outros RNAs pequenos (transferência) RNA polimerase II – RNA mensageiro Promotor a b 5’ 3’ c intron d e f g exon 3’ 5’ h Gene c Fita molde Trancrição – RNA pol II 5’ 5’ Pré-mRNA do gene c Processamento AAAAAAA mRNA maduro • Os pré-mRNA de eucariotos podem ter desde algumas centenas até centenas de milhares de nucleotídeos. • Os mRNA maduros variam entre algumas dezenas de nucleotídeos até pouco mais de 10.000 nucleotídeos. • Os RNAs são moléculas pequenas, comparados ao DNA PROMOTOR essencial (core promoter) • Mínimo necessário para a montagem do complexo de pré-iniciação (PIC) e à transcrição basal - 40 +1 + 40 Arcabouço para montagem do PIC (sequência de nts bastante variável) PIC RNA polimerase II + Transcription factors II (TFII) TFIIA TFIIB TFIID TFIIE TFIIH Total: ~ 3,5 MDa Elementos regulatórios comuns no promotor de eucariotos Figure 6-17 DPE: típico de promotores sem TATA box INR e TATA têm efeito sinérgico INR pode Science funcionar Molecular Biology of the Cell (© Garland 2008) sem TATA Algumas sequências são inovações específicas da linhagem Lenhard B et al Nature 13:233, 2012 BRE: factor B recognition factor DPE: downstream promoter element MTE: motive ten element TBP(TFIID) TFIIB DCE: downstream core element TFIID TFIIB TFIID/TAF6-TAF9 TFIID/TAF1 • Promotor proximal: Entre -45 e -250 pares de base na região flaqueadora 5’ do gene (5’FS) • Reguladores positivos (enhancers) e “reguladores negativos” (inhibitors) estendendo-se por milhares de pares de bases • Isoladores: englobando regiões maiores • TSS: transcription start site • TFBS: tanscription factor binding sites • CRM: Cis Regulatory Module Tipo I Promotores com TATA-box têm sítio de início de transcrição bem definido. Correspondem a 10-12% dos genes, e são genes regulados. Tipo II Promotores com ilhas CpG – estendem por 100-200 nts Sem TATA-box Promotores com início de transcrição disperso Genes de expressão constitutiva (79-88% dos genes) Promotores ricos em sequências consensuais para Sp1 Montagem dos fatores gerais de transcrição observada in vitro TATA binding protein: TBP - Uma subunidade do TFIID TBP induz uma dobra no DNA O mediador é um complexo com 26 subunidades (1,2 MDa) Um complexo que permite a interação de vários fatores (scafolding) O mediador também pode ser incluído como um fator geral de transcrição Montagem dos fatores gerais de transcrição • O mediador é recrutado precemente na montagem do PIC • Mediador recruta a acetilase p300. • P-300 acetila todos os alvos e de dissocia do promotor • A montagem do PIC continua Fatores gerais de transcrição: TFIID (com TATA biding protein – TBP e TAFs), TFII A, - B, -D, -E, -F e –H Mediator SAGA TF TFIID Ac A TBP Ac B F Ac Ac E PIC = Pré-Iniciation Complex Complexo remodelador Mediador: MD1-26 Mediador pode funcionar como ativador ou inibidor MD1/MID14: interagem com receptores nucleares (p/ horm. tireoide; glicocorticoides ...) O subcomplexo MD12-MD13-Ciclina C- Cdk8: impede o recrutamento da RNA pol II • TFIIB e TFIIF são necessários para o recrutamento da RNA Pol II • TFIIE junta-se após Pol II • TFIIE recruta TFIIH e modula sua atividade de helicase e cinase NC2 Mediator TFIID TF TBP TF Mot1 B RNA Pol II F E Ac Ac H PIC pronto para dar a largada Início ineficiente: pequenos segmentos de RNA com 3 a 4 nts RNA polimerase II está desfosforilada ao juntar-se ao PIC Início da fosforilação de serinas (Ser5) na cauda C-terminal da Pol II Perda de afinidade pelos FGT RNA com 9 nts aumento da estabilidade DSIF: DRB-Sensitive Inducing Factor NELF: Negative Elongator Factor 5,6-Dichloro-1-beta-D-ribofuranosylbenzimidazole (DRB): inibidor da fase de alongamento da pol II P-TEFb (Positive Trancription Elongation Factor b) cinase P-TEFb: heterodímero Cck9/Ciclina T fosforila Serinas da posição 2 (Ser2) P-TEFb: fosforila NELF e este se desprende da Pol II P-TEFb: fosforila DSIF que troca sua interação com alguns fatores, como enzimas do CAP, pela interação com outros moduladores que alteram a estrutura da cromatina e com enzimas do splicing Atuam ainda na regulação da transcrição de genes: - Locus control regions: LCR Estes elementos regulatórios regulam uma região cromossômica inteira, por ex.: Locus gênico da beta-globina, no qual existem 5 genes, numa extensão de 90 kb. -“Boundary elements” ou isoladores: Funcionam como delimitadores da região de um grupo de genes, de modo a prevenir a interferência de elementos regulatórios vizinhos Processamento do pré-mRNA: 1) adição do CAP CAP é adicionado quando o transcrito atinge ~30 nts. Este é o momento em que a extremidade 5’ emerge da RNA Pol II O complexo enzimático liga-se à cauda C-terminal da Pol II fosforilada em Ser5 Complexo enzimático do CAP •Fosfatase: remove o fosfato g do nucleotídeo trifosfato inicial •Guanilil transferase: transfere GMP numa ligação 5’ – 5’ •Metil transferase: adiciona metil à guanosina CBC: Cap Binding Complex Importante para: • Proteger a extremidade 5’ • Interagir com outras proteínas que processam o RNA • Exportação do núcleo para o citoplasma • Tradução pelos ribossomos CBC Processamento do pré-mRNA: 2) Remoção dos introns (poda ou “splicing”) A porção codificante do pré-RNA de eucariotos é fragmentado por sequências intervenientes, que se interpõem entre os exons: os INTRONS As porções expressas são os EXONS Os introns são bem maiores que os exons. Os intros não têm armação de leitura aberta. São cheios de códigos de parada (stop condons) Cerca de 95% dos genes apresentam splicing alternativo UTR = untranslated region Maquinaria de splicing Spliceosome: • 5 small nuclear RNAs – snRNAs: U1, U2, U4/U5 e U6 • Os snRNAs + proteínas que a eles se associam – snRNPs (small nuclear ribonucleoproteínas) Outros fatores: •Fatores protéicos de splicing: U2AF, SF1, SRFs e outras enzimas (helicases/RNPases, cinase, fosfatases etc) •Proteínas que se ligam ao RNA – RBPs U2AF: U2 Auxiliary Factor SF1: splicing factor 1 SRFs: Splicing regulator factos Y = pirimidina (C/T/U) R = purina (A/G) A reação de corte consiste em duas reações de transesterificação 1) U2AF liga-se ao trato polipirimidina e 3’ SS BBP/SF1 liga-se ao branch point 2) U1-snRNP liga-se ao 5’ SS e U2-snRNP liga-se ao branch point 3) Juntam-se ao complexo U4/U6.U5 formando spliceosome maduro 4) Forma-se o laço (lariat) e ocorre a clivagem da extremidade 5’do intron SS = splicing site Cerca de 200 proteínas são envolvidas nesse processo As demais informações necessárias para o splicing correto estão contidas dentro de sequencias relativamente pequenas (~6 nts) localizadas dentro de exons e introns (elementos cis), às quais se ligam proteínas (elementos trans) • ESEs (exonic splicing enhancers) • ESSs (exonic splicing silencers) • ISEs (intronic splicing enhancers) • ISSs (intronic splicing silencers) Splicing alternativo: processo regulado pelas interações entre RBPs e pré-mRNA RBP = RNA binding proteins Muitas destas proteínas interagem com a cauda C-terminal da Pol II A ação combinada de fatores da maquinaria basal de splicing + fatores auxiliares resulta na modulação do splicing alternativo O comprometimento com o splicing ocorre durante a transcrição O corte pode ocorrer depois, embora seja comum que ocorra durante a transcrição Nova1/2: Neuro-oncological ventral antigen – específico de tecido neural. Binding site: cluster of YCAY Fox-1 family (Fox) proteins: [Fox-1 (A2BP1), Fox-2 (Rbm9) and Fox-3 (NeuN)]. Binding site: UGCAUG PTB: Polypyrimidine binding proteins Efeitos silenciadores Witter JT e Ule J. Trends in genetics, 2011 T-cells intracellular antigen 1 (TIA1) Efeitos ativadores Witter JT e Ule J. Trends in genetics, 2011 RNABPs: RNA binding proteins (podem ser regulados por mecanismos pós-tradução, como fosforilação) Inclusão do exon 2 Exclusão do exon 2 Fatores reguladores com expressão tecido específica Estrutura da cromatina e modificações epigenéticas de histonas Modulam o processo de splicing Nucleossomos Intron Exon Intron Pseudo-exon Intron Depletado de nucleossomos Nucleossomos estão mais concentrados na junções intron/exon Auxiliam na marcação dos exons Marcas da cromatina nos exons: Maior número de: H3K36me3, H3K4me3 e H3K27me2 Depleção de: H3K9me3 Diferentes marcas da cromatina interagem com diferentes adaptadores Exemplos de adaptadores da cromatina que interagem com fatores de splicing Modelo de modificação em histonas interferindo com o processo de splicing PTB: polypyrimidine tract binding protein “Splicing” alternativos do gene da a-tropomiosina. Após o corte, as junções exon-exon são marcadas com um complexo protéico: Exon-Junction-Complex: EJC Stop Exon 1 exon 2 intron Stop SKAR EJC 5' MAGOH PYM Y14 eIF4AIII AAAA 3' Processamento do pré-Mrna: 3) Clivagem e poliadenilação Todos os mRNAs têm cauda Poli-A, com exceção dos mRNAs de histonas Elementos no mRNA (cis) importantes para a maturação da extremidade 3’ 10 a 30 nts Upstream Sequence Element (auxiliar) Sinal de poliadenilação PAS ~30 nts Auxiliar Downstream Sequence Element Downstream Sequence Element Sítio de clivagem Elementos principais PAS: presente em 95% dos mRNAs; 70% (AAUAAA) e 15% (AUUAAA) DSE: YGUGUUYY (Y = pirimidina) ou UUUUU USE: UUUU ou UGUA ou UAUA Na ausência de PAS é necessário USE - UGUA Complexos protéicos essenciais para a maturação da extremidade 3’ CPSF: cleavage and polyadenylation specific factor CstF: cleavage stimulation factor CFI: cleavage factor I CFII: cleavage factor II Participam também da maquinaria básica .... PAP: poly-A polymerase PABP: poly-A binding protein Symplekin: proteína “andaime” RNA Pol II: domínio C terminal, fosforilado em Ser2 RNA Polimerase II Cap mRNA Após a clivagem, a RNA Pol II continua transcrevendo por algumas centenas de nucleotídeos ... A extremidade 5’ sem CAP sofre clivagem pela exonuclease Xm2 Quando a velocidade da Xm2 se iguala a velocidade da Pol II, esse resto mRNA se desprende, o desprendimento da Pol II ocorre em seguida Cerca de 50% dos genes apresentam poliadenilação alternativa APA: alternative polyadenylation Stop pA3 Stop pA2 pA1 m7G pA1 m7G AAAAAAAAAAA siRNA pA2 m7G AAAAAAAAAAA UTR-APA: untranslated alternative polyadenilation Codificam a mesma proteína Mesma proteína, com RNAs com estabilidades diferentes 3’ UTR mais curtos são mais eficientemente transcritos Stop pA3 Stop pA2 pA1 m7G pA1 m7G pA3 m7G AAAAAAAAAAA AAAAAAAAAAA CR-APA: coding region alternative polyadenilation Codificam proteínas distintas 10 a 30 nts Upstream Sequence Element (auxiliar) Sinal de poliadenilação PAS ~30 nts Auxiliar Downstream Sequence Element Downstream Sequence Element Sítio de clivagem Elementos principais PAS canônicos + DSE maior afinidade pela maquinaria basal de PA PAS não canônicos em geral à montante do mais favorecido Aumento do nível de expressão de fatores basais de PA (CPSF160 e Cst64) Favorece a utilização do sítio menos favorecido (não canônico) Donny D. Licatalosi and Robert B. Darnell Nature Genetics 10:75, 2010 Processamentos distintos do mRNA propicia uma grande variedade de mRNAs e proteínas sem aumento do tamanho do genoma. Todos os passos da biogênese do mRNA involve rigoroso controle de qualidade para detectar possíveis erros. Os reprovados, assim como introns, são degradados no núcleo (exsossoma) Edição de mRNA pós-transcrição – RNA guia (gRNA): Remoção ou adição de nucleotídeos para aquisição do “frame” correto de leitura. Edição de RNA do gene coxII de tripanossoma Edição de mRNA do gene da apolipoproteína B humana Introdução de stop codon por deaminação de uma citosina que resulta em uracila (CAA UAA) mRNA + proteínas = mRNP (messenger ribonucleoprotein) m7G AAAAAAAAAAA CAP binding complex PolyA binding protein Exon junction complex Exportação do mRNP do núcleo para o citoplasma Última fase nuclear do mRNA Figure 6-40 Molecular Biology of the Cell (© Garland Science 2008) Síntese de RNAs ribossomais (rRNA) rRNAs: 75% da massa de RNA das células Genes de rRNA: múltiplas cópias no genoma • Humanos: ~200 cópias no genoma haplóide (cromossomos 13, 14, 15. 21 e 22) A biogênese dos ribossomos requer a participação da três RNA-polimerases: Pol I – rRNA 37S que da origem a rRNAs 18S, 5,8S e 28S Pol II – mRNAs das proteínas que compõem o ribossomo Pol III – rRNA 5S A síntese ocorre nos nucléolos, para onde se estendem as alças dos cromossomos com genes de rRNA ativos Nucléolo Genes de rRNA sendo transcritos Figure 6-41 Molecular Biology of the Cell (© Garland Science 2008) Espaçadores não transcritos Organização estrutural dos clusters de genes rRNA Promotor A transcrição é feita pela RNA polimerase I Figure 6-42 Molecular Biology of the Cell (© Garland Science 2008) Figure 6-63 Molecular Biology of the Cell (© Garland Science 2008) RNAs de transferência – tRNAs e outros RNAs pequenos • Várias cópias dos genes distintos que codificam tRNAs em humanos, espalhados no genoma • tRNAs maduros: 74 a 95 nts • Transcritos pela RNA polimerase III: Pol III transcreve genes menores de 400 nts tRNAs, rRNA 5S, U6 snRNA, Alu RNA, RNAs envolvidos no processamento dos tRNAS e outros RNA pequenos RNA Pol III utiliza três tipos de promotores: Promotor do tipo I: para rRNA 5S +1 ICR +120 TTTTTT A IE C box box Promotor ICR: Internal Control Region Promotor tipo I Fatores gerais de transcrição: RNA Pol III, TFIIIA, TFIIIB e TFIIIC Promotor do tipo II: em genes de tRNAs +1 +80 TTTTTT A B box box Promotor A-box e B-box, além de funcionarem como promotores, também codificam as alças D e T dos tRNAs Promotor tipo II Fatores gerais de transcrição: RNA Pol III, TFIIIB e TFIIIC Promotor do tipo III, dos genes: • U6 snRNA componente do spliceosome • 7SK envolvido na regulação do complexo Cdk9/Ciclina T do complexo pTEFb • H1-RNA componente da RNase P, envolvida no processamento dos tRNAs • MRP-RNA envolvido no processamento do pré-rRNA de mitocôndrias 16 nts -215 a -240 +1 +106 TTTTT DSE PSE TATA box Promotor tipo III Fatores gerais de transcrição: RNA Pol III, TFIIIB e SNAPc (snRNA activating protein complex) Maturação dos tRNAs 1) Remoção da extremidade 5’ pela RNase P Rnase P: RNA H1 + 10 subunidades protéicas 2) Remoção da extremidade 3’pela ação de endo e exonucleases 3) Adição de CCA na extremidade 3’ pela ação da tRNA nucleotidil transferase 4) Remoção dos introns pela ação de uma endonuclease Que remove os introns e de uma ligase que liga os exons 5) Numerosas modificações em vários nucleotídeos do tRNA Figure 6-55 Molecular Biology of the Cell (© Garland Science 2008) Figure 6-52 Molecular Biology of the Cell (© Garland Science 2008) Extração de moléculas de RNA no laboratório Uma célula típica de mamíferos contém ~10-5 mg de RNA • 80 a 85% RNA ribossomal (28S, 18S, 5.8S e 5S). • 15-20% consiste de uma variedade de RNAs de baixo peso molecular: RNAs de transferência e pequenos RNAs nucleares com funções diversas. • 1 a 5% consiste de RNAs mensageiros de tamanho heterogêneo. A maioria destes tem cauda poliA. RNA: Uma Molécula INSTÁVEL • O grupo 2’OH da ribose e o fato de ser fita única torna o RNA facilmente degradável • As ribonucleases que degradam o RNA são enzimas muito estáveis e que não precisam de cofatores • Ribonucleases estão amplamente presentes no meio ambiente Material e soluções livres de RNases • LUVAS: absolutamente indispensável; trocar luvas com frequência • Tubos, pipetas volumétricas, ponteiras, pipetadores ... livres de RNases • Limpeza de superfícies para retirada de RNases: H2O2 3%: tratar pentes, bandejas e cubas de eletroforese etc 20 a 30 minutos em H2O2; em seguida lavar muito com água livre de RNases NaOH 0,5 N + SDS 0,5%: limpar as superfícies e depois Água e soluções sem RNases: • ÁGUA: tratar com Diethyl Pyrocarbonate (DEPC) – 0,05% Agitar levemente por algumas horas ou vigorosamente por 30 min. Autoclavar por 30 min e deixar no autoclave após término do ciclo por mais 30 min. DEPC tem que ser removido pois modifica a propriedades do RNA inviabilizando sua utilização posterior. (DEPC CO2 + etanol) • Químicos sólidos: pesar usando espátula RNase-FREE • Soluções: tratar com DEPC 0,05% Soluções que não devem ser autoclavadas: - SDS - NP-40 - NaOH Preparar essas soluções com água já livre de RNAses Para remover RNases, pode-se também filtrar a solução em membrana de nitrocelulose (0,22 mm), por duas vezes. Observação: Não trate cubas, bandejas e pentes com DEPC. Para extração de RNA: • Fragmentos de tecidos podem ser armazenados em “RNA later” até o momento do processamento ou congelados em Nitrogênio líquido. • Lisar as células ou homogeneizar o tecido em solução com agente desnaturante (agente caotrópico). Os mais indicados: guanidinium hydrochloride (G.HCl) ou guanidinium thiocyanate (GTC). • Os demais passos incluem extração com fenol/clorofórmio para remover proteínas, seguida de precipitação com isopropanol ou etanol. Fenol oxida-se facilmente. Cuidado com fenol oxidado!!! Isso compromete seriamente a qualidade do RNA • São também agentes desnaturantes: - SDS ou lauryl sulfate - N-laurylsarcosine (similar a SDS mas solúvel em solução com alta concentração de sal) • Tampão de lise mais leve, para preservar separadamente RNA citoplasmático e de núcleo ou mitocôndrias: - NP-40 tudo no e adicionar inibidores de RNases à ApósFazer separação dasgelo frações, prosseguir a extração com GTC e/oupreparação fenol e clorofórmio Extração com GTC: 1) 106 células – 100 uL solução D - (TECIDO: 1 mg por 100 uL) • Solução D: 4 M GTC; 25 mM citrato de sódio, pH 7,0; 0,5% Sarcosyl; 100 mM 2-mercaptoetanol 2 – Para cada 1 mL de solução D: 0,1 mL 2 M acetato de sódio, pH 5,2 1 mL fenol saturado com água (pH 4,0) 0,2 mL clorofórmio:ácool isoamil (49:1) Agitação vigorosa e gelo por 15 min Centrifugue a 4oC por 5 min para separar as fases 3 – Transfira o sobrenadante contendo RNA para outro tubo e adicione 0,75 volume de isopropanol gelado. Incube a –20oC por 1 h e centrifugue a 10.000 g a 4oC por 20 minutos - Redissolva o pellet em 300 uL de solução D e transfira para tubo de 1,7 mL - Adicione 0,75 volume de isopropanol gelado e incube a –20oC por 1 h - Centrifugue a 10.000 g a 4oC por 10 min. Descarte o sobrenadante. - Lave o pellet com etanol 70% (500 uL) por 3 a 4 vezes - Seque em ar ambiente e dissolva o pellet no menor volume possível de tampão TE ou água previamente tratada com DEPC. Freezer –80oC Caso a quantidade de RNA seja muito pequena, é recomendável adicionar um carreador para auxiliar a precipitação: - Acrilamida linear - tRNA de levedura - Glicogênio Fundamental para o sucesso da purificação de RNA: RAPIDEZ! RNases devem ser inativadas imediamente após a coleta do tecido ou células Verificação da integridade em gel de agarose 28 S 18 S Integridade do RNA íntegro degradado Diferença na migração eletroforética Diferentes concentrações de sal na amostra RNA não completamente dissolvido: Contaminação com DNA genômico: Quantificação em espectrofotômetro: [RNA] ug/mL = A260 x diluição x 40 A260 = absorbância (em densidade óptica) a 260 nm 40 = coeficiente médio de extinção do RNA A solução na qual é medida a absorbância deve pH entre 7,5 e 8.5 (TE ou tampão fosfato) A diluição deve ser tal que A260 não seja menor que 0,1 Contaminação com outras moléculas: Proteínas Medir a absorbância a 280 nm Razão A260/A280 = 2,0 é o ideal para RNA < 2,0 – Refazer extração com fenol/clorofórmio Guanidinium ou beta-mercaptoetanol Medir a absorbância a 230 nm Razão A260/A230: maior que 2 e menor 2,4 > 2,4: guanidinium ou BME em excesso Precipitar novamente em acetato de sódio e etanol e lavar bem com etanol 70% Purificação de mRNA a partir de RNA total Purificação de mRNA com esferas magnéticas
Download